文章信息
- 蒋方圆, 王丽娟, 孙静, 余丽丽, 周璇, 朱益民, 李雪.
- Jiang Fangyuan, Wang Lijuan, Sun Jing, Yu Lili, Zhou Xuan, Zhu Yimin, Li Xue
- 全表型组关联研究方法学研究进展
- Research progress in the methodology used in phenome-wide association studies
- 中华流行病学杂志, 2022, 43(7): 1154-1161
- Chinese Journal of Epidemiology, 2022, 43(7): 1154-1161
- http://dx.doi.org/10.3760/cma.j.cn112338-20211104-00853
-
文章历史
收稿日期: 2021-11-04
2. 浙江大学医学院公共卫生学院流行病与卫生统计学系, 杭州 310058
2. Department of Epidemiology and Biostatistics, School of Public Health, Zhejiang University School of Medicine, Hangzhou 310058, China
对于探索影响疾病发生的遗传易感因素,早期研究多采用基于关键基因或通路的候选策略,并鉴定出一批功能性较强的影响疾病发生和发展的易感基因。但这种方法的准确性和可靠性受群体遗传背景、连锁不平衡和样本量大小等因素的影响较大,且没有考虑基因-基因交互作用。近十年来,全基因组关联研究(genome-wide association study,GWAS)作为一种新兴高通量芯片技术,已发展成为一种用于探索复杂疾病遗传易感机制的成熟检测手段[1]。GWAS发现了单核苷酸多态性(single nucleotide polymorphism,SNP)对复杂疾病易感性的影响,一般是在全基因组范围内通过比较因等位基因突变产生的不同基因型在病例和对照之间的频率差异来鉴定与疾病易感性显著相关的遗传变异。截至2021年6月8日,美国国家人类基因组研究所编制的全基因组关联研究目录(NHGRI GWAS catalog)已收录了5 106篇GWAS,共发现了258 738个遗传变异[2]。
近年来,由于生物医疗大数据如英国生物样本库[3]、中国慢性病前瞻性研究[4]、美国电子病历与基因组学网络[5]和百万退伍军人队列(million veteran program)[6]等大型队列研究电子化病历(electronic medical records,EMR)的广泛应用,使得探索某一暴露因素与成百上千个疾病表型之间的关联,即全表型组关联研究(phenome-wide association study,PheWAS),成为了可能。PheWAS利用EMR作为临床表型的有效数据源,同时将基因型数据与EMR数据相关联,在全疾病谱范围内探索特定遗传变异与所有疾病结局之间的关联。此方法的独特优势是可以同时探索某一暴露与多种疾病表型之间的统计学关系,从而有助于揭示多重因果关联以及疾病间共同的致病机制[7]。此外,流行病学研究也可以作为PheWAS暴露或表型数据的来源。传统的队列研究或临床试验以规范化流程在各研究点通过实验室测量、体格检查、自填问卷等途径收集表型信息,相比EMR不仅具有严格标准化和统一性的优势,并且能选择性地覆盖参与者的健康信息,而EMR往往只能取得依据病例年龄、性别或健康状况而进行的临床化验或检查结果[7]。但是人力物力的较高成本也使得传统队列研究的设计偏向于个别已被认为有价值的研究问题或表型结局,其表型信息的广度与深度存在局限。PheWAS方法对表型的广泛或系统性探索可以较好地弥补这个空缺,发掘意料之外的关联,尤其是基于EMR的PheWAS,其观察次数与变量信息类型更为丰富。在分析阶段,PheWAS基于一系列映射策略,快速从EMR或队列研究中提取多种疾病表型的病例和对照并分析特定基因型频率在其间的分布差异[8]。这种“病例-对照”的分析思路常见于候选基因关联研究、GWAS等遗传关联研究,用以识别与表型相关的遗传变异。
不同于GWAS,PheWAS是一种反向遗传学分析方法。GWAS从特定表型出发,探究其与全基因组范围内遗传变异的关联以揭示目标表型的遗传易感性机制,遵循由“表型”到“基因型”的研究思路;而PheWAS通常基于已有的生物学机制或遗传学背景,关注某种表型,应用该表型相关的特定遗传变异作为工具变量来分析其与大量疾病表型结局的关联,体现了它从“基因型或其他变量”到“表型”的反向遗传学研究特征。有别于GWAS仅关注一种或一类疾病,PheWAS将大量疾病表型结局用表型代码或其他形式表示为系统化的疾病表型组。此类疾病表型编码系统的构建是PheWAS的关键贡献,使得PheWAS相比GWAS能够对遗传多效性、共病负担等关联进行更全面系统的探索[9]。
已有多项研究证明PheWAS方法不仅可以验证GWAS鉴定的基因-表型关联,而且能挖掘GWAS未发现的多效性遗传变异位点[10-12]。2010年,Denny等[13]首次使用PheWAS方法并成功验证了既往已报道的基因型-表型关联(例如rs1333049-冠心病、rs3135388-多发性硬化、rs2200733-房颤、rs6457620-类风湿性关节炎以及rs17234657-克罗恩病,均为既往GWAS已发现的基因型与某一种疾病的关联),此外还发现以上5个遗传变异位点与其他多种疾病表型存在相关性,即表型-遗传-表型关联。随后,PheWAS得到快速发展,该方法被应用于具有更大人群样本和不同数据结构的数据集中,并逐渐发展成为一种应用遗传变异作为工具变量来鉴定表型-表型关联的有效手段。Bush等[12]于2016年开展的一项系统综述以宏观视角对PheWAS的基本原理、方法与发现进行介绍,着重强调了未来PheWAS面临的挑战,对随后的研究起到宏观的指向性作用。针对PheWAS分析已经开发了多种不同的算法和研究策略,因此需要一个更为系统和详细的概述,为选择适当的方法以便更好地实施PheWAS分析提供建议。基于以上研究背景,本研究结合近五年PheWAS的新策略以及本研究团队在相关领域的贡献,一方面聚焦于疾病表型编码策略、关联解读与因果推断等PheWAS关键问题的进展,为既往研究挑战提供了一份最新的答卷;另一方面,本研究针对PheWAS的关键实施步骤、常用分析软件、未来发展方向以及临床研究应用作出详尽总结,为读者提供了更具体的研究图景。
一、PheWAS实施步骤1. 基本原理:表型,即指特定基因型的个体,在一定环境条件下,所表现出来的性状特征的总和[14]。人类能被观察到的结构和功能方面的特性,包括健康表现、疾病特征等都可称之为表型。近些年,研究者提出表型组学这一概念,是指生物体形态特征、功能、行为、分子组成规律等所有生物学性状的集合,是联系生物体基因型和表现型的桥梁。表型组学可通过高通量的表型分析技术和平台与基因组学、转录组学、蛋白质组学、代谢组学结合起来,应用于复杂的生命系统研究[15]。PheWAS分析方法是在表型组学发展过程中建立起来的新研究策略。PheWAS通过对以上多组学数据进行关联和整合,利用遗传因素工具变量在因果关系推断中的优势(例如不受常见混杂因素干扰、因果时序明确等)鉴定表型-基因-表型之间的关联[16],识别疾病易感基因和位点,在早期把风险人群筛选出来,然后对其环境因子、生活方式实施干预,以降低多种疾病的发生风险,为疾病的预防与干预提供了关键线索和指引。结合基因组学等多组学数据,PheWAS有助于全面理解疾病发生发展的机理,发现药物新靶标,从而加速医药产业的发展[17]。研究策略见图 1。其中,数量性状位点(quantitative trait locus,QTL)作为联系基因组和其他组学的重要存在,可用于挖掘各组学研究在基因型到表型发展过程中提供的有效生物信息。基于PheWAS发现的遗传因素或生物标志物与疾病表型的关联,可以通过QTL分析判断遗传变异对基因表达、蛋白质丰度以及代谢产物的影响,帮助明确PheWAS所呈现遗传变异与表型结局关联背后的生物学机制并实现对疾病表型的科学预测。
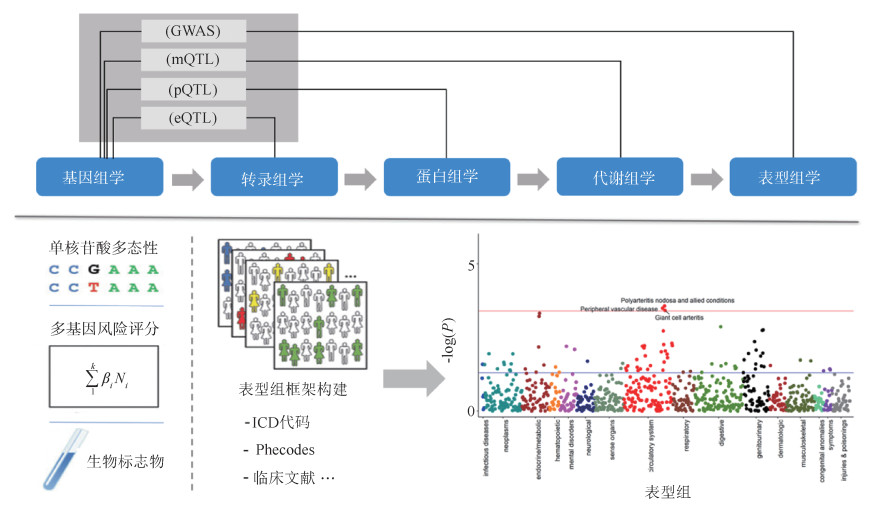
|
| 注:GWAS:全基因组关联研究;mQTL:代谢数量性状基因座;pQTL:蛋白质数量性状基因座;eQTL:表达数量性状基因座;ICD:全球疾病分类;Phecodes:表型代码 图 1 全表型组关联研究策略图—基于组学建立用于预测疾病表型的方法或算法示意图 |
2. PheWAS研究设计的关键步骤和常用的分析软件:
(1)数据质量控制:开展PheWAS的第一个关键步骤是进行质量控制以确定最终纳入分析的疾病表型。与候选研究、临床随访研究不同,PheWAS涉及成百上千个疾病表型,为保障统计学效能,在进行统计分析前需进行严格的质量控制;质量控制需要考虑样本量、唯一值及缺失值等信息。此外,最小病例数作为筛选疾病表型的重要指标,常用于剔除病例数小于该界值的一部分罕见疾病表型。具体研究中考虑到统计效能、结局类型等因素,该界值的选定会存在差异,但通常不低于20例。根据PheWAS Catalog来源的权威性研究,Simonti等[18]在探究28 000名欧洲人种相关遗传变异与疾病表型的关联时,以最小界值20例剔除了罕见疾病表型;而Denny等[10]基于GWAS Catalog健康相关SNP位点的PheWAS,以及Karnes等[19]基于人类白细胞抗原(HLA)遗传变异的PheWAS,则分别以25例和40例作为最小病例数。其后,Verma等[20]还进行模拟分析调整了多项PheWAS常见参数(如最小病例数、病例-对照比)并观察其对统计功效的影响,提出以200例作为常见遗传变异与二分类疾病结局关联分析的最小病例数界值。此外,研究者还可以通过Brion等[21]开发的在线分析工具,根据结局类型(连续型或二分类)和效应量等参数进行统计效能或样本量计算。目前可用于表型质量控制的软件有QUANTO[22]和CLARITE[23]。
(2)暴露因素选择:遗传因素和非遗传因素(如血清生物标志物)均可作为PheWAS分析的暴露因素用于探索其与多种表型之间的关联。其中,遗传因素即遗传变异,可使用既往GWAS报道的单个或多个遗传变异位点以及候选基因关联研究鉴定的功能性遗传变异,作为指代特定表型的工具变量[10]。鉴于表型的多基因遗传特征,另一种方法是通过构建遗传风险评分来代表特定表型的水平进而分析该风险评分与多种疾病表型之间的关联,该研究策略在近几年的PheWAS中已被相继报道[24-26]。值得注意的是,高遗传风险并不完全等同于直接的表型观测或疾病诊断,它体现的是特定表型遗传风险的暴露水平。在PheWAS探究交叉表型的共同致病机制、药物重定位等过程中,遗传因素起到良好的工具变量作用,尤其对于需要大量样本长期随访的研究问题或难以测得的生理指标。此外,在广义的PheWAS中,非遗传因素,包括实验室检查指标(如血清胆固醇)[27]、社会经济因素(如医疗费用支出)[28]乃至特定疾病表型[9]等也可作为PheWAS的暴露因素[11]。例如Zhang等[9]用PheWAS评估不同程度多发性硬化的共病负担,发现并验证了严重的多发性硬化与泌尿生殖系统疾病、代谢性疾病、癫痫和运动障碍等16种其他疾病表型的关联,为多发性硬化重症病例的个性化治疗、监测与管理提供了重要思路。
(3)疾病表型组框架构建:构建疾病表型组框架是PheWAS的主要步骤。对于基于EMR的PheWAS,最直接的疾病表型定义方法是使用国际疾病分类(ICD)编码系统[12]。ICD编码为人群和个体健康状况提供了统一的评价标准。目前ICD系统已进行第十次修订,即ICD-10。该版本相较于ICD-9,不仅在内容上有较大的增补,疾病分类也更加细化(如ICD-9的“神经系统和感觉习惯疾病”在ICD-10中被详细分为“神经系统疾病”“眼和附器疾病”及“耳和乳突疾病”),其代码形式也由单纯数字组成向字母加数字形式转变。由于ICD的编码之间存在相关性,不能直接用于定义独立的疾病表型,因此目前的PheWAS多采用两种不同的疾病表型框架绘制和分析策略。
首先是基于表型代码系统的PheWAS分析策略。表型代码系统最初由美国范德堡大学团队开发,它能够将一个或多个相关的ICD-9代码根据疾病的相似性聚合成相应的疾病组[13]。为了更广泛地应用该表型组学框架,本研究团队与范德堡大学研究团队合作创建了一系列映射策略(图 2),将ICD-10编码成功映射到表型代码系统,并构建了1 866个不同的表型代码[8]。应用这一映射策略可以将ICD-9/10代码转换为表型代码,并可以把具有相似或潜在重叠疾病状态的病例从相应的对照组中排除(例如,在分析2型糖尿病这一疾病表型时,将把1型糖尿病排除在对照组之外),同时允许以高通量的方式对数千种表型进行统计分析(图 2)。第二种策略基于树状表型模型(tree-structured phenotypic model,TreeWAS)[29]。TreeWAS是建立在贝叶斯分析方法下的一种新的表型组学框架,其树状结构是根据ICD-10编码系统的分类层次来确定,树状结构中的每个节点代表一个分类的疾病表型[29]。目前用于定义疾病表型的分析软件工具主要包括PheProb[30]、PheNorm[31]和MAP[32]。

|
| 注:ICD-10-CM:国际疾病分类临床修订第十版;ICD-9-CM:国际疾病分类临床修订第九版;ICD-10:国际疾病分类第十版 图 2 将ICD编码映射到表型代码编码系统并构建表型组学框架 |
(4)统计学分析:连续性变量采用线性回归进行关联分析,分类变量采用logistic回归计算暴露和疾病结局之间的关联,同时进行协变量调整。一般来说,年龄、性别和人群主成分应作为协变量进行校正以控制人口结构偏倚,提高病例组和对照组的可比性[10]。由于PheWAS分析同时对上千种遗传变异和疾病结局进行关联分析,因此需要对分析结果进行多重校正以控制Ⅰ类错误概率,其中最常用的校正方法包括Bonferroni法、Benjamini-Hochberg法、错误发现率(false discovery rate,FDR)和置换测试等[12]。关联分析常用R语言软件包括PheWAS包[13]和TreeWAS包[33]。此外,针对不同数据类型可以选择适当的软件。例如,BioBin[34]适用于罕见遗传变异的PheWAS分析;SPAtest[35]和SAIGE[36]适用于大型队列和生物样本库(病例对照比例失衡)的PheWAS分析等。
(5)PheWAS结果解读:与GWAS相似,PheWAS可被视为一种探索性研究策略。对于PheWAS发现的阳性结果,可能的情况包括真正的因果关联、多效性(Pleiotropy)、临床共病和由于混杂因素导致的虚假关联[12]。为了区分以上几种情况并确定最终的因果联系,可根据PheWAS的结果,在另外一个或几个独立的研究样本中对阳性结果进行验证[12]。此外,孟德尔随机化分析方法(mendelian randomization,MR)也常被用来检验暴露因素与疾病表型之间的因果关联[37]。MR选择与暴露因素具有强相关的遗传变异作为工具变量以代表暴露因素的水平,通过分析遗传变异与暴露因素、遗传变异与结局之间的关联,推断暴露因素与结局之间的因果关系[16]。其中,MR-Egger方法通过对遗传变异-暴露与遗传变异-结局的关联效应拟合回归模型,检验并校正由工具变量多效性所产生的偏倚,从而将真正的因果关联与多效性区别开来[38]。Zhu等[39]还开发了基于GWAS汇总数据的广义孟德尔随机化,该方法可执行双向MR分析以确定因果关联的方向。目前MR分析常用的R语言软件包括Mendelian Randomization[40]和TwoSampleMR[41]。
(6)结果可视化:由于分析的疾病表型数量较多,PheWAS的结果常以图形的方式展示。曼哈顿图(Manhattan plot)是展示关联分析结果最常用的可视化方式,即以染色体位置为横轴,以各关联分析所得P值的变换值-log10(P)为纵轴的散点图[10, 19]。此外,可根据暴露与所有表型之间的相关系数得出表型与表型之间的相关性,并以热图(heatmap)的形式展示出来[39, 42]。常用的画图软件包括PheWAS-View[43]和PhenoGram[44]。PheWAS流程见图 3。
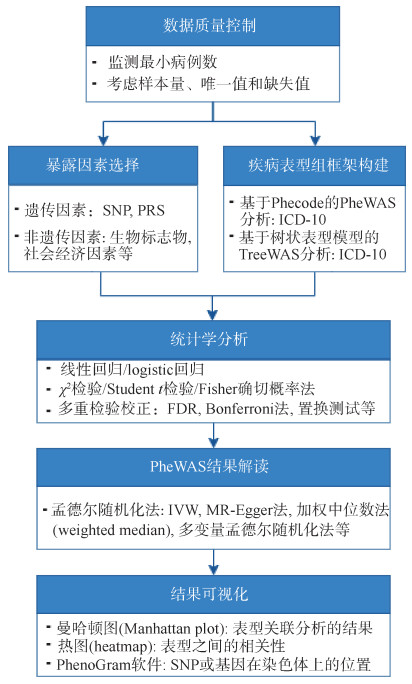
|
| 注:SNP:单核苷酸多态性;PRS:多基因遗传风险评分;Phecode:表型代码;ICD-10:国际疾病分类第十版;TreeWAS:树状表型模型;FDR:错误发现率;IVW:逆方差加权法 图 3 全表型组关联研究(PheWAS)流程 |
截至目前,已发展形成多种PheWAS分析策略。传统的PheWAS方法针对单个或某几个遗传变异位点,即鉴定该遗传变异不同基因型与多种疾病结局之间的关联,可用于验证既往已报道的基因型-表型关联以及鉴定新的关联。例如,Denny等[10]对既往已报道的3 144个GWAS位点进行了系统的PheWAS,共发现有202个基因型-表型关联满足FDR校正,其中有137个(68%)与既往研究结论一致,63个(31%)为新鉴定的基因型-表型关联。
然而,考虑到单个遗传变异位点对疾病表型的效应可能较弱,多个遗传变异位点的联合效应通常更能代表特定表型的实际水平。因此,研究者提出了一种新的基于多基因遗传风险评分(polygenic risk score,PRS)的PheWAS策略,即使用基于多个易感位点计算得到的遗传风险评分来代替特定表型的水平,检测其与多种表型之间的统计学关联。Meng等[24]从既往GWAS中筛选出与25(OH)D相关的6个SNPs,通过构建PRS来代表由遗传因素决定的血清维生素D水平,应用PheWAS分析发现在白种人中由遗传因素决定的血清维生素D水平升高并不会导致任何疾病的发生,但该研究也提出其PheWAS分析可能存在统计效力不足而导致研究结果出现假阴性。Leppert等[26]构建了5种精神性疾病(重度抑郁症、双相情感障碍、精神分裂症、注意力缺陷/多动症和自闭症)的PRS并分别进行PheWAS分析,共发现294个显著性关联,其中大部分与心理健康相关表型有关。
另一个新策略被称为“MR-PheWAS”,目前对于该策略尚没有精准的定义。Li等[45]首先通过PheWAS分析确定与血尿酸水平相关的疾病结局,之后对于PheWAS发现的存在显著性关联的疾病结局,采用MR分析明确血尿酸水平与这些疾病表型之间的因果相关性,并成功验证了血尿酸水平升高与痛风、风湿性关节炎、原发性高血压、心肌梗死等心血管疾病之间的因果关联。因此,该分析策略可被用于在探索暴露因素与疾病结局关联的基础上对其结果的进一步验证和解析。Saunders等[37]基于MR-Base平台提供的GWAS汇总数据,使用MR方法分析了316种中间表型与胶质瘤发病风险之间的因果关系。尽管这种研究策略为在无法获得个体水平数据时开展PheWAS分析提供了可能,但仍具有一定的局限性。首先,这种研究设计是一种候选策略,对于无法获得GWAS汇总数据或者没有开展GWAS的表型,则无法探索其与暴露因素之间的关联。其次,PheWAS使用个体水平数据的一个显著优势是研究单个群体中的交叉表型关联,若使用多种表型的GWAS汇总数据,由于不同GWAS人群之间存在异质性,研究交叉表型关联的统计学效能则会因引入的人群结构偏倚而显著降低。
除了以上两种常见策略外,Cortes等[29]开发的基于ICD-10编码的TreeWAS也可用于全表型组学分析(TreeWAS分析)。以ICD-10编码的树状结构为基础,该方法应用贝叶斯分析法将所有表型的遗传系数建模为一组随机变量,然后应用马尔科夫过程(Markov process)使遗传系数沿树干和树枝进行传递,进而定义更为详细的非独立疾病亚表型。由于TreeWAS将表型划分为更为详细的非独立的疾病亚表型,其关联分析相较于传统的PheWAS分析具有更高的统计学效能,即可以发现更多的暴露-结局关联[29]。Cortes等[29]的研究发现,相较于其他分析方法,TreeWAS检测遗传关联(基因型-疾病表型)的统计学效能提高了20%以上,并发现了HLA等位基因突变与其他免疫性疾病包括风湿性多肌痛、巨细胞动脉炎和甲状腺功能减退之间的关联。此外,Li等[25]应用PheWAS和TreeWAS两种分析方法探究血尿酸水平与多种疾病表型之间的关联,其中PheWAS发现了13个暴露-结局关联,TreeWAS发现了27个关联,二者均发现血尿酸水平升高会显著增加痛风和心脑血管疾病的发病风险。
三、未来展望与临床应用1. PheWAS的优势:PheWAS是一种探索基因型-表型关联的有效方法。不同于GWAS,PheWAS的独特优势之一是它能够同时探索遗传变异与成百上千个表型之间的关联。此外,PheWAS具有发现多效性遗传位点的能力,即遗传变异与两种及以上表型显著相关。遗传多效性的鉴定为揭示不同疾病的共同遗传易感机制提供了重要线索。例如,Zheutlin等[46]的研究发现精神分裂症还与其他多种精神性疾病相关,包括焦虑、精神和人格障碍、自杀行为和记忆力减退,表明这些表型之间可能存在共同的致病机制。PheWAS还可用于探索表型之间的潜在因果关系。PheWAS可利用功能性遗传变异(已知生物学功能和/或临床意义)来指代暴露因素水平,并与分析鉴定的显著表型进行关联分析,通过遗传变异介导的生物学机制对PheWAS结果进行解析,从而揭示潜在的因果关联。例如,Nielsen等[47]探索锌指蛋白529的功能性遗传变异(可显著降低LDL-C水平)与其他疾病表型之间的关联,并得出LDL-C水平与心血管疾病之间潜在的因果关系(LDL-C降低可增加心血管疾病的发病风险)。
2. PheWAS面临的挑战和未来发展方向:PheWAS在方法学方面仍面临几大挑战。首先,大多数PheWAS设计使用既往GWAS报道的遗传变异位点作为工具变量来评估暴露对结局的影响[12],然而,GWAS发现的位点仅占遗传变异的一小部分,且由于GWAS本身的局限性(GWAS发现的位点大多数并不位于功能区,从生物学角度难以阐释遗传变异-暴露-结局的发生发展过程)导致大量信息未被充分利用[48]。近些年随着高通量技术的快速发展,研究者可通过下一代测序技术捕获整个人类基因组的大量遗传数据,包括插入缺失、染色体重排、拷贝数变异和罕见突变等[49]。因此,后续的PheWAS应关注除GWAS位点以外更多的遗传变异作为工具变量来指代暴露因素的水平,这可视作对当前基于GWAS的PheWAS方法学上的补充。
此外,随着各大生物样本库健康数据收集范围的扩大,将会有更多的疾病或疾病中间表型被纳入到PheWAS分析中,从而导致繁重的多重比较负担。PheWAS的另外一项挑战是对发现的暴露-结局关联进行结果解析。GWAS发现的遗传变异位点多是标签SNP,主要位于功能未知的基因间区域,如何从生物学角度阐释遗传变异-暴露-结局的发生发展过程及其临床意义具有很大的挑战[48]。因此,后续研究需要开展一系列生物信息学分析,包括精细定位、功能注释、QTL分析、通路富集分析等,以明确统计学关联背后的生物学机制。其中,精细定位有助于揭示与标签SNP存在高连锁不平衡关联的真正的致病位点[50]。此外,可利用PolyPhen[51]和SIFT[52]等公共数据库对遗传变异进行功能注释,包括所在基因区域、是否位于功能性区域(启动子区、增强子区、脱氧核糖核酸酶高敏感位点区等)以及是否会对所在基因编码的蛋白质功能产生有害影响等,从而预测该遗传变异潜在的生物学功能。还可以基于多组学数据使用QTL分析判断遗传变异是否通过影响基因表达、蛋白质活性和代谢物水平发挥作用[53]。PheWAS鉴定的交叉表型关联也可能是由于相关基因参与不同的生物学过程或通过同种通路影响不同表型所致[12]。在这种情况下,可以利用GO[54]和KEGG[55]等公共数据库进行通路富集分析,以明确基因可能参与的生物学过程。
3. PheWAS在临床上的应用:传统的药物临床试验不仅需要较高的经济成本和较长的时间周期,而且往往成功率较低。因此,应用新的研究方法挖掘药物新的适应证并预测其不良反应(即药物重定位)已成为药物研发的重要手段之一。药物重定位可以识别更有价值的治疗方法,同时也可为潜在的药物不良反应做出预警[56]。PheWAS是实现药物重定位的重要分析工具。研究者可应用PheWAS设计来分析某药物靶基因的遗传多态性与多种疾病结局的关联,进而推测药物作用的新机制和新目标疾病,同时探索药物不良反应[56]。此方法的独特优势是通过由遗传变异所介导的药物靶基因编码蛋白质的功能抑制或增强来模拟该药物在体内的生化效应,并分析该效应与多种疾病结局的关联,从而更系统和全面地探讨药物的有效性和安全性。例如,齐多夫定作为一种反转录酶抑制剂,能靶向抑制端粒反转录酶(TERT)的活性,常用于艾滋病和乙型肝炎的治疗。Rastegar-Mojarad等[56]开展的一项PheWAS发现,TERT编码基因的功能缺失变异与糖尿病发病风险相关,而齐多夫定会抑制TERT的功能,该项研究发现提示了齐多夫定有被应用于糖尿病临床治疗的潜力。近期发表于《自然-通讯》的一项大规模人群研究表明,齐多夫定可以大幅降低糖尿病风险,进一步证实了TERT抑制剂类药物对糖尿病预防的有益作用[57]。脂蛋白相关磷脂酶A2(Lp-LA2)最初被认为通过炎性途径参与动脉粥样硬化的发生,Darapladib作为目前先进的选择性Lp-LA2抑制剂[58],在葛兰素公司开展的两项大型三期临床试验却发现该药物的服用并没有显著降低心血管疾病相关风险[59-61]。Darapladib对东亚人群心血管疾病影响的研究结果也存在矛盾[62-63]。其后Millwood等[64]利用Lp-PLA2编码基因的功能性遗传变异(使得该酶失活)模拟Darapladib对Lp-PLA2的抑制作用,通过PheWAS方法再次验证了Lp-PLA2活性与中国人群心血管疾病风险不存在相关性,为临床药物研发和应用提供了重要的指导和思路。此外,Jerome等[65]使用PheWAS策略对16种靶向药物进行了药物重定位分析,共发现13种新的适应证。除了发现药物适应证之外,PheWAS方法还可用于预测与药物使用有关的潜在副作用[66]。大量临床试验已经确认了血管紧张素转换酶抑制剂、β受体阻滞剂和钙通道阻滞剂(CCB)等一线降压药的疗效,然而临床试验大多局限于高龄或高危群体,随访时间通常在5年以下并且已发现的药物副作用局限于相对常规的几类[67]。在此基础上,Gill等[68]的一项PheWAS对降压药的潜在不良反应进行了全面探索,以降压药靶基因的多态性模拟其抗高血压疗效,发现憩室病风险增加可能是CCB未被发现的药物不良反应,随后的观察性分析进一步验证了服用非二氢吡啶类CCB与憩室病风险增加之间的关联。此外,PheWAS还成功阐明了降脂药[69]和抗抑郁药[70]可能的不良反应。以上研究充分体现了PheWAS分析方法在药物重定位中的应用价值,即通过PheWAS分析药物作用靶基因的多态性与多种疾病表型之间的关联,以预测与药物使用相关的疗效和潜在不良反应,进而为药物早期开发及临床试验提供新型研究证据。
四、总结PheWAS提供了一种基于高通量分析鉴定暴露与多种疾病表型关联的有效方法。近年来,PheWAS方法学和相关软件的发展使其更加适用于大样本、多样化数据的全表型组关联分析。未来的研究应注重解决PheWAS面临的几大挑战,包括如何克服方法学本身的局限性以及如何应用生物学知识阐释PheWAS结果。综上所述,PheWAS能够为暴露因素与结局之间的关联提供有力证据,并有望指导临床药物研发,为临床和公共卫生决策提供理论依据。
利益冲突 所有作者声明无利益冲突
| [1] |
Visscher PM, Wray NR, Zhang Q, et al. 10 Years of GWAS discovery: biology, function, and translation[J]. Am J Hum Genet, 2017, 101(1): 5-22. DOI:10.1016/j.ajhg.2017.06.005 |
| [2] |
MacArthur J, Bowler E, Cerezo M, et al. The new NHGRI-EBI Catalog of published genome-wide association studies (GWAS Catalog)[J]. Nucleic Acids Res, 2017, 45(D1): D896-901. DOI:10.1093/nar/gkw1133 |
| [3] |
Bycroft C, Freeman C, Petkova D, et al. The UK Biobank resource with deep phenotyping and genomic data[J]. Nature, 2018, 562(7726): 203-209. DOI:10.1038/s41586-018-0579-z |
| [4] |
Chen ZM, Chen JS, Collins R, et al. China Kadoorie Biobank of 0.5 million people: survey methods, baseline characteristics and long-term follow-up[J]. Int J Epidemiol, 2011, 40(6): 1652-1666. DOI:10.1093/ije/dyr120 |
| [5] |
Gottesman O, Kuivaniemi H, Tromp G, et al. The electronic medical records and genomics (eMERGE) Network: past, present, and future[J]. Genet Med, 2013, 15(10): 761-771. DOI:10.1038/gim.2013.72 |
| [6] |
Gaziano JM, Concato J, Brophy M, et al. Million Veteran Program: A mega-biobank to study genetic influences on health and disease[J]. J Clin Epidemiol, 2016, 70: 214-223. DOI:10.1016/j.jclinepi.2015.09.016 |
| [7] |
Denny JC, Bastarache L, Roden DM. Phenome-wide association studies as a tool to advance precision medicine[J]. Annu Rev Genomics Hum Genet, 2016, 17: 353-373. DOI:10.1146/annurev-genom-090314-024956 |
| [8] |
Wu P, Gifford A, Meng X, et al. Mapping ICD-10 and ICD-10-CM codes to phecodes: workflow development and initial evaluation[J]. JMIR Med Inform, 2019, 7(4): e14325. DOI:10.2196/14325 |
| [9] |
Zhang TT, Goodman M, Zhu F, et al. Phenome-wide examination of comorbidity burden and multiple sclerosis disease severity[J]. Neurol Neuroimmunol Neuroinflamm, 2020, 7(6): e864. DOI:10.1212/NXI.0000000000000864 |
| [10] |
Denny JC, Bastarache L, Ritchie MD, et al. Systematic comparison of phenome-wide association study of electronic medical record data and genome-wide association study data[J]. Nat Biotechnol, 2013, 31(12): 1102-1111. DOI:10.1038/nbt.2749 |
| [11] |
Bastarache L, Denny JC, Roden DM. Phenome-wide association studies[J]. JAMA, 2022, 327(1): 75-76. DOI:10.1001/jama.2021.20356 |
| [12] |
Bush WS, Oetjens MT, Crawford DC. Unravelling the human genome-phenome relationship using phenome- wide association studies[J]. Nat Rev Genet, 2016, 17(3): 129-145. DOI:10.1038/nrg.2015.36 |
| [13] |
Denny JC, Ritchie MD, Basford MA, et al. PheWAS: demonstrating the feasibility of a phenome-wide scan to discover gene-disease associations[J]. Bioinformatics, 2010, 26(9): 1205-1210. DOI:10.1093/bioinformatics/btq126 |
| [14] |
Ahnert SE. Structural properties of genotype-phenotype maps[J]. J Roy Soc Interface, 2017, 14(132): 20170275. DOI:10.1098/rsif.2017.0275 |
| [15] |
Houle D, Govindaraju DR, Omholt S. Phenomics: the next challenge[J]. Nat Rev Genet, 2010, 11(12): 855-866. DOI:10.1038/nrg2897 |
| [16] |
Emdin CA, Khera AV, Kathiresan S. Mendelian randomization[J]. JAMA, 2017, 318(19): 1925-1926. DOI:10.1001/jama.2017.17219 |
| [17] |
Hebbring S. Genomic and phenomic research in the 21 st century[J]. Trends Genet, 2019, 35(1): 29-41. DOI:10.1016/j.tig.2018.09.007 |
| [18] |
Simonti CN, Vernot B, Bastarache L, et al. The phenotypic legacy of admixture between modern humans and Neandertals[J]. Science, 2016, 351(6274): 737-741. DOI:10.1126/science.aad2149 |
| [19] |
Karnes JH, Bastarache L, Shaffer CM, et al. Phenome-wide scanning identifies multiple diseases and disease severity phenotypes associated with HLA variants[J]. Sci Transl Med, 2017, 9(389): eaai8708. DOI:10.1126/scitranslmed.aai8708 |
| [20] |
Verma A, Bradford Y, Dudek S, et al. A simulation study investigating power estimates in phenome-wide association studies[J]. BMC Bioinformatics, 2018, 19(1): 120. DOI:10.1186/s12859-018-2135-0 |
| [21] |
Brion MJA, Shakhbazov K, Visscher PM. Calculating statistical power in Mendelian randomization studies[J]. Int J Epidemiol, 2013, 42(5): 1497-1501. DOI:10.1093/ije/dyt179 |
| [22] |
Namjou B, Marsolo K, Caroll RJ, et al. Phenome-wide association study (PheWAS) in EMR-linked pediatric cohorts, genetically links PLCL1 to speech language development and IL5-IL13 to eosinophilic esophagitis[J]. Front Genet, 2014, 5: 401. DOI:10.3389/fgene.2014.00401 |
| [23] |
Lucas AM, Palmiero NE, McGuigan J, et al. CLARITE facilitates the quality control and analysis process for EWAS of metabolic-related traits[J]. Front Genet, 2019, 10: 1240. DOI:10.3389/fgene.2019.01240 |
| [24] |
Meng XR, Li X, Timofeeva MN, et al. Phenome-wide Mendelian-randomization study of genetically determined vitamin D on multiple health outcomes using the UK Biobank study[J]. Int J Epidemiol, 2019, 48(5): 1425-1434. DOI:10.1093/ije/dyz182 |
| [25] |
Li X, Meng XR, He YZ, et al. Genetically determined serum urate levels and cardiovascular and other diseases in UK Biobank cohort: A phenome-wide mendelian randomization study[J]. PLoS Med, 2019, 16(10): e1002937. DOI:10.1371/journal.pmed.1002937 |
| [26] |
Leppert B, Millard LAC, Riglin L, et al. A cross-disorder PRS-pheWAS of 5 major psychiatric disorders in UK Biobank[J]. PLoS Genet, 2020, 16(5): e1008185. DOI:10.1371/journal.pgen.1008185 |
| [27] |
Feng QP, Wei WQ, Chung CP, et al. Relationship between very low low-density lipoprotein cholesterol concentrations not due to statin therapy and risk of type 2 diabetes: A US-based cross-sectional observational study using electronic health records[J]. PLoS Med, 2018, 15(8): e1002642. DOI:10.1371/journal.pmed.1002642 |
| [28] |
Cai W, Cagan A, He ZL, et al. A phenome-wide analysis of healthcare costs associated with inflammatory bowel diseases[J]. Dig Dis Sci, 2021, 66(3): 760-767. DOI:10.1007/s10620-020-06329-9 |
| [29] |
Cortes A, Dendrou CA, Motyer A, et al. Bayesian analysis of genetic association across tree-structured routine healthcare data in the UK Biobank[J]. Nat Genet, 2017, 49(9): 1311-1318. DOI:10.1038/ng.3926 |
| [30] |
Sinnott JA, Cai F, Yu S, et al. PheProb: probabilistic phenotyping using diagnosis codes to improve power for genetic association studies[J]. J Am Med Inform Assoc, 2018, 25(10): 1359-1365. DOI:10.1093/jamia/ocy056 |
| [31] |
Yu S, Ma YM, Gronsbell J, et al. Enabling phenotypic big data with PheNorm[J]. J Am Med Inform Assoc, 2018, 25(1): 54-60. DOI:10.1093/jamia/ocx111 |
| [32] |
Liao KP, Sun JH, Cai TA, et al. High-throughput multimodal automated phenotyping (MAP) with application to PheWAS[J]. J Am Med Inform Assoc, 2019, 26(11): 1255-1262. DOI:10.1093/jamia/ocz066 |
| [33] |
Cox NJ. Reaching for the next branch on the biobank tree of knowledge[J]. Nat Genet, 2017, 49(9): 1295-1296. DOI:10.1038/ng.3946 |
| [34] |
Moore CB, Wallace JR, Frase AT, et al. BioBin: a bioinformatics tool for automating the binning of rare variants using publicly available biological knowledge[J]. BMC Med Genomics, 2013, 6 Suppl 2(Suppl 2): S6. DOI:10.1186/1755-8794-6-S2-S6 |
| [35] |
Dey R, Schmidt EM, Abecasis GR, et al. A fast and accurate algorithm to test for binary phenotypes and its application to PheWAS[J]. Am J Hum Genet, 2017, 101(1): 37-49. DOI:10.1016/j.ajhg.2017.05.014 |
| [36] |
Zhou W, Nielsen JB, Fritsche LG, et al. Efficiently controlling for case-control imbalance and sample relatedness in large-scale genetic association studies[J]. Nat Genet, 2018, 50(9): 1335-1341. DOI:10.1038/s41588-018-0184-y |
| [37] |
Saunders CN, Cornish AJ, Kinnersley B, et al. Searching for causal relationships of glioma: a phenome-wide Mendelian randomisation study[J]. Br J Cancer, 2021, 124(2): 447-454. DOI:10.1038/s41416-020-01083-1 |
| [38] |
Burgess S, Thompson SG. Interpreting findings from Mendelian randomization using the MR-Egger method[J]. Eur J Epidemiol, 2017, 32(5): 377-389. DOI:10.1007/s10654-017-0255-x |
| [39] |
Zhu ZH, Zheng ZL, Zhang FT, et al. Causal associations between risk factors and common diseases inferred from GWAS summary data[J]. Nat Commun, 2018, 9(1): 224. DOI:10.1038/s41467-017-02317-2 |
| [40] |
Yavorska OO, Burgess S. MendelianRandomization: an R package for performing Mendelian randomization analyses using summarized data[J]. Int J Epidemiol, 2017, 46(6): 1734-1739. DOI:10.1093/ije/dyx034 |
| [41] |
Hemani G, Zheng J, Elsworth B, et al. The MR-Base platform supports systematic causal inference across the human phenome[J]. eLife, 2018, 7. DOI:10.7554/eLife.34408 |
| [42] |
Verma A, Lucas A, Verma SS, et al. PheWAS and beyond: the landscape of associations with medical diagnoses and clinical measures across 38, 662 individuals from geisinger[J]. Am J Hum Genet, 2018, 102(4): 592-608. DOI:10.1016/j.ajhg.2018.02.017 |
| [43] |
Pendergrass SA, Dudek SM, Crawford DC, et al. Visually integrating and exploring high throughput Phenome- Wide Association Study (PheWAS) results using PheWAS- View[J]. BioData Min, 2012, 5(1): 5. DOI:10.1186/1756-0381-5-5 |
| [44] |
Wolfe D, Dudek S, Ritchie MD, et al. Visualizing genomic information across chromosomes with PhenoGram[J]. BioData Min, 2013, 6(1): 18. DOI:10.1186/1756-0381-6-18 |
| [45] |
Li X, Meng XR, Spiliopoulou A, et al. MR-PheWAS: exploring the causal effect of SUA level on multiple disease outcomes by using genetic instruments in UK Biobank[J]. Ann Rheum Dis, 2018, 77(7): 1039-1047. DOI:10.1136/annrheumdis-2017-212534 |
| [46] |
Zheutlin AB, Dennis J, Karlsson Linnér R, et al. Penetrance and pleiotropy of polygenic risk scores for schizophrenia in 106, 160 patients across four health care systems[J]. Am J Psychiatry, 2019, 176(10): 846-855. DOI:10.1176/appi.ajp.2019.18091085 |
| [47] |
Nielsen JB, Rom O, Surakka I, et al. Loss-of-function genomic variants highlight potential therapeutic targets for cardiovascular disease[J]. Nat Commun, 2020, 11(1): 6417. DOI:10.1038/s41467-020-20086-3 |
| [48] |
Tam V, Patel N, Turcotte M, et al. Benefits and limitations of genome-wide association studies[J]. Nat Rev Genet, 2019, 20(8): 467-484. DOI:10.1038/s41576-019-0127-1 |
| [49] |
Hardwick SA, Deveson IW, Mercer TR. Reference standards for next-generation sequencing[J]. Nat Rev Genet, 2017, 18(8): 473-484. DOI:10.1038/nrg.2017.44 |
| [50] |
Schaid DJ, Chen W, Larson NB. From genome-wide associations to candidate causal variants by statistical fine-mapping[J]. Nat Rev Genet, 2018, 19(8): 491-504. DOI:10.1038/s41576-018-0016-z |
| [51] |
Adzhubei IA, Schmidt S, Peshkin L, et al. A method and server for predicting damaging missense mutations[J]. Nat Methods, 2010, 7(4): 248-249. DOI:10.1038/nmeth0410-248 |
| [52] |
Kumar P, Henikoff S, Ng PC. Predicting the effects of coding non-synonymous variants on protein function using the SIFT algorithm[J]. Nat Protoc, 2009, 4(7): 1073-1081. DOI:10.1038/nprot.2009.86 |
| [53] |
The ENCODE Project Consortium. The ENCODE (ENCyclopedia Of DNA Elements) Project[J]. Science, 2004, 306(5696): 636-640. DOI:10.1126/science.1105136 |
| [54] |
The Gene Ontology Consortium. Expansion of the gene ontology knowledgebase and resources[J]. Nucleic Acids Res, 2017, 45(D1): D331-338. DOI:10.1093/nar/gkw1108 |
| [55] |
Kanehisa M, Goto S, Kawashima S, et al. The KEGG resource for deciphering the genome[J]. Nucleic Acids Res, 2004, 32 Suppl 1: D277-280. DOI:10.1093/nar/gkh063 |
| [56] |
Rastegar-Mojarad M, Ye Z, Kolesar JM, et al. Opportunities for drug repositioning from phenome-wide association studies[J]. Nat Biotechnol, 2015, 33(4): 342-345. DOI:10.1038/nbt.3183 |
| [57] |
Ambati J, Magagnoli J, Leung H, et al. Repurposing anti-inflammasome NRTIs for improving insulin sensitivity and reducing type 2 diabetes development[J]. Nat Commun, 2020, 11(1): 4737. DOI:10.1038/s41467-020-18528-z |
| [58] |
Huang FB, Wang K, Shen JH. Lipoprotein-associated phospholipase A2:The story continues[J]. Med Res Rev, 2020, 40(1): 79-134. DOI:10.1002/med.21597 |
| [59] |
O'Donoghue ML, Braunwald E, White HD, et al. Effect of darapladib on major coronary events after an acute coronary syndrome: the SOLID-TIMI 52 randomized clinical trial[J]. JAMA, 2014, 312(10): 1006-1015. DOI:10.1001/jama.2014.11061 |
| [60] |
The STABILITY Investigators. Darapladib for preventing ischemic events in stable coronary heart disease[J]. N Engl J Med, 2014, 370(18): 1702-1711. DOI:10.1056/NEJMoa1315878 |
| [61] |
Hassan M. STABILITY and SOLID-TIMI 52:Lipoprotein associated phospholipase A2 (Lp-PLA2) as a biomarker or risk factor for cardiovascular diseases[J]. Glob Cardiol Sci Pract, 2015, 2015(1): 6. DOI:10.5339/gcsp.2015.6 |
| [62] |
Jang Y, Waterworth D, Lee JE, et al. Carriage of the V279F null allele within the gene encoding Lp-PLA2 is protective from coronary artery disease in South Korean males[J]. PLoS One, 2011, 6(4): e18208. DOI:10.1371/journal.pone.0018208 |
| [63] |
Wang QQ, Hao YC, Mo XB, et al. PLA2G7 gene polymorphisms and coronary heart disease risk: a meta-analysis[J]. Thromb Res, 2010, 126(6): 498-503. DOI:10.1016/j.thromres.2010.09.009 |
| [64] |
Millwood IY, Bennett DA, Walters RG, et al. A phenome-wide association study of a lipoprotein- associated phospholipase A2 loss-of-function variant in 90 000 Chinese adults[J]. Int J Epidemiol, 2016, 45(5): 1588-1599. DOI:10.1093/ije/dyw087 |
| [65] |
Jerome RN, Joly MM, Kennedy N, et al. Leveraging human genetics to identify safety signals prior to drug marketing approval and clinical use[J]. Drug Saf, 2020, 43(6): 567-582. DOI:10.1007/s40264-020-00915-6 |
| [66] |
Diogo D, Tian C, Franklin CS, et al. Phenome-wide association studies across large population cohorts support drug target validation[J]. Nat Commun, 2018, 9(1): 4285. DOI:10.1038/s41467-018-06540-3 |
| [67] |
Frieden TR. Evidence for health decision making - beyond randomized, controlled trials[J]. N Engl J Med, 2017, 377(5): 465-475. DOI:10.1056/NEJMra1614394 |
| [68] |
Gill D, Georgakis MK, Koskeridis F, et al. Use of genetic variants related to antihypertensive drugs to inform on efficacy and side effects[J]. Circulation, 2019, 140(4): 270-279. DOI:10.1161/circulationaha.118.038814 |
| [69] |
Rao AS, Lindholm D, Rivas MA, et al. Large-scale phenome-wide association study of PCSK9 variants demonstrates protection against ischemic stroke[J]. Circ Genom Precis Med, 2018, 11(7): e002162. DOI:10.1161/CIRCGEN.118.002162 |
| [70] |
Verma SS, Josyula N, Verma A, et al. Rare variants in drug target genes contributing to complex diseases, phenome-wide[J]. Sci Rep, 2018, 8(1): 4624. DOI:10.1038/s41598-018-22834-4 |
 2022, Vol. 43
2022, Vol. 43


