文章信息
- 陈璐, 司佳卉, 孙点剑一, 余灿清, 郭彧, 裴培, 陈君石, 陈铮鸣, 吕筠, 李立明, 代表中国慢性病前瞻性研究项目协作组.
- Chen Lu, Si Jiahui, Sun Dianjianyi, Yu Canqing, Guo Yu, Pei Pei, Chen Junshi, Chen Zhengming, Lyu Jun, Li Liming, for the China Kadoorie Biobank Collaborative Group
- 中国成年人生活方式和心血管代谢因素与甲基化年龄加速的相关性分析
- Association of lifestyle and cardiometabolic risk factors with epigenetic age acceleration in adults in China
- 中华流行病学杂志, 2022, 43(7): 1019-1029
- Chinese Journal of Epidemiology, 2022, 43(7): 1019-1029
- http://dx.doi.org/10.3760/cma.j.cn112338-20211020-00806
-
文章历史
收稿日期: 2021-10-20
2. 北京大学公众健康与重大疫情防控战略研究中心, 北京 100191;
3. 中国医学科学院阜外医院, 国家心血管疾病中心, 北京 100037;
4. 中国医学科学院, 北京 100730;
5. 国家食品安全风险评估中心, 北京 100022;
6. 英国牛津大学临床与流行病学研究中心纳菲尔德人群健康系, 英国 OX3 7LF;
7. 北京大学分子心血管学教育部重点实验室, 北京 100191
2. Peking University Center for Public Health and Epidemic Preparedness & Response, Beijing 100191, China;
3. Fuwai Hospital, Chinese Academy of Medical Sciences, National Center for Cardiovascular Diseases, Beijing 100037, China;
4. Chinese Academy of Medical Sciences, Beijing 100730, China;
5. China National Center for Food Safety Risk Assessment, Beijing 100022, China;
6. Clinical Trial Service Unit and Epidemiological Studies Unit, Nuffield Department of Population Health, University of Oxford, Oxford OX3 7LF, UK;
7. Key Laboratory of Molecular Cardiovascular Sciences (Peking University), Ministry of Education, Beijing 100191, China
生物学年龄(biological age)可衡量个体的功能状态[1],解释相同实足年龄(chronological age,CA)者间衰老速率的变异[2]。表观遗传时钟(epigenetic clock),又称为DNA甲基化年龄(DNA methylation age,DNAm age),是近年来备受关注、最具潜力的反映生物学年龄的指标之一,不仅可以很好地拟合实足年龄,还在预测死亡、心血管疾病等健康结局方面具有良好表现[1, 3]。近年来研究中常见的甲基化年龄包括传统的Horvath时钟[4]和Hannum时钟[5],以及衍生的第二代甲基化年龄,如DNAm PhenoAge[6]和DNAm GrimAge[3]等。Horvath时钟的构建是基于血液和更多其他组织,其他更多的甲基化年龄是基于血液构建。传统甲基化年龄在构建模型时仅纳入年龄相关胞嘧啶鸟嘌呤(cytosine-phosphate-guanine,CpG)双核苷酸位点,第二代模型则增加利用死亡结局及与死亡风险相关的临床指标、生物标志、吸烟量等来筛选CpG位点。各种甲基化年龄纳入的CpG位点数目从几十到上千不等。多数甲基化年龄是基于西方人群构建的,目前仅见一个在中国人群中基于传统方法构建的甲基化年龄——Li时钟[7]。
环境因素会影响个体的表观遗传调控。有研究表明生活方式和心血管代谢因素与以甲基化年龄衡量的衰老速率存在相关性,但是已有研究结果并不一致[1, 3, 6, 8-10],可能与研究人群的种族和使用的甲基化年龄指标不同等有关。目前尚无研究在同一人群中比较分析多种甲基化年龄指标与生活方式及心血管代谢因素的关联。本研究旨在利用中国慢性病前瞻性研究(China Kadoorie Biobank,CKB)中有基线外周血全基因组甲基化检测数据的研究对象,探索5种甲基化年龄与生活方式和心血管代谢因素间的相关性。
对象与方法1. 研究对象:CKB项目基线调查于2004-2008年在5个城市及5个农村地区开展,共纳入51万余名30~79岁的研究对象,具体设计见文献[11-13]。项目随后开展专题研究,选取部分研究对象的基线血标本进行DNA甲基化检测。抽样设计:首先,从全部队列人群中排除基线时自报患有缺血性心脏病、脑卒中、恶性肿瘤及自报服用降脂药者。然后,选择完成基线调查之日起至2015年12月31日间新发急性心肌梗死(ICD-10:I21)或因缺血性心脏病(I20~I25)死亡者作为病例组,随访中新发恶性肿瘤(C00~C97)或脑血管疾病(I60~I69)者除外。从随访期间未发生缺血性心脏病、恶性肿瘤或脑血管疾病的队列成员中,为每个病例按照基线年龄(相差≤3岁)、出生年(相差≤3年)、性别(男、女)、地区(10个项目地区)以及基线采血时空腹时间(0~6、6~8、8~10或≥10 h)匹配一名对照。截至2015年12月31日,按照上述流程,剔除无法匹配对照的病例后,共得到4 603对病例和对照。进一步剔除既往没有生化指标、代谢组学或全基因组DNA分型检测结果者或DNA样本浓度 < 50 ng/μl者,最终确定494对病例和对照,进行全基因组DNA甲基化检测。
2. 生活方式相关因素及心血管代谢因素的评价和定义:研究对象的基线人口学特征(年龄、性别、地区和文化程度)、生活方式因素(吸烟、饮酒、膳食摄入和体力活动)、疾病史以及用药情况通过面对面询问获取。吸烟量、饮酒量、体力活动代谢当量以及膳食摄入的转换和计算参见已发表文献[14-17]。身高、体重和腰围等体格指标及血压由经过统一培训的调查员采用统一工具测量[11, 18]。本研究参考我国卫生行业标准对体格状态进行判定[19]:①根据BMI(kg/m2)进行体重分类:低体重(< 18.5)、正常体重(18.5~23.9)、超重(24.0~27.9)、肥胖(≥28.0);②根据腰围(cm)判定中心性肥胖:非中心性肥胖(男性 < 85.0/女性 < 80.0)、中心性肥胖前期(男性85.0~89.9/女性80.0~84.9)、中心性肥胖(男性≥90.0/女性≥85.0)。
基线调查时对每名研究对象采集血样,不要求研究对象在采血前禁食或空腹。取微量血样采用SureStep Plus强生稳步倍加型血糖仪现场测量血糖。若研究对象任意时点血糖水平异常(7.8~11.0 mmol/L),则要求该对象次日早上复测FPG。在英国牛津大学实验室(Wolfson Laboratory,Clinical Trial Service Unit & Epidemiological Studies Unit)对基线血浆进行生化指标检测。TC的测定使用Beckman-Coulter AU680生化分析仪(Beckman Coulter Incorporation,United Kingdom)。
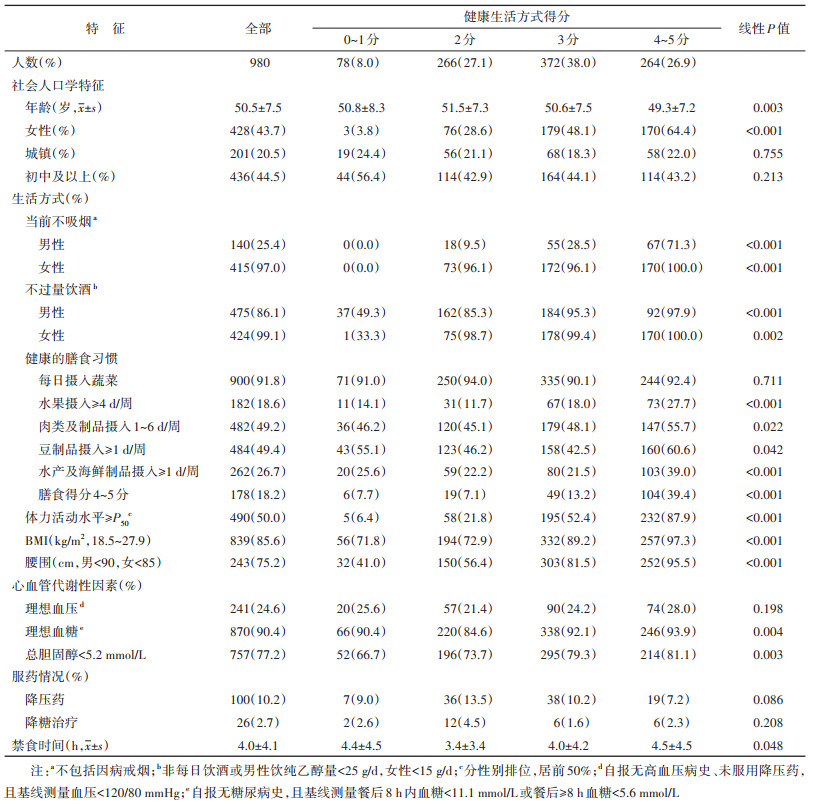
参考CKB项目既往相关研究[20-22],本研究纳入的生活方式相关因素包括:吸烟、饮酒、饮食习惯(新鲜蔬菜、新鲜水果、肉类及制品、豆制品、水产及制品)、体力活动和用来反映能量平衡的体格状态。对健康的饮食习惯定义:每日摄入蔬菜,水果摄入≥4 d/周,肉类及制品摄入1~6 d/周,豆制品摄入≥1 d/周,水产及制品摄入≥1 d/周。每符合一条计1分,合计值为膳食得分。健康生活方式定义为:当前不吸烟(排除因病戒烟);非每日饮酒或男性每日摄入纯乙醇量 < 25 g、女性 < 15 g;膳食得分4~5分;总体力活动水平分性别排位居前50%;BMI介于18.5~27.9 kg/m2,且男性腰围 < 90.0 cm/女性腰围 < 85.0 cm。每符合一条定义计1分,合计值为健康生活方式得分,取值范围为0~5分。
本研究纳入的心血管代谢因素包括血压、血糖及TC水平。健康的心血管代谢因素:自报无高血压病史、未服用降压药,且基线测量SBP < 120 mmHg(1 mmHg=0.133 kPa)及DBP < 80 mmHg;自报无糖尿病史,且基线测量餐后8 h内血糖 < 11.1 mmol/L或餐后8 h或以上血糖 < 5.6 mmol/L;TC < 5.2 mmol/L[23-24]。每符合一条定义计1分,合计值为健康心血管代谢因素得分,取值范围为0~3分。
理想心血管健康得分综合考虑了生活方式和心血管代谢因素,等于健康生活方式得分与健康心血管代谢因素得分的合计值,取值范围为0~8分。
3. 全基因组甲基化检测及DNA甲基化年龄构建:采用Illumina公司的Infinium MethylationEPIC BeadChip(850K)对基线外周血DNA提取液进行全基因组甲基化检测。使用minfi包处理甲基化数据,质控剔除以下情况的位点:①检测的为单核苷酸多态性(SNPs)而非CpGs位点(n=59);② 5%的样本检测到信号的磁珠数目 < 3(n=1 644),> 1%的样本检出P > 0.05(n=2 536);③ CpGs位点、Ⅰ型探针的单碱基延伸位点或探针体与SNPs重合,且SNPs位点在东亚人群中最小等位基因频率 > 0.05(1 000 Genome Project:20 130 502)[25];④可能与其他基因组位点杂交[25](3和4共包含132 762个位点),最终纳入747 726个CpGs位点[26]。本研究剔除性别错误(n=2)、各探针缺失率 > 0.01(n=2)、单独批次检测(n=2)和样本聚类共甲基化模式为异常值(n=2)的样本,最终共纳入980人[26]。
本研究应用既往研究提出的算法分别计算Horvath时钟、Hannum时钟、DNAm PhenoAge、GrimAge和Li时钟5种甲基化年龄[3-7]。前4种甲基化年龄主要是基于西方人群研究构建的甲基化年龄预测模型,分别纳入了353、71、513和1 030个CpG位点;其中包含了来自Illumina公司Infinium HumanMethylation27、HumanMethylation450和MethylationEPIC(850K)三款不同芯片的位点。本研究采用850K芯片,质控后数据能匹配到的位点数分别为311、59、474和853个位点。将这些CpG位点的甲基化水平输入Horvath[4]开发的甲基化年龄的计算平台(https://dnamage.genetics.ucla.edu/),基于K最邻近算法对缺失CpG位点进行插补,进一步基于平台中内嵌的各位点对应系数,该平台可自动计算出这4种甲基化年龄,并可以推算出不同类型白细胞的构成[27]。Li时钟纳入239个CpG位点,所用芯片为Infinium HumanMethylation450;本研究中经过质控后可匹配205个位点。
表观遗传年龄加速(epigenetic age acceleration,AA)指机体的甲基化年龄相比实足年龄的偏离,具体计算为甲基化年龄对实足年龄回归的残差值,正负值分别代表表观遗传年龄加速或减速[28]。本研究用于DNA甲基化检测的为基线血样,甲基化年龄反映了研究对象基线无重大疾病状态时的生理状态。
4. 统计学分析:按照健康生活方式得分分组,报告各组研究对象的基线特征,并进行线性趋势检验。计算各甲基化年龄之间及与实足年龄之间的Pearson相关系数;并进一步计算各甲基化年龄加速之间的Pearson相关系数。利用一般线性模型分析生活方式和心血管代谢因素与各甲基化年龄加速的相关性。分别以各生活方式因素、各心血管代谢因素或健康生活方式得分、健康心血管代谢因素得分、理想心血管健康得分为自变量,以标准化后的各甲基化年龄加速指标(Horvath_AA、Hannum_AA、DNAm PhenoAge_AA、GrimAge_AA、Li_AA)为因变量。当分析单个生活方式因素时,模型同时调整其他4种生活方式因素以及年龄、性别、10个项目地区、餐后≥8 h(是、否)、设计分组(病例组或对照组)、文化程度、服用降压药(是、否)、接受降糖治疗(是、否)、细胞组分(CD8+T细胞、CD4+T细胞、浆细胞、自然杀伤细胞、单核细胞和粒细胞)以及检测批次。当分析单个心血管代谢因素时,模型同时调整其他3个心血管代谢因素以及上述各生活方式因素和协变量。将吸烟量、纯乙醇摄入量、体力活动水平、BMI或腰围按连续型变量重新纳入模型进行线性趋势检验,其中吸烟量或纯乙醇摄入量的线性趋势检验仅在当前吸烟者或饮酒者中进行。当各类健康因素得分为自变量时,模型调整上述协变量,并在健康心血管代谢因素得分的分析中,额外调整健康生活方式得分组。将各类健康因素得分重新按连续型变量纳入模型进行线性趋势检验。所有分析均采用Stata 15.0软件进行。以双侧P < 0.05为相关性有统计学意义。
结果1. 研究对象基本特征:在980名研究对象中,男性占56.3%,农村居民占79.5%,基线调查时年龄为(50.5±7.5)岁。仅26.9%的研究对象具有4或5项健康生活方式(表 1)。随健康生活方式得分增加,女性和具有理想TC水平者的比例越高。
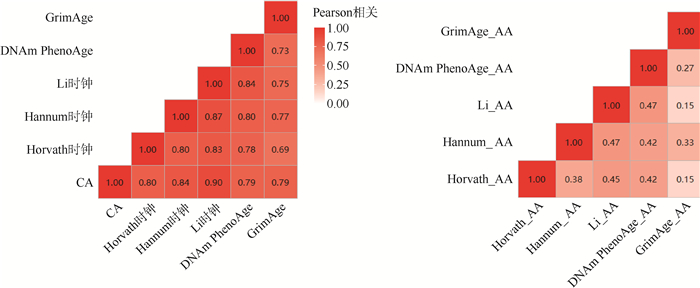
2. 各甲基化年龄相关性及预测误差:各甲基化年龄均与实足年龄呈中高度相关,相关系数(r)介于0.79~0.90之间。各甲基化年龄之间也呈中高度相关,r介于0.69~0.87之间。但是,当调整实足年龄后,甲基化年龄加速之间的相关性减弱,r介于0.15~0.47之间。其中,GrimAge_AA与其他甲基化年龄加速指标之间的相关性最低。见图 1。Horvath时钟、Hannum时钟、Li时钟、DNAm PhenoAge和GrimAge对实足年龄的预测误差(root mean square error)分别为4.51、4.06、3.30、4.57和4.59年(图中未展示)。
|
| 图 1 980名研究对象各甲基化年龄与实足年龄(CA)之间(左)及各甲基化年龄加速(AA)之间(右)的相关性 |
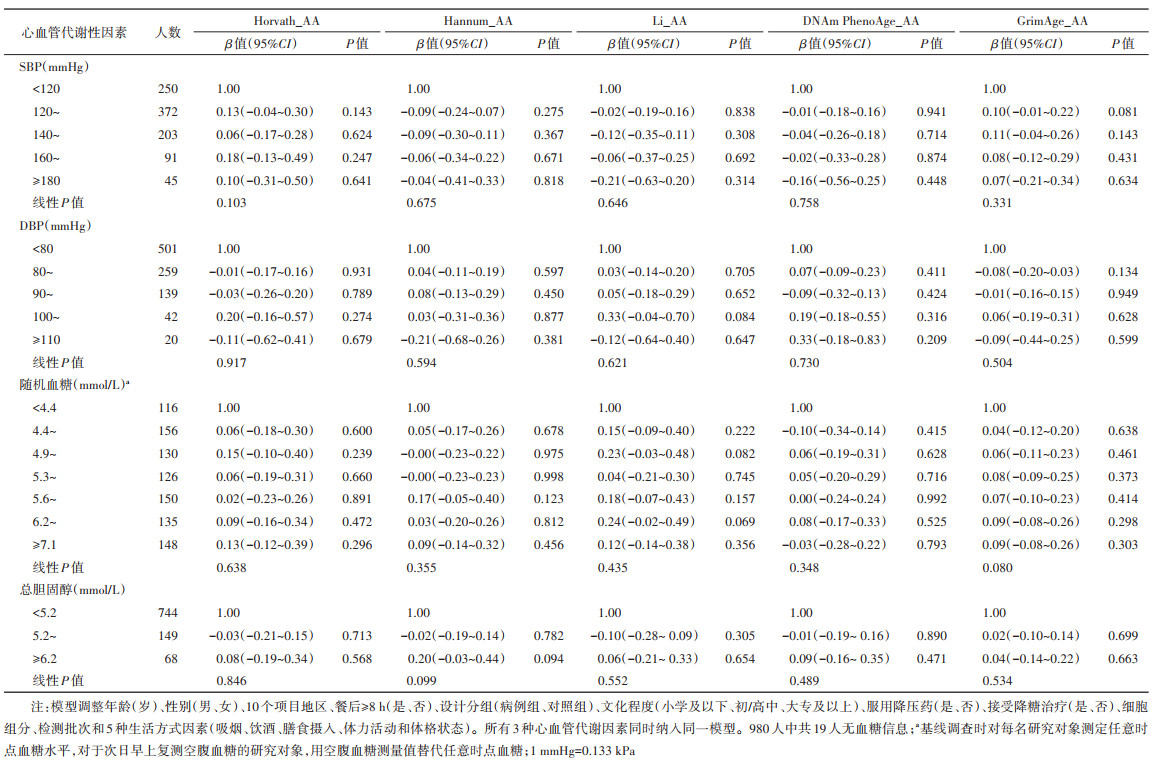
3. 甲基化年龄加速与单个生活方式因素的相关性:调整协变量后5种甲基化年龄加速与各个生活方式因素的相关性,见表 2。以GrimAge_AA为例,在当前吸烟者中,随每日吸烟量增加,GrimAge_AA呈上升趋势(线性P=0.040);与不吸烟者相比,吸烟量≥25支者的GrimAge_AA增加约1.0个标准差(P < 0.001)。与不饮酒者相比,每日饮纯乙醇量≥60 g者的GrimAge_AA增加约0.3个标准差(P=0.003)。而中等水平体力活动水平可降低GrimAge_AA。相比正常体重者,BMI < 18.5 kg/m2者的GrimAge_AA增加。Horvath_AA、Hannum_AA和DNAm PhenoAge_AA仅在个别生活方式类别表现出有统计学显著性的相关。Li_AA未见与各生活方式因素存在有统计学显著性的相关。
4. 甲基化年龄加速与单个心血管代谢因素的相关性:见表 3。各甲基化年龄加速与单个心血管代谢因素的相关性均差异无统计学意义。
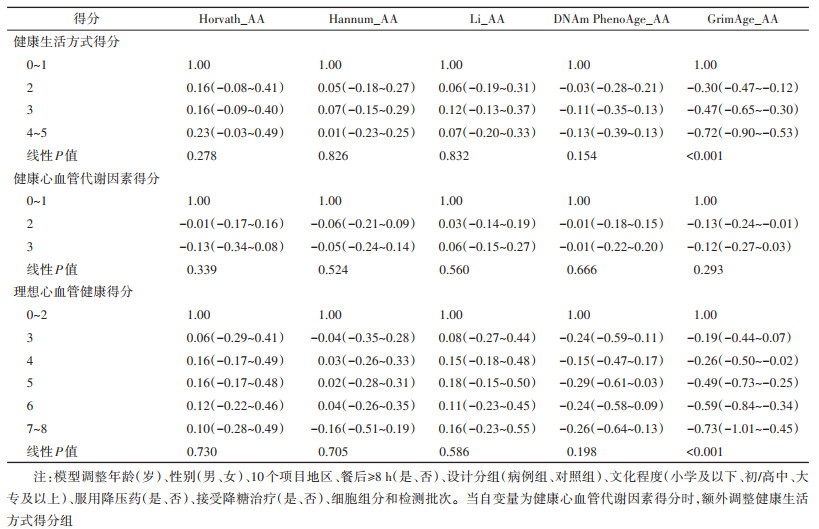
5. 甲基化年龄加速与健康得分的相关性:GrimAge_AA与健康生活方式得分呈负相关,随着健康生活方式得分的增加,GrimAge_AA呈现下降趋势(线性P < 0.001)。相比健康心血管代谢因素得分为0~1分组,2分或3分组的GrimAge_AA均降低,2分组表现出差异有统计学意义(P=0.035)。当整合生活方式和代谢因素得到理想心血管健康得分时,≥4分者GrimAge_AA增加,差异有统计学意义,GrimAge_AA与理想心血管健康得分呈负相关(线性P < 0.001)。见表 4。
本研究参照既往研究构建了5种甲基化年龄。结果显示,各甲基化年龄均可较好地拟合实足年龄。GrimAge_AA与吸烟、饮酒、体力活动水平及BMI存在关联,吸烟者、重度饮酒者、低体力活动水平者及低BMI者表现为加速衰老。随着健康生活方式得分的增加,GrimAge_AA呈现下降趋势。而其他4个指标与多数生活方式因素不存在有统计学显著性的关联。5种甲基化年龄加速指标与血压、血糖和TC均不存在关联。
Lu等[3]在构建GrimAge预测模型时,首先筛选出与某些血浆蛋白质或吸烟数密切相关的CpG位点;其中的血浆蛋白质在既往研究中发现与死亡风险存在关联。随后,再利用这些位点构建年龄预测模型并与死亡等结局进行关联分析。本研究中GrimAge_AA与每日吸烟量的正相关印证了该指标的构建基础,也与既往研究结果一致[29]。既往白人或非裔美国人的横断面研究发现饮酒量与GrimAge_AA存在线性相关,但方向不一致[3, 29];可能的原因包括不同种族间乙醇代谢能力[30]及其对甲基化组的影响[29, 31],以及不同研究人群饮酒量的变异范围不同。Beach等[32]则发现饮酒量与衰老速率间存在非线性关联,低饮酒量和重度饮酒都会表现为生物学衰老加速,而中间水平的摄入量表现为延缓的生物学衰老。
Lu等[3]在女性健康倡议(WHI)队列4 200名绝经后女性中的分析发现,部分食物摄入量与加速衰老的关联因种族而异,如在非西班牙裔白人中,蔬菜水果的摄入量增加可减缓GrimAge_AA,而在非裔美国人和西班牙裔中则无此发现。本研究未发现几大类食物组的摄入与GrimAge_AA存在相关,可能是因为只采集了食物组的每周摄入频率,没有每日摄入量的信息。Lu等[3]在WHI队列中还发现体力活动水平与加速衰老的关联也因种族而异。Kankaanpää等[33]报告休闲性体力活动可减缓GrimAge衡量的加速衰老,而职业性体力活动则相反。本研究人群中,与总体力活动水平低者相比,中、高水平者的GrimAge_AA降低,但线性趋势检验差异无统计学意义;中国人群中职业性体力活动占主要部分,可能对结果有一定程度的影响。
许多西方研究显示体格测量指标与GrimAge_AA呈正相关[3, 29, 34-35]。当BMI按分类变量进行分析时,一项美国女性研究在超重和肥胖组中观察到加速衰老,低体重组与正常体重组未见有统计学显著的差异[34]。韩国的一项横断面研究未发现BMI与GrimAge_AA相关[36]。而在本研究中,低体重组的GrimAge_AA大于正常体重组;而超重和肥胖组与正常体重组未见差异有统计学意义。
既往横断面研究观察到的心血管代谢性因素与GrimAge_AA的关联结果也不一致[3, 35-38]。以SBP为例,在WHI研究的非西班牙裔白人中,SBP上升表现为GrimAge_AA加速,在非裔美国人和西班牙裔人中则无此发现[3]。而Ammous等[38]则在非裔美国人中观察到SBP与GrimAge_AA存在正相关。
一项系统综述分析了2018年前发表的61项关于生活方式、环境或健康因素与Horvath时钟或Hannum时钟的关联研究,共纳入5万余人。结果发现Horvath时钟或Hannum时钟均与BMI呈正关联,但与吸烟不存在关联;对于其他生活方式因素,不同研究结果不一致,难以得出结论[39]。Pottinger等[9]在WHI研究中根据膳食、体力活动、吸烟、BMI、血压、FPG和TC构建理想心血管健康得分,发现该得分与Horvath_AA相关,得分每增加1分,年龄小2个月。Peng等[10]在143对中国双生子中也发现健康生活方式得分与Li_AA呈负关联,但关联强度较弱。
然而,本研究中未观察到GrimAge以外的其他4种甲基化年龄指标与多数生活方式因素、心血管代谢性因素和综合得分间存在有统计学显著性的关联。对于这个结果还需谨慎解读,主要考虑有以下两个方面。第一,Horvath等[1]指出Horvath时钟和Hannum时钟等传统甲基化年龄更多反映了受遗传调控的内在衰老过程,可能与生活方式因素以及心血管代谢性因素的相关性微弱。而DNAm PhenoAge和GrimAge的构建过程额外考虑了生物标志或吸烟等与死亡风险相关的信息,是内在基因调控与外源性应激综合作用的结果,可能会与生活方式及心血管代谢性因素表现出更强的相关性[1, 3]。第二,本研究分析的几种甲基化年龄在构建人群中所用的甲基化检测芯片各不相同,而不同芯片覆盖的CpG位点有所差异,本研究使用850 K芯片检测,且仅纳入经过质控后的位点计算甲基化年龄,纳入位点数与构建所需位点数存在一定的差异。但是,甲基化年龄计算平台具有探针插补功能[40]。有研究显示,虽然850 K芯片分别缺失了19和6个构建Horvath时钟和Hannum时钟所用的CpG位点,但是并不影响甲基化年龄的精度[41]。
本研究比较分析了5种甲基化年龄加速指标与生活方式因素、心血管代谢因素及其组合之间的关联,既往未见这样全面的分析。研究基于横断面设计,所有分析的因素都是基线调查时的状态。然而,本研究分析中纳入的研究对象在基线时不患有缺血性心脏病、脑卒中、恶性肿瘤等重大疾病,所以因病改变生活方式的可能性小,一定程度避免了因果倒置。本研究基于GrimAge_AA指标发现生活方式不健康者表现出表观遗传年龄的加速,即DNA甲基化预测年龄老于实足年龄。这一结果从不同角度为普及健康生活的必要性提供了证据支持。未来可采取前瞻性研究设计在更大样本人群中开展更多环境因素与DNA甲基化年龄和其他衰老指标的关联研究。
利益冲突 所有作者声明无利益冲突
作者贡献声明 陈璐:撰写文章、分析数据;司佳卉:分析数据、解释数据、修改文章;孙点剑一、余灿清:采集数据、解释数据、修改文章;郭彧、陈君石、陈铮鸣、李立明:采集数据、项目管理、经费支持;裴培:采集数据;吕筠:构思研究、采集数据、解释数据、修改文章、项目管理、经费支持
志谢 感谢所有参加中国慢性病前瞻性研究项目的队列成员和各项目地区的现场调查队调查员;感谢项目管理委员会、国家项目办公室、牛津协作中心和10个项目地区办公室的工作人员
| [1] |
Horvath S, Raj K. DNA methylation-based biomarkers and the epigenetic clock theory of ageing[J]. Nat Rev Genet, 2018, 19(6): 371-384. DOI:10.1038/s41576-018-0004-3 |
| [2] |
Li X, Ploner A, Wang YZ, et al. Longitudinal trajectories, correlations and mortality associations of nine biological ages across 20-years follow-up[J]. eLife, 2020, 9: e51507. DOI:10.7554/eLife.51507 |
| [3] |
Lu AT, Quach A, Wilson JG, et al. DNA methylation GrimAge strongly predicts lifespan and healthspan[J]. Aging, 2019, 11(2): 303-327. DOI:10.18632/aging.101684 |
| [4] |
Horvath S. DNA methylation age of human tissues and cell types[J]. Genome Biol, 2013, 14(10): R115. DOI:10.1186/gb-2013-14-10-r115 |
| [5] |
Hannum G, Guinney J, Zhao L, et al. Genome-wide methylation profiles reveal quantitative views of human aging rates[J]. Mol Cell, 2013, 49(2): 359-367. DOI:10.1016/j.molcel.2012.10.016 |
| [6] |
Levine ME, Lu AT, Quach A, et al. An epigenetic biomarker of aging for lifespan and healthspan[J]. Aging, 2018, 10(4): 573-591. DOI:10.18632/aging.101414 |
| [7] |
Li J, Zhu XY, Yu K, et al. Exposure to polycyclic aromatic hydrocarbons and accelerated DNA methylation aging[J]. Environ Health Perspect, 2018, 126(6): 067005. DOI:10.1289/EHP2773 |
| [8] |
Quach A, Levine ME, Tanaka T, et al. Epigenetic clock analysis of diet, exercise, education, and lifestyle factors[J]. Aging, 2017, 9(2): 419-446. DOI:10.18632/aging.101168 |
| [9] |
Pottinger TD, Khan SS, Zheng YN, et al. Association of cardiovascular health and epigenetic age acceleration[J]. Clin Epigenet, 2021, 13(1): 42. DOI:10.1186/s13148-021-01028-2 |
| [10] |
Peng HX, Gao WJ, Cao WH, et al. Combined healthy lifestyle score and risk of epigenetic aging: a discordant monozygotic twin study[J]. Aging, 2021, 13(10): 14039-14052. DOI:10.18632/aging.203022 |
| [11] |
Chen ZM, Lee LM, Chen JS, et al. Cohort profile: the kadoorie study of chronic disease in China (KSCDC)[J]. Int J Epidemiol, 2005, 34(6): 1243-1249. DOI:10.1093/ije/dyi174 |
| [12] |
Chen ZM, Chen JS, Collins R, et al. China kadoorie biobank of 0.5 million people: survey methods, baseline characteristics and long-term follow-up[J]. Int J Epidemiol, 2011, 40(6): 1652-1666. DOI:10.1093/ije/dyr120 |
| [13] |
李立明, 吕筠, 郭彧, 等. 中国慢性病前瞻性研究: 研究方法和调查对象的基线特征[J]. 中华流行病学杂志, 2012, 33(3): 249-255. Li LM, Lv J, Guo Y, et al. The China Kadoorie Biobank: related methodology and baseline characteristics of the participants[J]. Chin J Epidemiol, 2012, 33(3): 249-255. DOI:10.3760/cma.j.issn.0254-6450.2012.03.001 |
| [14] |
王昕, 吕筠, 郭彧, 等. 中国慢性病前瞻性研究: 10个项目地区成年人群吸烟行为特征差异分析[J]. 中华流行病学杂志, 2015, 36(11): 1200-1204. Wang X, Lv J, Guo Y, et al. Regional differences in adults' smoking pattern: findings from China Kadoorie Biobank study in 10 areas in China[J]. Chin J Epidemiol, 2015, 36(11): 1200-1204. DOI:10.3760/cma.j.issn.0254-6450.2015.11.004 |
| [15] |
吕筠, 郭彧, 卞铮, 等. 中国慢性病前瞻性研究: 10个项目地区人群饮酒行为特征差异的分析[J]. 中华流行病学杂志, 2014, 35(8): 875-881. Lv J, Guo Y, Bian Z, et al. Regional differences in patterns of alcohol consumption: findings from the China Kadoorie Biobank study on half a million people from 10 regions[J]. Chin J Epidemiol, 2014, 35(8): 875-881. DOI:10.3760/cma.j.issn.0254-6450.2014.08.001 |
| [16] |
樊萌语, 吕筠, 郭彧, 等. 中国慢性病前瞻性研究: 10个项目地区成人体力活动和休闲静坐时间特征差异的分析[J]. 中华流行病学杂志, 2015, 36(8): 779-785. Fan MY, Lv J, Guo Y, et al. Regional differences on patterns of physical activity and leisure sedentary time: findings from the China Kadoorie Biobank study, including a million people from 10 regions[J]. Chin J Epidemiol, 2015, 36(8): 779-785. DOI:10.3760/cma.j.issn.0254-6450.2015.08.002 |
| [17] |
刘琪, 吴曼, 温俏睿, 等. 中国10个地区成年人膳食模式与低肌肉重量、力量和质量的相关性分析[J]. 中华流行病学杂志, 2021, 42(5): 780-786. Liu Q, Wu M, Wen QR, et al. The correlation of dietary patterns with low muscle mass, strength and quality in adults from 10 regions of China[J]. Chin J Epidemiol, 2021, 42(5): 780-786. DOI:10.3760/cma.j.cn112338-20200618-00855 |
| [18] |
田园, 杨淞淳, 余灿清, 等. 中国成年人中心性肥胖与缺血性心脏病发病风险的前瞻性研究[J]. 中华流行病学杂志, 2018, 39(9): 1172-1178. Tian Y, Yang SC, Yu CQ, et al. Association between central obesity and risk for heart disease in adults in China: a prospective study[J]. Chin J Epidemiol, 2018, 39(9): 1172-1178. DOI:10.3760/cma.j.issn.0254-6450.2018.09.006 |
| [19] |
中华人民共和国国家卫生和计划生育委员会. WS/T 428-2013成人体重判定[M]. 北京: 中国标准出版社, 2013. National Health and Family Planning Commission of the People's Republic of China. WS/T 428-2013 Criteria of weight for adults[S]. Beijing: Standards Press of China, 2013. |
| [20] |
Lv J, Yu CQ, Guo Y, et al. Adherence to healthy lifestyle and cardiovascular diseases in the Chinese population[J]. J Am College Cardiol, 2017, 69(9): 1116-1125. DOI:10.1016/j.jacc.2016.11.076 |
| [21] |
Lv J, Yu CQ, Guo Y, et al. Adherence to a healthy lifestyle and the risk of type 2 diabetes in Chinese adults[J]. Int J Epidemiol, 2017, 46(5): 1410-1420. DOI:10.1093/ije/dyx074 |
| [22] |
祝楠波, 周密, 余灿清, 等. 中国成年人健康生活方式状况分析[J]. 中华流行病学杂志, 2019, 40(2): 136-141. Zhu NB, Zhou M, Yu CQ, et al. Prevalence of 'healthy lifestyle' in Chinese adults[J]. Chin J Epidemiol, 2019, 40(2): 136-141. DOI:10.3760/cma.j.issn.0254-6450.2019.02.003 |
| [23] |
Lloyd-Jones DM, Hong YL, Labarthe D, et al. Defining and setting national goals for cardiovascular health promotion and disease reduction: the American Heart Association's strategic Impact Goal through 2020 and beyond[J]. Circulation, 2010, 121(4): 586-613. DOI:10.1161/CIRCULATIONAHA.109.192703 |
| [24] |
万学红, 卢雪峰. 诊断学[M]. 9版. 北京: 人民卫生出版社, 2018. Wan XH, Lu XF. Diagnostics[M]. 9thed. Beijing: People's Medical Publishing House, 2018. |
| [25] |
Pidsley R, Zotenko E, Peters TJ, et al. Critical evaluation of the illumina methylation EPIC Bead Chip microarray for whole-genome DNA methylation profiling[J]. Genome Biol, 2016, 17(1): 208. DOI:10.1186/s13059-016-1066-1 |
| [26] |
Si JH, Yang SC, Sun DJY, et al. Epigenome-wide analysis of DNA methylation and coronary heart disease: a nested case-control study[J]. eLife, 2021, 10: e68671. DOI:10.7554/eLife.68671 |
| [27] |
Houseman EA, Accomando WP, Koestler DC, et al. DNA methylation arrays as surrogate measures of cell mixture distribution[J]. BMC Bioinformatics, 2012, 13: 86. DOI:10.1186/1471-2105-13-86 |
| [28] |
Dhingra R, Nwanaji-Enwerem JC, Samet M, et al. DNA methylation age-environmental influences, health impacts, and its role in environmental epidemiology[J]. Curr Environ Health Rep, 2018, 5(3): 317-327. DOI:10.1007/s40572-018-0203-2 |
| [29] |
Zhao W, Ammous F, Ratliff S, et al. Education and lifestyle factors are associated with DNA methylation clocks in Older African Americans[J]. Int J Environ Res Public Health, 2019, 16(17): 3141. DOI:10.3390/ijerph16173141 |
| [30] |
Chan AW. Racial differences in alcohol sensitivity[J]. Alcohol Alcohol, 1986, 21(1): 93-104. |
| [31] |
Liu C, Marioni RE, Hedman ÅK, et al. A DNA methylation biomarker of alcohol consumption[J]. Mol Psychiatry, 2018, 23(2): 422-433. DOI:10.1038/mp.2016.192 |
| [32] |
Beach SRH, Dogan MV, Lei MK, et al. Methylomic aging as a window onto the influence of lifestyle: tobacco and alcohol use alter the rate of biological aging[J]. J Am Geriatr Soc, 2015, 63(12): 2519-2525. DOI:10.1111/jgs.13830 |
| [33] |
Kankaanpää A, Tolvanen A, Bollepalli S, et al. Leisure-time and occupational physical activity associates differently with epigenetic aging[J]. Med Sci Sports Exerc, 2021, 53(3): 487-495. DOI:10.1249/MSS.0000000000002498 |
| [34] |
Kresovich JK, Garval EL, Martinez Lopez AM, et al. Associations of body composition and physical activity level with multiple measures of epigenetic age acceleration[J]. Am J Epidemiol, 2021, 190(6): 984-993. DOI:10.1093/aje/kwaa251 |
| [35] |
Arpón A, Milagro FI, Santos JL, et al. Interaction among sex, aging, and epigenetic processes concerning visceral fat, insulin resistance, and dyslipidaemia[J]. Front Endocrinol, 2019, 10: 496. DOI:10.3389/fendo.2019.00496 |
| [36] |
Lee HS, Park T. The influences of DNA methylation and epigenetic clocks, on metabolic disease, in middle-aged Koreans[J]. Clin Epigenet, 2020, 12(1): 148. DOI:10.1186/s13148-020-00936-z |
| [37] |
Roshandel D, Chen Z, Canty AJ, et al. DNA methylation age calculators reveal association with diabetic neuropathy in type 1 diabetes[J]. Clin Epigenet, 2020, 12(1): 52. DOI:10.1186/s13148-020-00840-6 |
| [38] |
Ammous F, Zhao W, Ratliff SM, et al. Epigenetic age acceleration is associated with cardiometabolic risk factors and clinical cardiovascular disease risk scores in African Americans[J]. Clin Epigenet, 2021, 13(1): 55. DOI:10.1186/s13148-021-01035-3 |
| [39] |
Ryan J, Wrigglesworth J, Loong J, et al. A systematic review and meta-analysis of environmental, lifestyle, and health factors associated with DNA methylation age[J]. J Gerontol Ser A, 2020, 75(3): 481-494. DOI:10.1093/gerona/glz099 |
| [40] |
Dhingra R, Kwee LC, Diaz-Sanchez D, et al. Evaluating DNA methylation age on the Illumina MethylationEPIC Bead Chip[J]. PLoS One, 2019, 14(4): e0207834. DOI:10.1371/journal.pone.0207834 |
| [41] |
McEwen LM, Jones MJ, Lin DTS, et al. Systematic evaluation of DNA methylation age estimation with common preprocessing methods and the Infinium MethylationEPIC BeadChip array[J]. Clin Epigenet, 2018, 10(1): 123. DOI:10.1186/s13148-018-0556-2 |
 2022, Vol. 43
2022, Vol. 43