文章信息
- 江河, 冯毅, 邵一鸣.
- Jiang He, Feng Yi, Shao Yiming
- HIV传播网络分析方法及测量指标研究进展
- Progress in research of HIV transmission network analysis methods and metrics
- 中华流行病学杂志, 2022, 43(1): 123-127
- Chinese Journal of Epidemiology, 2022, 43(1): 123-127
- http://dx.doi.org/10.3760/cma.j.cn112338-20211116-00888
-
文章历史
收稿日期: 2021-11-16
2. 中国疾病预防控制中心性病艾滋病预防控制中心传染病预防控制国家重点实验室,北京102206
2. State Key Laboratory of Infectious Disease Prevention and Control, National Center for AIDS/STD Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
HIV通过无保护的性行为和共用注射器吸毒等传播途径主要在高危人群中传播。由于HIV传播网络遵循结构化和非随机化的模式,网络成员的位置和网络结构对HIV传播的贡献大于个体的社会人口学和行为学特征[1]。HIV传播网络分析从群体角度分析HIV的传播机制,为精准防控艾滋病提供了新的思路和方法。虽然社会网络及相应的测量指标已广泛应用于识别HIV高风险传染源和易感人群,但局限性在于难以触及到隐蔽的高危人群。随着分子流行病学的发展,HIV分子网络和新的测量指标迅速应用于艾滋病防控领域。但该方法也存在局限性,需要社会网络进一步验证。目前尚无统一的HIV传播网络分析标准用于指导艾滋病防控。本文综述了HIV传播网络分析方法的选择、网络测量指标的定义、解释和应用,为深入研究HIV传播规律和制定精准防控措施提供参考依据。
一、HIV传播网络分析方法的选择狭义上的HIV传播网络仅包括HIV感染者。研究HIV感染者及其传播链的目的是阻断未来可能出现的传播链和减少新的HIV感染者,因此,完整的HIV传播网络应覆盖到尚未感染HIV的高危人群组成的风险网络。由于宿主的社交行为和病毒毒株进化均与病毒传播相关,HIV传播网络分析可以基于社会网络和分子网络开展。
1. 基于社会网络的分析方法:社会网络由社会成员及其之间的关系链组成。网络信息主要通过现场问卷调查[2]或网络社交平台问卷调查[3]收集,提名生成法(name generator)和提名诠释法(name interpreter)是较常用的调查方法[4]。具体到HIV传播网络研究领域,提名生成法是请受访者列举出与其发生危险行为的同伴,提名诠释法则是在提名同伴的基础上调查该同伴的社会属性和行为特征。由于研究对象包括HIV阴性者、既往确诊和尚未确诊的HIV感染者,社会网络的优点是可以超越狭义HIV传播网络的界限,进一步研究风险网络(图 1A),从而制定保护易感人群的精准干预措施[5]。社会网络分析的缺点是易产生选择偏倚[6]。由于发生艾滋病危险行为的个体和地点较隐蔽,调查采用滚雪球抽样[7]、同伴推断抽样[8]和方便抽样[9]等不同抽样方式开展,难以准确识别HIV传播网络的核心成员。

|
| 注:A:分子网络、传播网络和风险网络的关系;B:节点a在星形网络中处于核心位置;C:节点a在树形网络中处于核心位置 图 1 HIV传播网络的组成及网络拓扑结构对节点a位置重要性的影响 |
2. 基于分子网络的分析方法:分子网络根据HIV序列的遗传学相似性构建。当HIV序列的基因距离小于设定阈值时,即可将宿主连接成网。虽然分子网络应用于推断HIV传播网络晚于社会网络,但发展速度远快于社会网络。中国和美国均发布了HIV传播网络监测指南,推荐使用常规耐药监测收集的HIV序列构建分子网络,识别近期出现的快速传播簇。分子网络的优点:能整合不同来源的序列库,从空间、时间和人群层面上宏观地推断HIV传播网络。我国学者通过4次全国HIV分子流行病学调查收集的HIV序列,分别构建了CRF07_BC、CRF08_BC和CRF55_01B等重组亚型的流行毒株分子网络,展示了HIV的跨省传播关系图和传播路线[10-12]。如果将本地有限的HIV序列与国际上已发表的HIV序列合并,还能推断本地HIV流行毒株的来源[13]。分子网络的缺点:①病毒基因连接表示宿主间的感染密切相关,但无法判断是否存在中介传播者;②基因距离不代表传播关系紧密程度和病毒传播方向[14]。
3. 应用分子网络分析结果的社会网络实践方法:在现实世界的HIV传播网络中,社会网络分析包含了已发生、新发生或尚未发现、将要发生的传播事件,分子网络分析仅包含已发生的传播事件。根据分子网络分析的结果指导社会网络分析及时发现新近的传播事件,并阻止预期的传播事件发生,将是精准防控艾滋病的全新思路。首先,社会-分子联合网络分析表明,分子网络分析能帮助社会网络分析发现和梳理复杂的危险行为关系[15-16],并进一步预测HIV传播网络的增长趋势[17]。其次,近年来在世界各地广泛开展了应用分子网络分析推断HIV传播网络的研究。Chen等[18]在中国广西壮族自治区贵港市农村地区收集暗娼和老年嫖客HIV感染者血样及流行病学资料构建HIV分子网络,展示了该地区老年人的性交易网络。de Oliveira等[19]对南非2个邻近社区15~49岁普通人群开展HIV检测与收集HIV序列构建其分子网络,结果显示当地HIV主要流行方式为较年长男性HIV感染者向较年轻女性传播。Stecher等[20]发现德国科隆中部地区是HIV在邻近区域相互传播的重要枢纽。Hassan等[21]认为在HIV感染者的分子网络中聚集成簇者具有相似的抗病毒治疗效果。分子网络从重点人群、社区、地理空间、抗病毒治疗随访观察等方面分析,为进一步开展社会网络实践提供重要线索。另外,以HIV基因型耐药位点为标志物,分子网络还可研究HIV基因型耐药在人群中的传播规律,指导临床实践。目前我国已建成全球最大的HIV耐药监测网络,包括1个亚太区域级实验室、4个核心实验室和31个省级实验室[22]。虽然我国艾滋病抗病毒治疗前耐药的总体流行率为3.6%[23],但地区和人群间的流行率差异较大,西部少数民族地区和吸毒人群是制定优化用药方案的重点关注人群[24]。
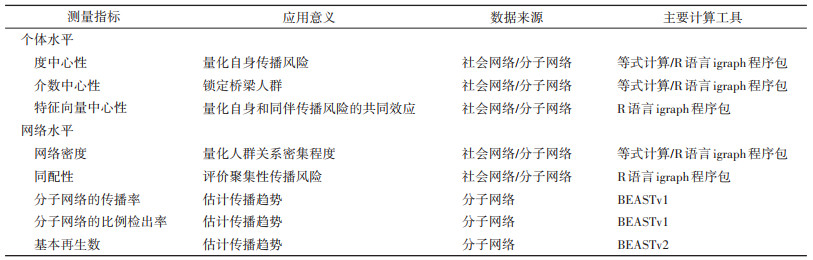
二、网络测量指标的概念和应用网络分析的3个重要步骤依次是绘制网络图,描述网络特性和探索网络发展趋势[6],因此选择合理的测量指标对网络分析至关重要。在网络图中,节点和边分别表示网络成员和他们的相互连接,网络测量指标多基于两者计算。本文总结近期HIV传播网络研究中使用的经典测量指标(表 1),并解释和说明其流行病学应用意义。
1. 个体水平测量指标:
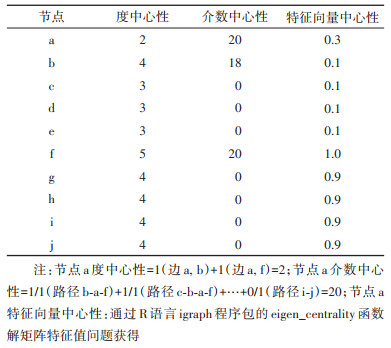
(1)度中心性(degree centrality,cD):与目标节点连接的边数总和。在网络分析中,用于量化目标节点与邻近节点的关联程度。公式:
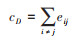
式中,eij为节点i与任意不同节点连接的边。
在HIV传播网络分析的应用中,度中心性指个体拥有的危险行为同伴数,是量化自身传播风险最直观的测量指标[25],Pang等[26]认为,分子网络中88.9%的MSM度中心性≥2,73.8%的MSM度中心性≥4。但是,现实世界的HIV传播网络不仅有简单的星形网络结构(图 1B),更多的是复杂的树形网络结构(图 1C)。这时还需引入合适的复杂网络的测量指标。
(2)介数中心性(betweenness centrality,cB):将目标节点在任意2个节点连接的最短路径中出现的概率相加,用于量化该节点连接子网络的能力指标。公式:
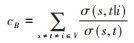
式中,σ(s,t|i)表示节点s通过i连接t的最短路径(边最少),σ(s,t)表示节点s连接t的最短路径。
在图 1C所示的树形网络中,节点a将2个独立的小网络连接成较大的网络。路径b-a-f是连接节点b和f的唯一路径,节点a在该路径中出现的概率是1。而节点b到c的最短路径是b-c,此时节点a出现的概率为0。将这些概率相加即可得到节点a的介数中心性,其他节点的介数中心性也按该过程计算(表 2)。介数中心性常用于锁定主导HIV在不同社交圈的人群间传播的桥梁人群[27]。另外,Mulawa等[28]将介数中心性用于寻找最适合的社区健康教育员。该研究发现,度中心性越高的健康教育员从社区居民获取的艾滋病、性暴力等隐私信息越少,而介数中心性越高的健康教育员却能获取更多隐私信息。
度中心性和介数中心性的优点是量化了网络成员自身行为特点对HIV传播的影响,其局限性是未考虑复杂网络中邻近网络成员的对自身传播和感染风险影响,已有学者使用矩阵理论解决该局限性。
(3)特征向量中心性(eigenvector centrality,cE):一种使用矩阵理论的特征值和特征向量概念来量化个体与邻近网络成员共同中心性的指标。公式:
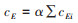
式中,α表示在解矩阵特征值问题中,经过迭代并达到稳态后得到的矩阵最大特征值。cEi是最大特征值对应的特征向量。
特征向量中心性受自身的度中心性和邻近网络成员的度中心性共同决定。对于自身度中心性低而邻近网络成员度中心性高的个体,特征向量中心性预示了其成为未来网络核心的潜力。Ragonnet-Cronin等[29]在英国、Yan等[30]在我国广州市分别开展的独立研究通过比较人群间的度中心性、介数中心性和特征向量中心性3个测量指标,强调MSM在HIV传播网络中的核心位置,以及不公开身份的MSM成为未来传播核心的可能性。
2. 网络水平测量指标:
(1)网络密度(density):测量网络成员关系密集程度的指标,指网络中实际存在的边数与最大期望边数的比值。公式:
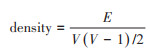
式中,E表示网络中所有的边,V表示网络中所有的节点。
由于不同人群的网络密度差异很大,单个调查缺乏可比性。Lee等[31]等在我国香港地区开展的纵向研究发现,近10年来MSM的网络密度持续增大,该结果解释了MSM的HIV感染率持续升高的原因。
(2)同配性(assortativity,ra):评价同类属性的节点是否倾向于相互连接的指标。公式:
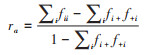
式中,fii 表示具有i属性的节点连接在邻接矩阵的全部连接中所占的比例,fi+是i属性节点对应的矩阵边际行之和,f+i是矩阵边际列之和。
同配性本质上是一种基于网络连接程度的皮尔森相关系数,取值在-1和1之间。①完全同配:1表示同属性节点完全连接;②完全异配:-1表示完全不连接;③0表示连接没有规律可循。在社会网络中,同配性体现了“物以类聚、人以群分”的社交特点。在HIV传播网络中,同配性较高提示聚集性传播[32],较低则提示HIV可能从高危人群向普通人群扩散[32-33]。
(3)分子网络的传播率(transmission rate,TR):估计分子簇演化趋势的指标,其中分子簇是组成分子网络的最小集团单位。公式:

公式的分子表示发生的传播事件,分母表示簇的观察人时。V表示分子簇包含的节点,t表示一个节点从出现到研究结束的时长,T表示分子簇从出现到研究结束的时长。每个节点出现的时间并非采样时间而是通过分子钟系统发育方法估算。
虽然艾滋病病例报告数据可以用于估计HIV传播趋势,但由于艾滋病潜伏期长,确诊时间不代表真实的HIV感染时间,该方法难以评估特定群体中发生的聚集性传播[34]。基于分子钟系统发育方法,用分子簇的演化趋势推断HIV传播趋势是传统流行病学方法的重要补充[35]。Oster等[36]从美国2013-2015年常规耐药监测中收集30 323条HIV序列,与基础HIV序列库合并构建分子网络。逐一研究全部73个分子簇后发现,其中的13个分子簇的TR远超其他分子簇,这些分子簇主要由西班牙裔或拉丁裔年轻MSM组成。
(4)分子网络的比例检出率(proportional detection rate,PDR):单位时间分子簇内节点增长比例来估计分子簇的演化趋势。公式:
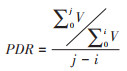
分子为分子簇内节点增长比例,分母为研究时长。V表示节点数,i表示研究开始年,j表示研究结束年。每个节点出现的时间均通过分子钟系统发育方法估算。
Dennis等[37]首先引入该指标,研究美国北卡罗来纳州2009-2014年HIV传播网络演变趋势发现,抗病毒治疗规模抑制了大多数分子簇,仅12.72%(288/2 264)的分子簇持续增长,其中15个分子簇的PDR≥2。该研究据此确定了优先干预目标人群。
(5)基本再生数(basic reproduction number,R0):在全由HIV易感者组成的人群中,1例HIV感染者在传染期内产生的平均二代传播数。公式:
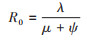
式中,λ表示传播率。μ表示感染者转变为非感染者的转化率。计算公式假设患者采样后即得到治疗和保持病毒抑制,转化为非感染者。ψ表示采样率。
虽然TR和PDR计算简便却未考虑采样率。相比之下,基于HIV序列的再生数(R)能更准确地评估HIV传播趋势[38]。R > 1提示传播持续扩散,R < 1提示传播逐渐消失。R的2个形式是R0和有效再生指数(Re),分别表示所有人和部分人为易感人群时,由一名感染者在传染期间产生的平均二代传播数。在HIV的研究中,R0与Re相等。Liu等[39]用PDR和R0比较分析早期抗病毒治疗对我国沈阳市区HIV传播动态的影响,研究发现2008-2016年绝大多数大型分子簇(≥10个节点)的增长减缓,另外,PDR适用于小型分子簇分析,R0适合于大型分子簇分析。
三、小结社会网络和分子网络分析方法已在艾滋病防控领域广泛应用。中国疾病预防控制中心性病艾滋病预防控制中心牵头组织和制定的《HIV传播网络监测和干预技术指南》(试行)于2019年9月发布[40],浙江省、四川省、广西壮族自治区、天津市、北京市、广东省和上海市等率先开展基于HIV分子网络的监测和干预试点工作。依赖于越加完善的HIV序列数据和目标人群流行病学资料,HIV分子网络分析具有较好的应用前景,高效发现高风险传染源和关键传播链,结合社会网络分析和临床实践,准确锁定优先干预的目标人群,实施艾滋病的精准预防措施和制定优化的抗病毒治疗方案。在实际应用中,应根据时间跨度、地区范围、人群特征和研究经验,应用合适的个体水平和网络水平的测量指标。
利益冲突 所有作者声明无利益冲突
| [1] |
Martin TCS, Chaillon A, Graves SK, et al. Genetic network analysis to assess the risk of human immunodeficiency virus transmission among men who have sex with men seeking partners on the internet[J]. Clin Infect Dis, 2020, 70(5): 925-932. DOI:10.1093/cid/ciz278 |
| [2] |
Kan M, Garfinkel DB, Samoylova O, et al. Social network methods for HIV case-finding among people who inject drugs in Tajikistan[J]. J Int AIDS Soc, 2018, 21(Suppl 5): e25139. DOI:10.1002/jia2.25139 |
| [3] |
Li L, Lin CQ, Feng N, et al. Using social media to enhance provider network for HIV and harm reduction service integration in Vietnam[J]. AIDS Behav, 2019, 23(11): 3175-3183. DOI:10.1007/s10461-019-02542-4 |
| [4] |
Shushtari ZJ, Hosseini SA, Sajjadi H, et al. Social network and HIV risk behaviors in female sex workers: a systematic review[J]. BMC Public Health, 2018, 18(1): 1020. DOI:10.1186/s12889-018-5944-1 |
| [5] |
Delva W, Leventhal GE, Helleringer S. Connecting the dots: network data and models in HIV epidemiology[J]. AIDS, 2016, 30(13): 2009-2020. DOI:10.1097/qad.0000000000001184 |
| [6] |
Luke DA, Harris JK. Network analysis in public health: history, methods, and applications[J]. Annu Rev Public Health, 2007, 28: 69-93. DOI:10.1146/annurev.publhealth.28.021406.144132 |
| [7] |
Friedland BA, Gottert A, Hows J, et al. The people living with HIV stigma index 2.0: generating critical evidence for change worldwide[J]. AIDS, 2020, 34(Suppl 1): S5-18. DOI:10.1097/qad.0000000000002602 |
| [8] |
Mcfall AM, Lau B, Latkin C, et al. Optimizing respondent-driven sampling to find undiagnosed HIV-infected people who inject drugs[J]. AIDS, 2021, 35(3): 485-494. DOI:10.1097/qad.0000000000002763 |
| [9] |
Maté T, Hoyos J, Guerras JM, et al. Potential of HIV self-sampling to increase testing frequency among gay, bisexual, and other men who have sex with men, and the role of online result communication: online cross-sectional study[J]. J Med Internet Res, 2020, 22(11): e21268. DOI:10.2196/21268 |
| [10] |
Ge ZW, Feng Y, Zhang H, et al. HIV-1 CRF07_BC transmission dynamics in China: two decades of national molecular surveillance[J]. Emerg Microbes Infect, 2021, 10(1): 1919-1930. DOI:10.1080/22221751.2021.1978822 |
| [11] |
Li K, Liu ML, Chen HH, et al. Using molecular transmission networks to understand the epidemic characteristics of HIV-1 CRF08_BC across China[J]. Emerg Microbes Infect, 2021, 10(1): 497-506. DOI:10.1080/22221751.2021.1899056 |
| [12] |
Gan MZ, Zheng S, Hao JJ, et al. The prevalence of CRF55_01B among HIV-1 strain and its connection with traffic development in China[J]. Emerg Microbes Infect, 2021, 10(1): 256-265. DOI:10.1080/22221751.2021.1884004 |
| [13] |
Rhee SY, Magalis BR, Hurley L, et al. National and international dimensions of human immunodeficiency virus-1 sequence clusters in a northern california clinical cohort[J]. Open Forum Infect Dis, 2019, 6(4): ofz135. DOI:10.1093/ofid/ofz135 |
| [14] |
Verhofstede C, Mortier V, Dauwe K, et al. Exploring HIV-1 transmission dynamics by combining phylogenetic analysis and infection timing[J]. Viruses, 2019, 11(12): 1096. DOI:10.3390/v11121096 |
| [15] |
Kostaki EG, Nikolopoulos GK, Pavlitina E, et al. Molecular analysis of human immunodeficiency virus type 1 (HIV-1)-infected individuals in a network-based intervention (transmission reduction intervention project): phylogenetics identify HIV-1-infected individuals with social links[J]. J Infect Dis, 2018, 218(5): 707-715. DOI:10.1093/infdis/jiy239 |
| [16] |
Wertheim JO, Pond SLK, Forgione LA, et al. Social and genetic networks of HIV-1 transmission in New York City[J]. PLoS Pathog, 2017, 13(1): e1006000. DOI:10.1371/journal.ppat.1006000 |
| [17] |
Wertheim JO, Murrell B, Mehta SR, et al. Growth of HIV-1 molecular transmission clusters in New York City[J]. J Infect Dis, 2018, 218(12): 1943-1953. DOI:10.1093/infdis/jiy431 |
| [18] |
Chen X, Qin CW, Chen RF, et al. Epidemiological profile and molecular genetic characterization of HIV-1 among female sex workers and elderly male clients in Guangxi, China[J]. Emerg Microbes Infect, 2021, 10(1): 384-395. DOI:10.1080/22221751.2021.1888659 |
| [19] |
de Oliveira T, Kharsany ABM, Gräf T, et al. Transmission networks and risk of HIV infection in KwaZulu-Natal, South Africa: a community-wide phylogenetic study[J]. Lancet HIV, 2017, 4(1): e41-50. DOI:10.1016/s2352-3018(16)30186-2 |
| [20] |
Stecher M, Hoenigl M, Eis-Hübinger AM, et al. Hotspots of transmission driving the local human immunodeficiency virus epidemic in the Cologne-Bonn Region, Germany[J]. Clin Infect Dis, 2019, 68(9): 1539-1546. DOI:10.1093/cid/ciy744 |
| [21] |
Hassan A, de Gruttola V, Hu YW, et al. The relationship between the human immunodeficiency virus-1 transmission network and the HIV care continuum in Los Angeles County[J]. Clin Infect Dis, 2020, 71(9): e384-391. DOI:10.1093/cid/ciaa114 |
| [22] |
汪宁, 钟平. 中国HIV分子流行病学30年[J]. 中华流行病学杂志, 2015, 36(6): 541-546. Wang N, Zhong P. Molecular epidemiology of HIV in China: 1985-2015[J]. Chin J Epidemio, 2015, 36(6): 541-546. DOI:10.3760/cma.j.issn.0254-6450.2015.06.001 |
| [23] |
Zhao S, Feng Y, Hu J, et al. Prevalence of transmitted HIV drug resistance in antiretroviral treatment na?ve newly diagnosed individuals in China[J]. Sci Rep, 2018, 8(1): 12273. DOI:10.1038/s41598-018-29202-2 |
| [24] |
Kang RH, Liang SJ, Ma YL, et al. Pretreatment HIV drug resistance in adults initiating antiretroviral therapy in China, 2017[J]. Infect Dis Poverty, 2020, 9(1): 54. DOI:10.1186/s40249-020-00668-5 |
| [25] |
Pines HA, Wertheim JO, Liu L, et al. Concurrency and HIV transmission network characteristics among MSM with recent HIV infection[J]. AIDS, 2016, 30(18): 2875-2883. DOI:10.1097/qad.0000000000001256 |
| [26] |
Pang XW, Wei H, Huang JH, et al. Patterns and risk of HIV-1 transmission network among men who have sex with men in Guangxi, China[J]. Sci Rep, 2021, 11(1): 513. DOI:10.1038/s41598-020-79951-2 |
| [27] |
Tieu HV, Liu TY, Hussen S, et al. Sexual networks and HIV risk among black men who have sex with men in 6 U.S. Cities[J]. PLoS One, 2015, 10(8): e0134085. DOI:10.1371/journal.pone.0134085 |
| [28] |
Mulawa MI, Yamanis TJ, Kajula LJ, et al. Structural network position and performance of health leaders within an HIV prevention trial[J]. AIDS Behav, 2018, 22(9): 3033-3043. DOI:10.1007/s10461-018-2126-1 |
| [29] |
Ragonnet-Cronin M, Hué S, Hodcroft EB, et al. Non-disclosed men who have sex with men in UK HIV transmission networks: phylogenetic analysis of surveillance data[J]. Lancet HIV, 2018, 5(6): e309-316. DOI:10.1016/s2352-3018(18)30062-6 |
| [30] |
Yan HC, He WY, Huang LP, et al. The central role of nondisclosed men who have sex with men in human immunodeficiency virus-1 transmission networks in Guangzhou, China[J]. Open Forum Infect Dis, 2020, 7(5): ofaa154. DOI:10.1093/ofid/ofaa154 |
| [31] |
Lee SS, Tam DKP, Ho RLM, et al. Social network methodology for studying HIV epidemiology in men having sex with men[J]. J Infect Public Health, 2009, 2(4): 177-183. DOI:10.1016/j.jiph.2009.09.002 |
| [32] |
Ragonnet-Cronin M, Hu YW, Morris SR, et al. HIV transmission networks among transgender women in Los Angeles County, CA, USA: a phylogenetic analysis of surveillance data[J]. Lancet HIV, 2019, 6(3): e164-172. DOI:10.1016/s2352-3018(18)30359-x |
| [33] |
Dong ZL, Gao GF, Lyu F. Advances in research of HIV transmission networks[J]. Chin Med J (Engl), 2020, 133(23): 2850-2858. DOI:10.1097/cm9.0000000000001155 |
| [34] |
Oster AM, France AM, Mermin J. Molecular epidemiology and the transformation of HIV prevention[J]. JAMA, 2018, 319(16): 1657-1658. DOI:10.1001/jama.2018.1513 |
| [35] |
Green N, Hoenigl M, Chaillon A, et al. Partner services in adults with acute and early HIV infection[J]. AIDS, 2017, 31(2): 287-293. DOI:10.1097/qad.0000000000001308 |
| [36] |
Oster AM, France AM, Panneer N, et al. Identifying clusters of recent and rapid HIV transmission through analysis of molecular surveillance data[J]. J Acquir Immune Defic Syndr, 2018, 79(5): 543-550. DOI:10.1097/qai.0000000000001856 |
| [37] |
Dennis AM, Hué S, Billock R, et al. Human immunodeficiency virus type 1 phylodynamics to detect and characterize active transmission clusters in North Carolina[J]. J Infect Dis, 2020, 221(8): 1321-1330. DOI:10.1093/infdis/jiz176 |
| [38] |
Stadler T, Kouyos R, von Wyl V, et al. Estimating the basic reproductive number from viral sequence data[J]. Mol Biol Evol, 2012, 29(1): 347-357. DOI:10.1093/molbev/msr217 |
| [39] |
Liu MC, Han XX, Zhao B, et al. Dynamics of HIV-1 molecular networks reveal effective control of large transmission clusters in an area affected by an epidemic of multiple HIV subtypes[J]. Front Microbiol, 2020, 11: 604993. DOI:10.3389/fmicb.2020.604993 |
| [40] |
中国疾病预防控制中心性病艾滋病预防控制中心. 我国《HIV传播网络监测和干预技术指南》(试行)正式发布[EB/OL]. (2019-09-29)[2021-11-01]. http://ncaids.chinacdc.cn/zxzx/zxdteff/201909/t20190929_205904.htm. National Center for AIDS/STD Control and Prevention, Chinese Center for Disease Control and Prevention. Guideline of monitor and intervention technique of HIV transmission network in China (trial version) has been issued[EB/OL]. (2019-09-29)[2021-11-01]. http://ncaids.chinacdc.cn/zxzx/zxdteff/201909/t20190929_205904.htm. |
 2022, Vol. 43
2022, Vol. 43