文章信息
- 严寒秋, 王永全, 崔海洋, 靳博, 高志勇, 王全意.
- Yan Hanqiu, Wang Yongquan, Cui Haiyang, Jin Bo, Gao Zhiyong, Wang Quanyi
- 应用2种RT-PCR方法检测和分析北京市市场销售牡蛎中诺如病毒基因特征
- Application of two RT-PCR methods for detection of norovirus in market-sold oysters and norovirus genetic characteristic analysis, a survey conducted in Beijing
- 中华流行病学杂志, 2022, 43(1): 92-97
- Chinese Journal of Epidemiology, 2022, 43(1): 92-97
- http://dx.doi.org/10.3760/cma.j.cn112338-20210519-00411
-
文章历史
收稿日期: 2021-05-19
2. 北京市西城区疾病预防控制中心, 北京 100120
2. Xicheng District Center for Disease Control and Prevention, Beijing 100120, China
诺如病毒共有3个开放阅读框(ORF1~ORF3),其中ORF2编码主要结构蛋白VP1。依据VP1基因特征,共分12个基因组,其中有10个已明确分类的基因组(GⅠ~GⅩ),有2个暂定的基因组(GNA1~ GNA2)。引起人感染的基因组主要是GⅡ和GⅠ[1]。牡蛎在滤食海水中的微型海藻生物和有机碎屑时,也过滤海水中微量的诺如病毒。10~100个诺如病毒粒子就可引起人体感染。牡蛎是引起诺如病毒急性胃肠炎的高风险食品之一,在欧洲等地区暴发的36起病毒性急性胃肠炎中,其中22起与食用被诺如病毒污染的牡蛎有关[2]。2018年报道实时荧光RT-PCR方法检测诺如病毒GⅠ/GⅡ组,其阳性标本用半巢式RT-PCR方法扩增诺如病毒GⅠ/GⅡ组衣壳蛋白区基因,对PCR阳性产物测序,分析其基因特征[3]。本研究应用实时荧光RT-PCR和半巢式RT-PCR方法对2014年11月至2015年10月北京市的市售牡蛎同时进行诺如病毒检测,以实时荧光RT-PCR方法为标准,用符合率和一致性检验(Kappa值)对半巢式RT-PCR方法进行可靠性评价,并对半巢式RT-PCR阳性产物进行测序,分析其基因特征,为诺如病毒防控工作提供参考依据。
材料与方法1. 样品来源:2014年11月至2015年10月,在北京市某出售水产品市场的固定摊位,每月固定日期采集新鲜牡蛎2次,每次采5~7只牡蛎为1份。每月6份,12个月共72份。4 ℃~8 ℃运送至实验室,-20 ℃及以下保存待检。
2. 样品前处理和病毒富集[4-5]:将冷冻的牡蛎提前放4 ℃~8 ℃过夜解冻,用流动自来水清洗牡蛎外壳污物,打开牡蛎,用灭菌剪子和镊子解剖牡蛎,剪取肠内容物和肠腺等消化组织放无菌平皿中。另取一灭菌的剪子将其充分剪碎并混匀,立即进行病毒富集。取(3.0±0.1)g剪碎物放均质袋中,加入21 ml甘氨酸/氯化钠溶液,用均质器以9次/s速度拍打2 min。取出组织匀浆物放入50 ml离心管中,加入5 ml氯仿充分振荡2 min。采用冷冻离心机10 000 r/min,离心半径7.5 cm,4 ℃离心30 min,取上清液放入50 ml离心管中,加等体积的聚乙二醇PEG8000(美国Sigma-Aldrich公司)充分振荡2 min,终浓度为8%。4 ℃ 16~20 h,使样本中病毒颗粒集聚发生沉淀。用冷冻离心机10 000 r/min,离心半径7.5 cm,4 ℃离心15 min,留沉淀立即进行核酸提取。
3. 病毒核酸提取和保存:取280 μl磷酸盐缓冲液(pH值7.0)将沉淀充分溶解,按QIAamp Viral RNA Mini Kit说明书进行病毒RNA提取,最终核酸洗脱体积为60 μl。在-70 ℃以下保存,用于诺如病毒检测。
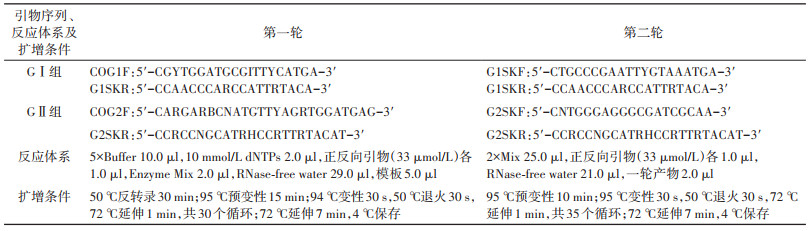
4. 实时荧光RT-PCR和半巢式RT-PCR方法检测诺如病毒[4, 6-7]:使用诺如病毒GⅠ/GⅡ组核酸检测试剂盒(江苏硕世生物科技有限公司生产)和7500实时荧光定量PCR仪(美国应用生物系统公司)对72份样品进行诺如病毒核酸检测,具体操作和结果判断按说明书进行。阳性表明检出诺如病毒。使用半巢式RT-PCR方法对72份样品分别扩增诺如病毒GⅠ和GⅡ组衣壳蛋白VP1区基因,使用One Step RT-PCR Kit[德国凯杰(QIAgen)生物公司生产]进行一轮扩增,AmpliTaq Gold® 360 Master Mix(美国生命技术公司)进行二轮扩增,引物序列和扩增参数见表 1。用QIAxcel全自动毛细管电泳仪[德国凯杰(QIAgen)生物公司生产]电泳,GⅠ和GⅡ组诺如病毒目的片段为330 bp和344 bp。
5. 半巢式RT-PCR方法可靠性评价[8]:以实时荧光RT-PCR为标准,采用符合率与Kappa值作为指标,对半巢式RT-PCR方法进行可靠性评价。
6. 基因型鉴定和进化分析:将半巢式RT-PCR扩增产物送生工生物工程(上海)股份有限公司进行测序。测序成功后,采用诺如病毒在线分型工具Norovirus Typing Tool Version 2.0(https://www.rivm.nl/mpf/typingtool/norovirus)进行基因分型和鉴定。采用BioEdit 7.0.9.0软件对序列进行编辑和比对。采用Mega 6.0软件的最大似然法(maximum likelihood)构建衣壳蛋白VP1区进化树,bootstrap值设置1 000次,诺如病毒GⅡ核酸替代模型选择K2+G。诺如病毒参考序列选自GenBank。
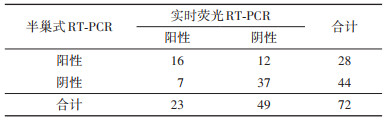
结果1. 诺如病毒检出情况:72份样品中,实时荧光RT-PCR、半巢式RT-PCR和并联试验的诺如病毒检出率分别为31.94%(23/72)、38.89%(28/72)和48.61%(35/72)。见表 2。
2. 半巢式RT-PCR方法可靠性:以实时荧光RT-PCR为标准,半巢式RT-PCR的符合率为73.61%(53/72),中度一致(Kappa值=0.43)。见表 2。
3. 诺如病毒基因组检出时间分布:72份样品检出诺如病毒35份,1和12月诺如病毒检出最多,各5份;春、夏、秋、冬季各检出7、7、8、13份。在检出的样品中,GⅡ15份,12月检出4份;春、夏、秋、冬季各检出2、4、2、7份。GⅠ 7份,9月检出3份;春、夏、秋、冬季各检出0、2、3、2份。GⅠ/GⅡ混合13份,5月检出4份;春、夏、秋、冬季各检出5、1、3、4份。见图 1。
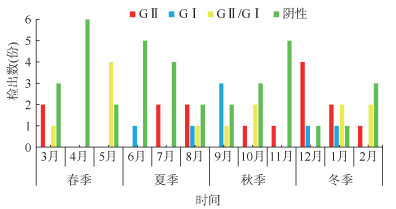
|
| 图 1 北京市市场销售牡蛎中诺如病毒基因组检出时间分布 |
4. 诺如病毒测序及分型鉴定:对28株半巢式RT-PCR阳性产物进行衣壳蛋白VP1区基因测序,共13株测序成功,用诺如病毒在线分型工具进行分型鉴定,同时将该序列在GenBank上进行BLAST序列比对,再次鉴定基因型。其中11株为2种RT-PCR方法检测诺如病毒阳性样本所得,7株GⅡ.17、2株GⅡ.4 Sydney_2012、1株GⅡ.1和1株GⅡ.21基因型;2株为半巢式RT-PCR方法检测诺如病毒阳性样本、实时荧光RT-PCR方法检测诺如病毒阴性样本所得,包括1株GⅡ.17和1株GⅡ.3基因型。将这些基因序列提交至GenBank数据库,获得序列号为MZ227509~MZ227521。
5. 诺如病毒GⅡ基因组序列进化分析:
(1)GⅡ.17型进化分析:GenBank下载GⅡ.17型参考株序列,与8株GⅡ.17型试验株序列进行同源性和进化分析。8株GⅡ.17型相似性为97.3%~99.6%,8株GⅡ.17型试验株与14株GⅡ.17型参考株相似性为84.4%~100.0%。这22株GⅡ.17型分为3个基因簇(ClusterⅠ~Ⅲ)。8株GⅡ.17型试验株与10株参考株同处于Cluster Ⅲ,相似性为97.3%~100.0%,其中XCML-46试验株与KT633395/Hu/2015/Beijing/CHN和MN461104/2016/ Sewage/ROK相似性均为100.0%,与其他8株GⅡ.17型参考株相似性均为99.3%~99.6%;XCML-58试验株与MT344181/Hu/2015/USA、LC433723/Hu/2015/JP、MN045200/Hu/2016/BRA、MK907791/Sewage/2014/FRA和MN461108/Sewage/2016/ROK相似性均为100.0%,与其他5株GⅡ. 17型参考株相似性为99.3%~99.6%;XCML-82试验株与KU557790/Hu/2014/Guangdong/CHN、MN904959/ Scallop/2014/Beijing/CHN和LC101784/Oyster/2015/JP相似性均为100.0%,与其他7株GⅡ.17型参考株相似性均为99.6%;其他5株试验株(XCML-121、XCML-85、XCML-55、XCML-52和XCML-43)与10株参考株相似性为97.3%~99.6%。8株GⅡ.17型试验株与位于ClusterⅠ的毒株JF970609和EF529741相似性介于84.4%~85.7%,与位于ClusterⅡ的毒株KR074152和KR074151相似性介于87.4%~89.0%。见图 2。
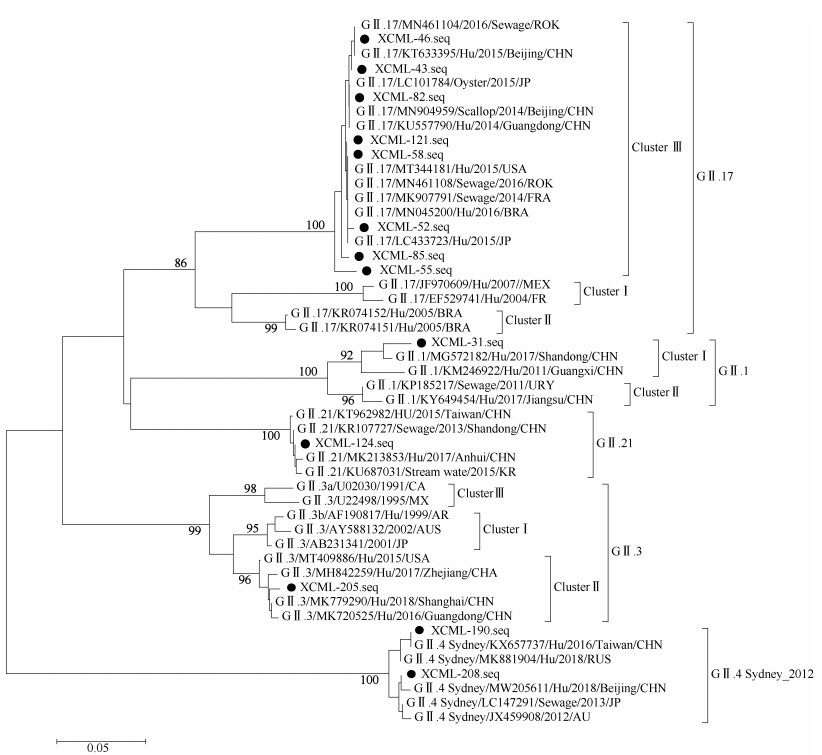
|
| 注:●本研究的诺如病毒毒株基因序列 图 2 北京市市场销售牡蛎中诺如病毒GⅡ组部分VP1区基因(302 bp)进化分析 |
(2)5株诺如病毒GⅡ其他型毒株的进化分析:从GenBank下载GⅡ.4 Sydney_2012、GⅡ.1、GⅡ.3和GⅡ.21型参考株序列进行同源性和进化分析。XCML-190和XCML-208试验株为GⅡ.4 Sydney_2012,二者序列相似性为98.0%,与5株GⅡ.4 Sydney_2012型参考株相似性为97.6%~100.0%,其中XCML-190试验株与KX657737/Hu/2016/Taiwan/CHN相似性为100.0%。XCML-31试验株与4株GⅡ.1型参考株相似性为94.0%~98.0%,与其中的参考株MG572182/Hu/2017/Shangdong/CHN相似性最高为98.0%。XCML-205试验株与9株GⅡ.3型参考株相似性为91.0%~99.3%,这10株GⅡ.3型分为3个基因簇(ClusterⅠ~Ⅲ)。该试验株与4株参考株同处于ClusterⅡ,相似性99.0%~99.3%,与其中的参考株MK779290/Hu/2018/Shangdong/CHN相似性最高为99.3%。该试验株与位于ClusterⅠ的毒株AY588132、AF190817和AB231341相似性介于95.3%~95.6%,与位于ClusterⅢ的毒株U02030和U22498相似性介于91.0%~92.0%。XCML-124试验株与4株GⅡ.21型参考株相似性为99.3%~99.6%,与其中的参考株KR107727/Sewage/2013/Shangdong/CHN相似性最高为99.6%。见图 2。
讨论目前分子生物学检测法已广泛应用于病毒检测[9-11],在诺如病毒的多种检测方法中[12-13],特别是RT-PCR方法是诺如病毒最常用的方法[14-17]。2002-2003年报道了用半巢式RT-PCR方法检测诺如病毒GⅠ和GⅡ,该方法比普通RT-PCR方法更为灵敏,特别适用于诺如病毒含量较低的样品检测[6-7]。随着科技的发展,实时荧光RT-PCR技术能更加快速准确检测诺如病毒核酸。实时荧光RT-PCR方法于2017年列入我国食品安全国家标准,是目前公认的诺如病毒金标准检测方法[4]。但是实时荧光RT-PCR阳性产物片段小,无法测序。
本研究发现,半巢式RT-PCR检出率(38.89%)高于实时荧光RT-PCR检出率(31.94%),但是对半巢式RT-PCR进行可靠性评价,符合率 < 80.00%,中度一致(Kappa值=0.43),说明在检测牡蛎中诺如病毒时,半巢式RT-PCR不能代替荧光RT-PCR方法。但是2种方法并联检出率(48.61%),分别比半巢式RT-PCR和实时荧光RT-PCR方法的检出率高了9.72%和16.67%。另外本研究共13株测序成功,11株为实时荧光RT-PCR和半巢式RT-PCR方法检测诺如病毒阳性样本所得,有GⅡ.17、GⅡ.4 Sydney_2012、GⅡ.21和GⅡ.1的4个基因型;2株为半巢式RT-PCR方法检测诺如病毒阳性而实时荧光RT-PCR方法检测阴性样本所得,有GⅡ.17和GⅡ.3的2个基因型。表明用2种RT-PCR同时对样品进行诺如病毒检测,任一种RT-PCR阳性说明检出诺如病毒,并联试验方法不仅提高了阳性检出率,还能获得更多基因型和序列。
应用实时荧光RT-PCR方法首先检测样品诺如病毒,其阳性再用半巢式RT-PCR方法扩增VP1区基因,阳性产物进行测序后获得基因型[3]。这一检测过程工作量少,试剂耗材的用量节省。应用2种RT-PCR方法同时对样品进行诺如病毒检测,工作量大,试剂耗材的用量较多。操作时间两者接近。建议根据实验室条件和样品来源选择检测方法:对病毒载量低的食品或水样品进行日常监督检测时,采用实时荧光RT-PCR方法。对病毒性急性胃肠炎疫情中病毒载量低的可疑食品或水等样品进行疫情溯源检测时,采用半巢式RT-PCR方法。
本研究中72份样品检出35份诺如病毒,测序成功13株诺如病毒,3株GⅡ.17型试验株(XCML-46、XCML-58和XCML-82)及1株GⅡ.4 Sydney_2012型(XCML-190)与不同国家不同年份的人源参考株、环境污水参考株和贝类水产品参考株(共10株)相似性100.0%,其他的9株试验株与人源、环境污水和贝类水产品的参考株相似性≥84.4%。在自然界中,诺如病毒在人群、环境及食品之间形成了一个闭环传播链,这与2020年的研究结论相似[18]。2020年在北京市开展的专项研究中,宾馆厨师的诺如病毒隐性感染率为0.99%,其环境污染阳性率为0.16%[19]。
综上所述,应用2种RT-PCR并联法检测牡蛎中诺如病毒,不仅能提高检出率还能获得更多基因型。牡蛎中诺如病毒毒株与人源、环境污水及贝类水产品参考毒株高度同源。因此,对引起诺如病毒急性胃肠炎的高风险食品监测时,还要对其生产、销售和加工等人群及相关环境进行诺如病毒监测和防控。
利益冲突 所有作者声明无利益冲突
作者贡献声明 严寒秋:研究设计、样品检测、论文撰写、试验数据分析、论文修改;王永全、崔海洋、靳博:现场实施采样、样品检测;高志勇:研究设计;王全意:研究设计、经费支持
| [1] |
沈玲羽, 姜博, 严寒秋, 等. 诺如病毒基因分型研究进展[J]. 中华流行病学杂志, 2020, 41(11): 1927-1932. Shen LY, Jiang B, Yan HQ, et al. Progress on genotyping of norovirus[J]. Chin J Epidemiol, 2020, 41(11): 1927-1932. DOI:10.3760/cma.j.cn112338-20200302-00234 |
| [2] |
Le Guyader FS, Atmar RL, Le Pendu J. Transmission of viruses through shellfish: when specific ligands come into play[J]. Curr Opin Virol, 2012, 2(1): 103-110. DOI:10.1016/j.coviro.2011.10.029 |
| [3] |
严寒秋, 高志勇, 刘白薇, 等. 北京市市售牡蛎诺如病毒污染状况调查[J]. 国际病毒学杂志, 2018, 25(6): 386-388. Yan HQ, Gao ZY, Liu BW, et al. Investigation of norovirus contamination in commercial oysters in Beijing[J]. Int J Virol, 2018, 25(6): 386-388. DOI:10.3760/cma.j.issn.1673-4092.2018.06.007 |
| [4] |
国家卫生和计划生育委员会, 国家食品药品监督管理总局. 食品安全国家标准食品微生物学检验诺如病毒检验(GB 4789.42-2016)[EB/OL]. (2016-12-23)[2021-04-01]. http://down.foodmate.net/standard/sort/3/50379.html. National Health and Family Planning Commission, State Administration for Food and Drug. National standard for food safety examination of food, microbiology, examination of norovirus (GB 4789.42-2016)[EB/OL]. (2016-12-23)[2021-04-01]. http://down.foodmate.net/standard/sort/3/50379.html. |
| [5] |
严寒秋, 霍达, 刘白薇, 等. 牡蛎中诺如病毒检测的前处理方法比较及应用[J]. 国际病毒学杂志, 2015, 22(3): 386-388. Yan HQ, Huo D, Liu BW, et al. Comparison and application of pretreatment methods for norovirus detection in oysters[J]. Int J Virol, 2015, 22(3): 386-388. DOI:10.3760/cma.j.issn.1673-4092.2015.03.003 |
| [6] |
Kojima S, Kageyama T, Fukushi S, et al. Genogroup- specific PCR primers for detection of Norwalk-like viruses[J]. J Virol Methods, 2002, 100(1/2): 107-114. DOI:10.1016/s0166-0934(01)00404-9 |
| [7] |
Kageyama T, Kojima S, Shinohara M, et al. Broadly reactive and highly sensitive assay for Norwalk-like viruses based on real-time quantitative reverse transcription-PCR[J]. J Clin Microbiol, 2003, 41(4): 1548-1557. DOI:10.1128/JCM.41.4.1548-1557.2003 |
| [8] |
詹思延, 叶冬青, 谭红专. 流行病学[M]. 8版. 北京: 人民卫生出版社, 2014: 108-113. Zhang SY, Ye DQ, Tan HZ. Epidemilogy[M]. 8th ed. Beijing: People's Medical Publishing House, 2014: 108-113. |
| [9] |
车荣飞, 白茹, 孙聪, 等. 呼吸道感染多病原检测试剂盒Luminex NxTAGTM RPP与一代测序性能对比[J]. 国际病毒学杂志, 2021, 28(1): 57-61. Che RF, Bai R, Sun C, et al. Comparison of the performance of Luminex NxTAGTM RPP and first-generation sequencing kit for multi-pathogen detection of respiratory infections[J]. Int J Virol, 2021, 28(1): 57-61. DOI:10.3760/cma.j.issn.1673-4092.2021.01.013 |
| [10] |
鲁飞凤, 吕陈昂, 石镇涛. 等. A组轮状病毒原位捕获RT-qPCR检测体系的建立及评估[J]. 国际病毒学杂志, 2021, 28(2): 98-102. Lu FF, Lyu CA, Shi ZT, et al. Development and evaluation of in situ capture RT-qPCR for detection of group A rotavirus[J]. Int J Virol, 2021, 28(2): 98-102. DOI:10.3760/cma.j.issn.1673-4092.2021.02.013 |
| [11] |
Tian Y, Gao ZY, Li WH, et al. Group A rotavirus prevalence and genotypes among adult outpatients with diarrhea in Beijing, China, 2011-2018[J]. J Med Virol, 2021. DOI:10.1002/JMV.27100 |
| [12] |
孙志强, 黄志成, 王修, 等. 诺如病毒检测技术的研究进展[J]. 中国实验诊断学, 2020, 24(10): 1750-1752. Sun ZQ, Huang ZC, Wang X, et al. Research progress of norovirus detection technology[J]. Chin J Lab Diag, 2020, 24(10): 1750-1752. DOI:10.3969/j.issn.1007-4287.2020.10.052 |
| [13] |
高志勇, 严寒秋, 刘白薇, 等. 一种快检试剂对不同基因型诺如病毒的检测评价[J]. 中华实验和临床病毒学杂志, 2015, 29(6): 547-550. Gao ZY, Yan HQ, Liu BW, et al. Evaluation of a rapid test kit for noroviruses of different genotypes[J]. Chin J Exp Clin Virol, 2015, 29(6): 547-550. DOI:10.3760/cma.j.issn.1003-9279.2015.06.020 |
| [14] |
纪蕾, 刘光涛, 沈月华, 等. 2013-2019年浙江省湖州市急性胃肠炎病例GⅡ. P7-GⅡ. 6型诺如病毒基因特征分析[J]. 疾病监测, 2021, 36(1): 53-58. Ji L, Liu GT, Shen YH, et al. Genetic characteristics of norovirus GⅡ. P7-GⅡ. 6 isolated from acute gastroenteritis cases in Huzhou of Zhejiang, 2013-2019[J]. Dis Survell, 2021, 36(1): 53-58. DOI:10.3784/jbjc.202006180218 |
| [15] |
李伟红, 高志勇, 严寒秋, 等. 2016-2019年北京市GⅡ. 2[P16]型诺如病毒全长基因组系统进化分析[J]. 国际病毒学杂志, 2020, 27(2): 97-101. Li WH, Gao ZY, Yan HQ, et al. Phylogenetic analysis of complete genomes of GⅡ. 2[P16] noroviruses in Beijing, 2016-2019[J]. Int J Virol, 2020, 27(2): 97-101. DOI:10.3760/cma.j.issn.1673-4092.2020.02.003 |
| [16] |
蒋翠莲, 曹雪, 艾静, 等. 诺如病毒GⅠ. 6[P11]与GⅡ. 13[P16]共感染的分子特征分析[J]. 国际病毒学杂志, 2020, 27(2): 102-106. Jiang CL, Cao X, Ai J, et al. Analysis on molecular characteristics of co-infection with GⅠ. 6[P11] and GⅡ. 13[P16] noroviruses[J]. Int J Virol, 2020, 27(2): 102-106. DOI:10.3760/cma.j.issn.1673-4092.2020.02.004 |
| [17] |
傅忠燕, 刘莉, 孔翔羽, 等. 诺如病毒GⅨ. 1[GⅡ. P15]型山东株SD20191568全基因组序列特征分析[J]. 中国病原生物学杂志, 2020, 15(12): 1365-1369. Fu ZY, Liu L, Kong XY, et al. The complete genomic sequencings of the Shandong Norovirus strain SD20191568[J]. J Path Biol, 2020, 15(12): 1365-1369. DOI:10.13350/j.cjpb.201201 |
| [18] |
严寒秋, 王永全, 田祎, 等. 北京市市售扇贝中诺如病毒监测分析[J]. 中国食品卫生杂志, 2020, 32(4): 427-431. Yan HQ, Wang YQ, Tian Y, et al. Surveillance of Norovirus contamination on scallops sold in Beijing[J]. Chin J Food Hyg, 2020, 32(4): 427-431. DOI:10.13590/j.cjfh.2020.04.015 |
| [19] |
郑阳, 贾蕾, 刘白薇, 等. 北京市宾馆住宿场所和旅游交通工具诺如病毒污染状况研究[J]. 国际病毒学杂志, 2020, 27(2): 120-123. Zheng Y, Jia L, Liu BW, et al. Investigation on contamination by norovirus in hotels and tourist buses in Beijing[J]. Int J Virol, 2020, 27(2): 120-123. DOI:10.3760/cma.j.issn.1673-4092.2020.02.008 |
 2022, Vol. 43
2022, Vol. 43