文章信息
- 陈璐, 樊俊宁, 孙点剑一, 李立明, 吕筠.
- Chen Lu, Fan Junning, Sun Dianjianyi, Li Liming, Lyu Jun
- 生物学年龄评价方法进展
- Progress in research of measurements of biological age
- 中华流行病学杂志, 2021, 42(9): 1683-1688
- Chinese Journal of Epidemiology, 2021, 42(9): 1683-1688
- http://dx.doi.org/10.3760/cma.j.cn112338-20201210-01396
-
文章历史
收稿日期: 2020-12-10
2. 北京大学公众健康与重大疫情防控战略研究中心 100191
2. Peking University Center for Public Health and Epidemic Preparedness & Response, Beijing 100191, China
衰老(aging)是指在正常环境条件下生理功能随年龄增长而退化,以及对应激的反应能力减弱,是许多慢性非传染性疾病(non-communicable diseases)、功能下降和死亡的主要危险因素之一[1]。虽然个体的实足年龄(chronological age)能够简便地反映衰老,但是具有相同实足年龄者的衰老速率可能不同,且随着年龄增加,个体间的认知功能[2]和健康状况[3]变异性增加。因此,用实足年龄来反映衰老水平存在一定的缺陷;我们需要更好的标志物来衡量个体的衰老进程,即生物学年龄(biological age)[1]。
近年来,发展出多种测量生物学年龄的指标,如基于微观组学的指标、生理指标、功能指标等,以及多种算法或不同层次交汇的复合指标。尽管如此,关于如何评价各种生物学年龄指标的效度和生物学合理性(biological plausibility)仍未达成共识。2018年,Justice等[4]提出,在临床试验中,衰老的生物标志物选择需要满足以下几个标准:①与衰老相关;②测量可靠和可行;③对死亡、健康结局和身体功能具有稳健且一致的预测力;④对干预有响应。本文简要介绍一些常见的生物学年龄评价指标。见表 1。
1. 临床或表型指标:
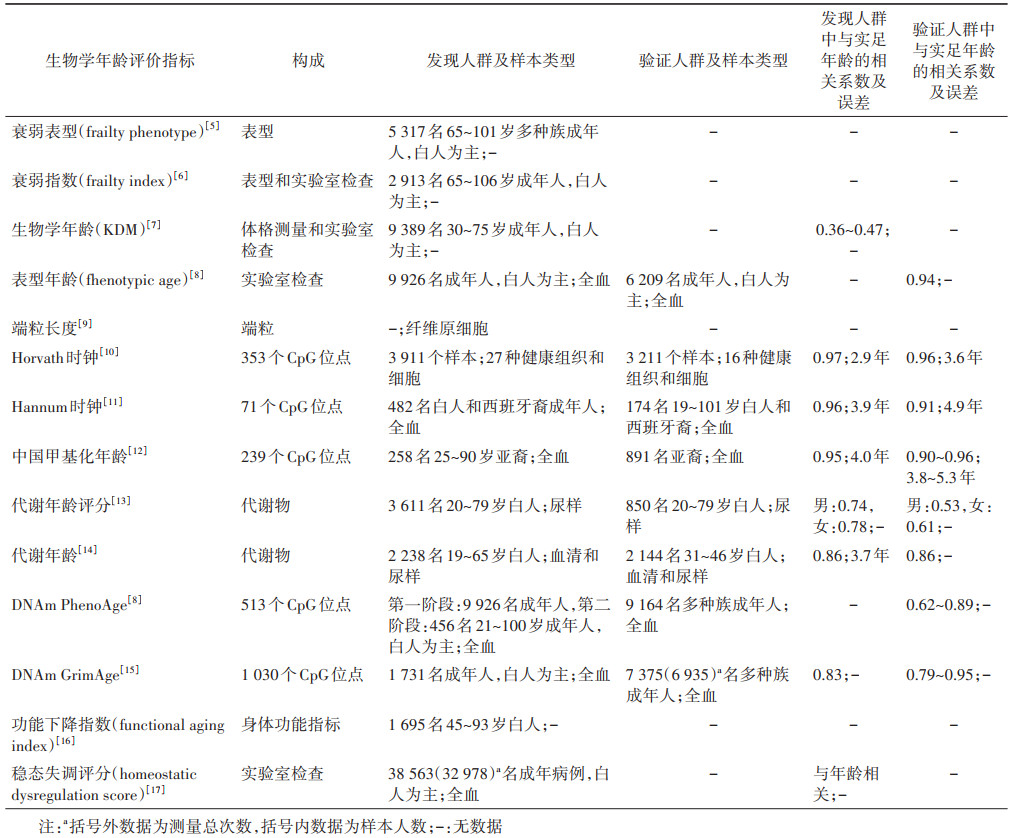
(1)衰弱(frailty)评价指标:衰弱是一种常见的老年综合征,表现为与年龄相关的多器官系统生理储备和功能下降,导致对应激源的易感性增加[18]。任何年龄段的成年人都可能出现衰弱,但在老年人以及慢性病患者中衰弱更为普遍[19]。衰弱导致个体发生不良健康结局的风险增加,进而增加医疗成本与经济负担。近年来涌现出很多衰弱评价工具,其中以衰弱表型(frailty phenotype,FP)和衰弱指数(frailty index,FI)在社区人群中的应用最为广泛。
FP最早由Fried等[5]在2001年提出,在美国心血管健康研究(Cardiovascular Health Study)的5 300多名社区老年人中得到验证。FP纳入的5项表型:手握力低、步行速度缓慢、体力活动水平低、精力不足或自诉疲劳、非主动减重而出现的体重降低。5条标准中,满足3条及以上为衰弱,满足1~2条为衰弱前期,均不满足为不衰弱[5]。近年来,FP的效度在中国老年人群的研究中也得到了验证。例如,在江苏省如皋市长寿与老龄化研究中,1 788名70~84岁老年人中衰弱者和衰弱前期者的全因死亡风险(hazard ratio,HR)分别是不衰弱者的2.24(95%CI:1.31~3.83)倍和1.51(95%CI:1.03~2.21)倍[20]。
Mitnitski等[6]基于加拿大健康与老龄化研究(Canadian Study of Heath and Aging,CSHA)开发了FI。FI为个体累积缺陷占所纳入指标的比例。纳入的缺陷可以是症状、体征、功能障碍和实验室检查异常等,且必须满足5个条件:①必须与健康状况相关,而与年龄相关的性状(如头发花白)不包括在内;②缺陷率随年龄增加而增大;③缺陷不能过早饱和,如老花眼在55岁人群中就已普遍存在,因此不宜纳入;④所纳入的缺陷应该涵盖尽可能多的器官系统;⑤不同研究中FI的构成指标可以不一样,FI的构成指标数量要比构成指标内容更重要,但是纵向比较同一人群FI时,构成变量应保持一致[6, 21]。通常,FI中包含的变量越多,估计值就越稳健,但一般纳入30~40个变量已经足够[21]。早在2011年,Rockwood等[22]利用加拿大全国人口健康调查(National Population Health Survey)的15~102岁社区人群数据验证了在成年人全年龄范围中FI均具有较好的预测效度。中国老年健康影响因素追踪调查(Chinese Longitudinal Healthy Longevity Survey)在≥65岁老年人数据中验证了FI的效度[23]。而中国慢性病前瞻性研究(China Kadoorie Biobank)进一步显示,FI可以有效预测人群的全因和多种死因别死亡风险,是一个较好的生物学年龄代理指标[24]。
相比FP,FI为连续变量,可能对衰弱水平较低的成年人发生不良健康结局的风险有更好的区分度。但近年来的研究发现FP对于预测中年人群的死亡与共病也具有潜在应用价值。Hanlon等[25]基于英国生物银行(UKB)近50万人数据发现,在37~73岁人群中FP测量的衰弱状态与死亡和共病风险存在关联,不过在37~45岁女性中未发现这种关联。FP能否作为成年人全年龄范围的生物学年龄代理指标还需要进一步研究证实。
(2)其他:众多研究揭示了临床指标(如人体测量指标、血液生物标志物等)与衰老、死亡或年龄相关疾病(age-related diseases)的关联。有研究根据临床指标与实足年龄之间的关联,通过一定的算法,例如多重线性回归、主成分分析以及Klemera Doubal法(KDM),开发了新的生物学年龄指标[7, 26];其中,以KDM计算得到的指标对寿命的预测能力最好。这些指标在构建时,各种标志物的相对权重是通过预测实足年龄获得,而非预测发病或死亡风险来获得权重。Levine等[8]基于惩罚Cox回归模型筛选出实足年龄和9个临床血液检测指标,然后通过Gompertz分布的死亡风险的参数比例风险模型构建表型年龄(phenotypic age),与实足年龄相关性达0.94,且对死亡、年龄相关疾病、共病数量和身体功能下降都有良好的预测力。
2. 分子生物学指标:
(1)端粒(telomere)长度:端粒是存在于真核生物染色体末端的DNA重复序列-蛋白质复合物[27]。Harley等[9]率先发现随着年龄的增长,人的成纤维细胞端粒长度不断缩短,开创了细胞衰老的端粒学说。Tsuji等[28]利用人外周血单核细胞测量端粒长度,结果显示其与实足年龄之间的相关系数达-0.832。越来越多的证据表明端粒长度是年龄相关疾病的生物决定因素。例如,超过15年的流行病学和动物模型研究在端粒长度和动脉粥样硬化性心血管疾病之间建立了稳健的联系;尽管二者之间的关联效应值较小,但是全基因组关联研究结果为这种联系提供了较强的因果证据支持[29]。2018年,一项荟萃分析纳入了25个前瞻性研究,包含来自瑞典、丹麦、美国及中国等的121 749例研究对象中21 763例死亡,结果显示端粒每缩短1个标准差,全因死亡HR值为1.09(95%CI:1.06~1.13)[30]。端粒长度与其他健康结局的关联研究,由于样本规模不足及端粒长度的测量偏倚等原因,尚缺乏较为一致的结果[31]。
(2)表观遗传时钟(epigenetic clock):DNA甲基化是指DNA序列上特定的碱基共价结合一个甲基的化学修饰过程;在哺乳动物中,这种DNA甲基化修饰发生在胞嘧啶-磷酸-鸟嘌呤(CpG)二核苷酸中胞嘧啶上的第5位碳原子上[32]。21世纪初,Bjornsson等[33]发现CpG位点甲基化水平与年龄高度相关,基于此,研究人员在包含高通量DNA甲基化数据和实足年龄信息的数据集上使用有监督的机器学习方法(例如弹性网回归)来“学习”人类基因组中哪些CpG位点可以高精度地预测实足年龄,并在验证数据集中估计预测准确性。利用这种方法,目前已经开发出几种基于人体组织DNA中的3~1 030个不等的CpG位点甲基化水平来预测实足年龄的表观遗传时钟,也称为DNA甲基化年龄(DNA methylation age)[15, 34]。其中,以Horvath时钟和Hannum时钟的应用最为广泛。
Horvath时钟利用从各种人类组织和细胞类型的公开数据集获得的DNA甲基化数据开发了由353个CpG位点组成的表观遗传时钟,在与训练样本相同来源数据集的验证样本中误差(median absolute deviation)为3.6年[10]。Hannum等[11]利用西方成年人血液的DNA甲基化数据开发了71个CpG位点的表观遗传时钟,在与训练样本相同来源人群的验证样本中误差(root mean square mean,RMSE)为4.9年。Horvath和Hannum时钟对实足年龄的预测准确性均较高。由于Hannum时钟的开发基于血液样本,对其他组织的预测精度下降[11]。相比之下,Horvath时钟在大多数组织中表现良好[10]。Li等[12]依据Horvath时钟的构建流程开发了中国成年人的239个CpG位点的表观遗传时钟,在验证人群中该指标与实足年龄的相关性≥0.90,RMSE最大为5.34年;然而在同一人群中,Horvath时钟与实足年龄的相关性仅为0.83,RMSE为8~10年,反映了不同人群间甲基化水平的差异。但是这个基于中国人群的表观遗传时钟还未得到其他研究的进一步验证。
目前,基于表观遗传时钟得到的年龄加速指标主要有以下3种[35-36]:①表观遗传年龄加速(epigenetic age acceleration,AA),指机体的表观遗传时钟与实足年龄的偏离,适用于大部分组织和细胞类型。通常有两种计算方式,即表观遗传时钟减去实足年龄或表观遗传时钟对实足年龄回归的残差值,其正负代表加速或减速。AA的计算方式也常用于评价其他生物学年龄指标对实足年龄的偏离;②内在AA(intrinsic epigenetic age acceleration),指的是在调整CD8细胞、CD4细胞、浆细胞、自然杀伤细胞、单核细胞和粒细胞等血细胞计数后,Horvath时钟对实足年龄回归的残差值,避免了血细胞计数差异对表观遗传时钟测量的影响;③外在AA(extrinsic epigenetic age acceleration),为Hannum时钟和3种随年龄变化的血细胞成分的加权平均,能够同时反映血液细胞组成与甲基化内在性质的变化。3种年龄加速指标具有较高相关性。
不断有研究证据表明表观遗传时钟可以代理生物学年龄,即AA与年龄相关疾病或身体状况相关,如衰弱、阿尔茨海默病、癌症和心血管疾病[37-38]。表观遗传时钟对全因死亡风险的预测在各种族均具有良好的表现[39]。然而,也有研究未发现表观遗传时钟与认知和身体功能下降[40]等存在关联。此外,动物干预实验表明延长小鼠寿命和延缓衰老的干预措施如热量限制可以减缓表观遗传衰老[41]。
(3)代谢年龄:代谢物是代谢过程产生的中间产物或终产物,代谢组学提供了基因、RNA、蛋白质和环境因素综合作用下的机体状态定量测量,是机体对病生理刺激或基因修饰的动态反应研究[42]。目前对于衰老过程的代谢变化研究相对较少。尽管有一些研究观察到代谢生物标志物与实足年龄的相关性[43],但基于代谢生物标志物估算代谢年龄的研究较少,且现有研究尚未得到广泛认可。2016年,Hertel等[13]提出代谢标志物需要满足:①与年龄高度相关;②独立于实足年龄,能反映与衰老结局相关的变化;③能够独立于实足年龄预测总寿命。他们在一项20~79岁人群研究中,对人尿液样品进行质子核磁共振光谱分析,利用59种代谢物构建了代谢年龄评分(metabolic age score),发现代谢年龄与各种临床结局独立相关,进一步的生存分析显示,代谢年龄在较低分位数的个体具有更高的生存率[13]。Robinson等[14]在两千余名中老年人中通过弹性网络回归模型构建代谢年龄,发现代谢年龄加速(即代谢年龄对实足年龄回归的残差)与一些早死危险因素(如超重肥胖、糖尿病、重度酒精依赖和抑郁)相关。总的来说,现有研究主要探索机体中代谢物与实足年龄或其他生物学标志物的关联,尽管已有研究构建了代谢年龄,但其外部效度尚未得到充分验证。
3. 复合型指标:近年来,不断有研究针对传统表观遗传时钟进行优化。Horvath团队着手开发新的表观遗传时钟——DNAm PhenoAge和DNAm GrimAge。Levine等[8]利用前文所述的表型年龄,进一步对DNA甲基化水平回归得到DNAm PhenoAge。DNAm PhenoAge时钟分别有41和6个CpG位点与Horvath时钟和Hannum时钟重叠。DNAm PhenoAge与实足年龄中高度相关(r=0.62~0.89),并且在使用各种组织和细胞样本预测疾病和死亡风险方面,胜过了Horvath时钟和Hannum时钟[8]。Lu等[15]通过DNA甲基化水平预测吸烟量(包/年)和血浆蛋白质水平,再基于预测值构建寿命代理指标DNAm GrimAge。DNAm GrimAge在死亡、冠心病和癌症风险等方面的预测表现优于Horvath时钟、Hannum时钟以及DNAm PhenoAge,而且DNAm GrimAge及其中包含的生物标志物预测值都表现出与生活方式危险因素的关联[15]。
4. 其他生物学年龄评价指标:除了上述提及的生物学年龄评价指标外,还有许多研究探索利用其他类型指标构建生物学年龄,如利用视听功能、肺功能、肌肉强度和运动能力构建的功能下降指数(functional aging index)[16],利用37种生物标志物代表的6个生理系统偏离正常值的距离计算稳态失调评分(homeostatic dysregulation score)[17],不一而足。这些指标在对寿命、住院或机体功能预测方面可能具备一定优势,但是与端粒长度、表观遗传时钟以及FI等研究较为成熟的指标相比,还缺少外部人群的验证,研究成果能否外推到其他人群,有无实际应用价值,还需要进一步探究。另外,衰老不仅与恶性肿瘤、自身免疫病等慢性非传染性疾病的发生相关,也导致感染等其他衰老相关性疾病的发生风险增加,这一过程伴随着免疫系统功能的进行性退化。多项研究发现免疫细胞和免疫分子与衰老相关。如何利用单个或复合免疫指标作为生物学年龄评价指标尚待流行病学研究的进一步检验[8]。
5. 各种生物学年龄评价指标的比较:上面提到的这些指标的构建均基于衰老的生物学机理,包含了多细胞生物衰老过程中从基因到细胞、组织、器官、个体的各个层次。利用询问、非侵入性临床检查、或临床实践中常规检测的一些生化指标构建临床或表型指标,可行性最好,成本也相对较低。而分子生物学指标的检测技术和成本一定程度地限制了其在大样本人群中的应用。
关于这些指标对死亡、健康结局和身体功能等方面的预测能力,在一些研究中,调整实足年龄后,端粒长度和表观遗传时钟在解释衰老结局方面的表现一般[39, 44],而临床或表型指标往往对衰老结局具有更好的预测力[8, 45]。前文所述的DNAm PhenoAge在传统表观遗传时钟基础上结合临床信息,在对全因死亡、癌症、健康寿命、机体功能和阿尔茨海默症的预测方面较Horvath时钟、Hannum时钟具有更好的表现[8]。分子生物学指标与临床或表型指标之间预测能力的差异可能提示,前者仅捕获了复杂衰老过程中的少数环节,而后者则反映了发生在机体各个系统的综合变化[46],但其中暗含的生物机制尚不明确。
现有大多数研究都是探索单个或单类生物学年龄评价指标对不同人群、不同结局的预测效度,或者比较少数几个指标的关联或预测效度,或者通过与发展较成熟的指标比较,验证新构建指标的效度。而在同一人群中同时研究多种生物学年龄指标的表现可以更好地比较这些指标的优劣,并从人群层面剖析不同层次的生物学标志物在衰老通路上可能发挥的作用。目前这类研究仍非常有限。
2018年Hastings等[45]利用新西兰1972-1973年出生的964名中年人从出生到38周岁有重复测量的生理指标和基因组数据,量化了11种生物学年龄指标,包括端粒长度、表观遗传时钟、KDM生物学年龄与稳态失调得分及各指标相应的变化速率。该研究发现不同层次指标间相关性较低,Hannum时钟以及临床或表型指标与衰老结局有统计学关联,但效应值较微弱,而大多数其他指标没有表现出统计学关联。该研究的研究对象相对年轻,而衰老特征可能需要达到一定年龄才有明显改变,且有的指标可能刻画的是生命后期的衰老进程,使得该研究的效应值低于既往研究。2019年,Hastings等[47]利用美国国家健康和营养调查(National Health and Nutrition Examination Surveys,NHANES)六千余名20~84岁研究对象的数据比较了4种生物学年龄指标,发现端粒长度代理生物学年龄的表现较KDM生物学年龄、表型年龄与稳态失调得分更差一些。遗憾的是,该研究没有纳入衰老研究中最重要的指标之一——表观遗传时钟;且研究基于横断面调查数据,也无法排除队列效应和生命早期暴露对生物标志物的影响。Li等[48]利用845名瑞典中老年双生子20年间共3 973人次的重复测量数据,比较了9种生物学年龄指标,包括端粒长度、2种表观遗传时钟、4种临床或表型指标及2种复合型指标。结果发现大多数指标随实足年龄基本呈线性变化;在调整实足年龄后,各指标间的相关性微弱甚至趋近于无。在控制实足年龄后,除端粒长度以外的指标都与死亡风险相关;当同时纳入所有生物学年龄指标后,Horvath时钟、DNAm GrimAge以及FI的效应值依然有统计学意义,反映了这3个指标至少部分地测量了衰老过程的不同方面[48]。但是该研究在同一模型比较不同指标对死亡的预测效果时,仅纳入了具有全部测量指标的288名研究对象,可能存在选择偏倚;且研究对象出生年份跨度大,队列效应导致的生物学年龄和死亡风险的变化不可忽视。
6. 小结:目前就各种生物学年龄评价指标对疾病、死亡以及机体功能下降的预测效度优劣还缺乏一致的结论。概观现有的指标,衰弱评价指标具有相对较好的表现,但其更多应用于中老年人群中,而其他表型或临床指标以及表观遗传时钟的表现不一致,复合型指标在对疾病、死亡以及机体功能下降等健康结局的预测效度方面显示出潜在应用价值,端粒长度的预测能力可能较弱,而其他评价指标的证据有限。在生物学年龄评价指标与实足年龄的关联强度方面,表观遗传时钟对实足年龄的预测精度较高,端粒长度与实足年龄的关联较弱,而其他指标与实足年龄的关联还缺少稳定的结论。
未来研究可加强对多类生物学年龄评价指标的综合分析,以便了解不同层次的生物学标志物在衰老通路上可能发挥的作用和相对效应大小,确定生物学衰老的可靠、精确的代理指标,以便更好地开展衰老相关的科学研究。由于临床与代谢指标在男、女性中存在较大差异,我们可以分性别构建或评价相应生物学年龄指标以改进现有方法的不足。此外,由于不同种族之间存在明显的遗传异质性,我们可以借鉴在西方人群中的生物学年龄指标构建过程,构建更适合我国人群的指标。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Khan SS, Singer BD, Vaughan DE. Molecular and physiological manifestations and measurement of aging in humans[J]. Aging Cell, 2017, 16(4): 624-633. DOI:10.1111/acel.12601 |
| [2] |
Bethlehem RAI, Paquola C, Seidlitz J, et al. Dispersion of functional gradients across the adult lifespan[J]. NeuroImage, 2020, 222: 117299. DOI:10.1016/j.neuroimage.2020.117299 |
| [3] |
Harris JR, Pedersen NL, McClearn GE, et al. Age differences in genetic and environmental influences for health from the Swedish adoption/Twin study of aging[J]. J Gerontol, 1992, 47(3): 213-220. DOI:10.1093/geronj/47.3.p213 |
| [4] |
Justice JN, Ferrucci L, Newman AB, et al. A framework for selection of blood-based biomarkers for geroscience-guided clinical trials: report from the TAME biomarkers workgroup[J]. Geroscience, 2018, 40(5-6): 419-436. DOI:10.1007/s11357-018-0042-y |
| [5] |
Fried LP, Tangen CM, Walston J, et al. Frailty in older adults: evidence for a phenotype[J]. J Gerontol, 2001, 56(3): M146-157. DOI:10.1093/gerona/56.3.m146 |
| [6] |
Mitnitski AB, Mogilner AJ, Rockwood K. Accumulation of deficits as a proxy measure of aging[J]. Scientific World J, 2001, 1: 321027. DOI:10.1100/tsw.2001.58 |
| [7] |
Levine ME. Modeling the rate of senescence: can estimated biological age predict mortality more accurately than chronological age?[J]. J Gerontol Ser A, 2013, 68(6): 667-674. DOI:10.1093/gerona/gls233 |
| [8] |
Levine ME, Lu AT, Quach A, et al. An epigenetic biomarker of aging for lifespan and healthspan[J]. Aging, 2018, 10(4): 573-591. DOI:10.18632/aging.101414 |
| [9] |
Harley CB, Futcher AB, Greider CW. Telomeres shorten during ageing of human fibroblasts[J]. Nature, 1990, 345(6274): 458-460. DOI:10.1038/345458a0 |
| [10] |
Horvath S. DNA methylation age of human tissues and cell types[J]. Genome Biol, 2013, 14(10): R115. DOI:10.1186/gb-2013-14-10-r115 |
| [11] |
Hannum G, Guinney J, Zhao L, et al. Genome-wide methylation profiles reveal quantitative views of human aging rates[J]. Mol Cell, 2013, 49(2): 359-367. DOI:10.1016/j.molcel.2012.10.016 |
| [12] |
Li J, Zhu XY, Yu K, et al. Exposure to polycyclic aromatic hydrocarbons and accelerated DNA methylation aging[J]. Environ Health Perspect, 2018, 126(6): 067005. DOI:10.1289/EHP2773 |
| [13] |
Hertel J, Friedrich N, Wittfeld K, et al. Measuring biological age via metabonomics: the metabolic age score[J]. J Proteome Res, 2016, 15(2): 400-410. DOI:10.1021/acs.jproteome.5b00561 |
| [14] |
Robinson O, Hyam MC, Karaman I, et al. Determinants of accelerated metabolomic and epigenetic aging in a UK cohort[J]. Aging Cell, 2020, 19(6): e13149. DOI:10.1111/acel.13149 |
| [15] |
Lu AT, Quach A, Wilson JG, et al. DNA methylation GrimAge strongly predicts lifespan and healthspan[J]. Aging, 2019, 11(2): 303-327. DOI:10.18632/aging.101684 |
| [16] |
Finkel D, Sternäng O, Jylhävä J, et al. Functional aging index complements frailty in prediction of entry into care and mortality[J]. J Gerontol Ser A, 2019, 74(12): 1980-1986. DOI:10.1093/gerona/glz155 |
| [17] |
Li Q, Wang SR, Milot E, et al. Homeostatic dysregulation proceeds in parallel in multiple physiological systems[J]. Aging Cell, 2015, 14(6): 1103-1112. DOI:10.1111/acel.12402 |
| [18] |
Fried LP, Hadley EC, Walston JD, et al. From bedside to bench: research agenda for frailty[J]. Sci Aging Knowledge Environ, 2005, 2005(31): pe24. DOI:10.1126/sageke.2005.31.pe24 |
| [19] |
Hoogendijk EO, Afilalo J, Ensrud KE, et al. Frailty: implications for clinical practice and public health[J]. Lancet, 2019, 394(10206): 1365-1375. DOI:10.1016/S0140-6736(19)31786-6 |
| [20] |
Shi GP, Ma T, Zhu YS, et al. Frailty phenotype, frailty index and risk of mortality in Chinese elderly population-Rugao longevity and ageing study[J]. Arch Gerontol Geriatr, 2019, 80: 115-119. DOI:10.1016/j.archger.2018.11.001 |
| [21] |
Searle SD, Mitnitski A, Gahbauer EA, et al. A standard procedure for creating a frailty index[J]. BMC Geriatr, 2008, 8: 24. DOI:10.1186/1471-2318-8-24 |
| [22] |
Rockwood K, Song XW, Mitnitski A. Changes in relative fitness and frailty across the adult lifespan: evidence from the Canadian National Population Health Survey[J]. CMAJ, 2011, 183(8): E487-494. DOI:10.1503/cmaj.101271 |
| [23] |
Yang F, Gu DN. Predictability of frailty index and its components on mortality in older adults in China[J]. BMC Geriatr, 2016, 16: 145. DOI:10.1186/s12877-016-0317-z |
| [24] |
Fan JN, Yu CQ, Guo Y, et al. Frailty index and all-cause and cause-specific mortality in Chinese adults: a prospective cohort study[J]. Lancet Public Health, 2020, 5(12): e650-660. DOI:10.1016/S2468-2667(20)30113-4 |
| [25] |
Hanlon P, Nicholl BI, Jani BD, et al. Frailty and pre-frailty in middle-aged and older adults and its association with multimorbidity and mortality: a prospective analysis of 493 737 UK Biobank participants[J]. Lancet Public Health, 2018, 3(7): e323-332. DOI:10.1016/S2468-2667(18)30091-4 |
| [26] |
Klemera P, Doubal S. A new approach to the concept and computation of biological age[J]. Mech Ageing Dev, 2006, 127(3): 240-248. DOI:10.1016/j.mad.2005.10.004 |
| [27] |
Blackburn EH, Szostak JW. The molecular structure of centromeres and telomeres[J]. Ann Rev Biochem, 1984, 53(1): 163-194. DOI:10.1146/annurev.bi.53.070184.001115 |
| [28] |
Tsuji A, Ishiko A, Takasaki T, et al. Estimating age of humans based on telomere shortening[J]. Forens Sci Int, 2002, 126(3): 197-199. DOI:10.1016/s0379-0738(02)00086-5 |
| [29] |
de Meyer T, Nawrot T, Bekaert S, et al. Telomere length as cardiovascular aging biomarker: JACC review topic of the week[J]. J Am Coll Cardiol, 2018, 72(7): 805-813. DOI:10.1016/j.jacc.2018.06.014 |
| [30] |
Wang Q, Zhan YQ, Pedersen NL, et al. Telomere length and all-cause mortality: a Meta-analysis[J]. Ageing Res Rev, 2018, 48: 11-20. DOI:10.1016/j.arr.2018.09.002 |
| [31] |
Jylhävä J, Pedersen NL, Hägg S. Biological age predictors[J]. EBioMedicine, 2017, 21: 29-36. DOI:10.1016/j.ebiom.2017.03.046 |
| [32] |
Attwood JT, Yung RL, Richardson BC. DNA methylation and the regulation of gene transcription[J]. Cell Mol Life Sci, 2002, 59(2): 241-257. DOI:10.1007/s00018-002-8420-z |
| [33] |
Bjornsson HT, Sigurdsson MI, Fallin MD, et al. Intra-individual change over time in DNA methylation with familial clustering[J]. JAMA, 2008, 299(24): 2877-2883. DOI:10.1001/jama.299.24.2877 |
| [34] |
Weidner CI, Lin Q, Koch CM, et al. Aging of blood can be tracked by DNA methylation changes at just three CpG sites[J]. Genome Biol, 2014, 15(2): R24. DOI:10.1186/gb-2014-15-2-r24 |
| [35] |
Dhingra R, Nwanaji-Enwerem JC, Samet M, et al. DNA methylation age-environmental influences, health impacts, and its role in environmental epidemiology[J]. Curr Environ Health Rep, 2018, 5(3): 317-327. DOI:10.1007/s40572-018-0203-2 |
| [36] |
Horvath S, Ritz BR. Increased epigenetic age and granulocyte counts in the blood of Parkinson's disease patients[J]. Aging, 2015, 7(12): 1130-1142. DOI:10.18632/aging.100859 |
| [37] |
Breitling LP, Saum KU, Perna L, et al. Frailty is associated with the epigenetic clock but not with telomere length in a German cohort[J]. Clin Epigenetics, 2016, 8: 21. DOI:10.1186/s13148-016-0186-5 |
| [38] |
Horvath S, Raj K. DNA methylation-based biomarkers and the epigenetic clock theory of ageing[J]. Nat Rev Genet, 2018, 19(6): 371-384. DOI:10.1038/s41576-018-0004-3 |
| [39] |
Chen BH, Marioni RE, Colicino E, et al. DNA methylation-based measures of biological age: Meta-analysis predicting time to death[J]. Aging, 2016, 8(9): 1844-1865. DOI:10.18632/aging.101020 |
| [40] |
Marioni RE, Shah S, McRae AF, et al. The epigenetic clock is correlated with physical and cognitive fitness in the Lothian Birth Cohort 1936[J]. Int J Epidemiol, 2015, 44(4): 1388-1396. DOI:10.1093/ije/dyu277 |
| [41] |
Wang TN, Tsui B, Kreisberg JF, et al. Epigenetic aging signatures in mice livers are slowed by dwarfism, calorie restriction and rapamycin treatment[J]. Genome Biol, 2017, 18(1): 57. DOI:10.1186/s13059-017-1186-2 |
| [42] |
Griffin JL, Atherton H, Shockcor J, et al. Metabolomics as a tool for cardiac research[J]. Nat Rev Cardiol, 2011, 8(11): 630-643. DOI:10.1038/nrcardio.2011.138 |
| [43] |
Yu ZH, Zhai GJ, Singmann P, et al. Human serum metabolic profiles are age dependent[J]. Aging Cell, 2012, 11(6): 960-967. DOI:10.1111/j.1474-9726.2012.00865.x |
| [44] |
Horvath S, Gurven M, Levine ME, et al. An epigenetic clock analysis of race/ethnicity, sex, and coronary heart disease[J]. Genome Biol, 2016, 17(1): 171. DOI:10.1186/s13059-016-1030-0 |
| [45] |
Belsky DW, Moffitt TE, Cohen AA, et al. Eleven telomere, epigenetic clock, and biomarker-composite quantifications of biological aging: do they measure the same thing?[J]. Am J Epidemiol, 2018, 187(6): 1220-1230. DOI:10.1093/aje/kwx346 |
| [46] |
Liu ZY, Kuo PL, Horvath S, et al. A new aging measure captures morbidity and mortality risk across diverse subpopulations from NHANES Ⅳ: a cohort study[J]. PLoS Med, 2018, 15(12): e1002718. DOI:10.1371/journal.pmed.1002718 |
| [47] |
Hastings WJ, Shalev I, Belsky DW. Comparability of biological aging measures in the national health and nutrition examination study, 1999-2002[J]. Psychoneuroendocrinology, 2019, 106: 171-178. DOI:10.1016/j.psyneuen.2019.03.012 |
| [48] |
Li X, Ploner A, Wang YZ, et al. Longitudinal trajectories, correlations and mortality associations of nine biological ages across 20-years follow-up[J]. eLife, 2020, 9: e51507. DOI:10.7554/eLife.51507 |
 2021, Vol. 42
2021, Vol. 42