文章信息
- 孙一鑫, 王淼, 杨明芳, 詹思延.
- Sun Yixin, Wang Miao, Yang Mingfang, Zhan Siyan
- 树状扫描统计量用于药品和疫苗安全性监测的综述
- Review on tree-based scan statistic in drug and vaccine safety monitoring
- 中华流行病学杂志, 2021, 42(7): 1286-1291
- Chinese Journal of Epidemiology, 2021, 42(7): 1286-1291
- http://dx.doi.org/10.3760/cma.j.cn112338-20201103-01297
-
文章历史
收稿日期: 2020-11-03
2. 首都医科大学附属北京安贞医院/北京市心肺血管疾病研究所流行病研究室 100029
2. Department of Epidemiology, Beijing Anzhen Hospital, Capital Medical University, Beijing Institute of Heart, Lung and Blood Vessel Diseases, Beijing 100029, China
药品上市后安全性监测是评价用药安全、保障公众健康的重要手段。传统监测方法是基于自发报告系统的被动监测(passive surveillance),可以在一定程度上发现药品不良反应(adverse drug reactions,ADR),是目前全球各国普遍采用的一类监测模式[1]。然而,被动监测往往缺乏时效性,存在漏报、重复报告、选择性报告等局限,造成ADR信号发现延迟、甚至无法检出[2]。随着信息时代的到来,覆盖大样本人群的电子医疗数据库不断被开发利用,多种数据挖掘方法也被引入到药品上市后安全性监测中,形成了主动监测(active surveillance)模式。该模式能够主动收集并快速发现ADR信号,弥补了被动监测的缺陷,极大提高了监测的效果与效率[3]。
树状扫描统计量(Tree-based scan statistic,TreeScan)于2003年由Kulldorff等[4]提出,最初被用于职业病死因监测。随后在2010年用于ADR信号监测[5]。与传统药物流行病学研究中对特定的药物-事件对进行假设检验相比,该方法的最大优势在于无需提前设定假设就能够同时对大量药物-事件对进行挖掘,而且能够克服传统多重比较中常出现的过于保守或过于激进估算这一不足,从而为监测药品安全性提供切实可靠的信号依据[5-6]。
本研究采用系统综述的方法对国内外使用TreeScan方法开展的药物流行病学研究进行系统梳理,总结TreeScan方法的发展与应用现状,阐述其方法学原理,为TreeScan方法在我国药品、疫苗安全性监测和其他相关研究领域的应用提供参考。
1. 基于TreeScan方法开展的药物流行病学现状:研究使用TreeScan等相关英文关键词,系统检索英文文献数据库(Medline、Embase与Web of Science),纳入2020年5月16日之前发表的使用TreeScan进行药品及疫苗上市后安全性监测的原始文献,并按照系统综述PRISMA报告规范的要求对文献进行筛选与信息提取[7]。对上述使用TreeScan方法开展的药物流行病学的基本特征、方法学等方面进行汇总描述,总结TreeScan方法的发展与应用现状,阐述其方法学原理。
在目前已经公布研究结果的研究中,探讨药物安全性评价的研究有9项[5, 8-15],探讨疫苗安全性的研究有6项[16-21]。其中美国开展的研究有11项(73.3%)[5, 8-11, 14-18, 21],在中国开展的研究有3项(20.0%)[12-13, 20],在韩国开展的研究有1项(6.7%)[19]。纳入的15项研究中,有7项研究以会议摘要的形式在国际药物流行病学及药物治疗风险管理会议(International Conference on Pharmacoepidemiology & Therapeutic Risk Management)上进行了报告[11-15, 20-21]。纳入文献的基本特征见表 1。研究所用数据大多来自累积4~13年的电子病历及保险/索赔数据库,样本量在3 600~7 500 000人之间。涉及的TreeScan方法包括3类模型:泊松概率模型(Poisson probability model)、伯努利概率模型(Bernoulli probability model)及树状时空扫描统计量(Tree-temporal scan statistic)。另外,8项研究在设计上采用了自身对照设计(药物3篇、疫苗5篇),2项研究选用其他药物或疫苗作为对照(药物1篇、疫苗1篇),5项药物研究由于是会议摘要而无法判断其对照类型。
2. TreeScan方法原理:TreeScan是一种数据挖掘的方法,目前已经在与职业病相关的暴露因素研究、疫苗或药物不良反应信号挖掘等方面开展了一些应用。该方法可以探讨多种因素或暴露与多个研究结局的关联,通过计算对数似然比获得统计值,并采用蒙特卡洛概率分析的方法来进行统计推断(获得P值)[22]。可以对数千甚至上百万组数据同时进行多重比较,而且能克服传统多重比较中常出现的过于保守或过于激进的估算这一不足[23]。
TreeScan是树状结构和扫描统计量两种方法的结合运用。树状结构是一种离散结构,表示单个元素或节点之间的关系。树状结构中的基本单位常被称为节点(node),树状扫描统计量中的树状结构可设计成从左向右辐射的排列结构(图 1)。在树状结构中节点处于不同位置时的名称不同。处于左侧的节点是与之关联的位于右侧的节点的亲代节点(Parent),而与之对应的,处于右侧的节点是与之关联的左侧节点的子代节点(Child)。处于树状结构中最左侧的节点被称为根(Root),处于树状结构的最右侧的节点被称为叶(Leaf)。连接在同一个亲代节点上的两个或两个以上节点,彼此间互为姊妹节点(Siblings)。节点之间的连线被称之为分支(Branch或Edge)[24]。
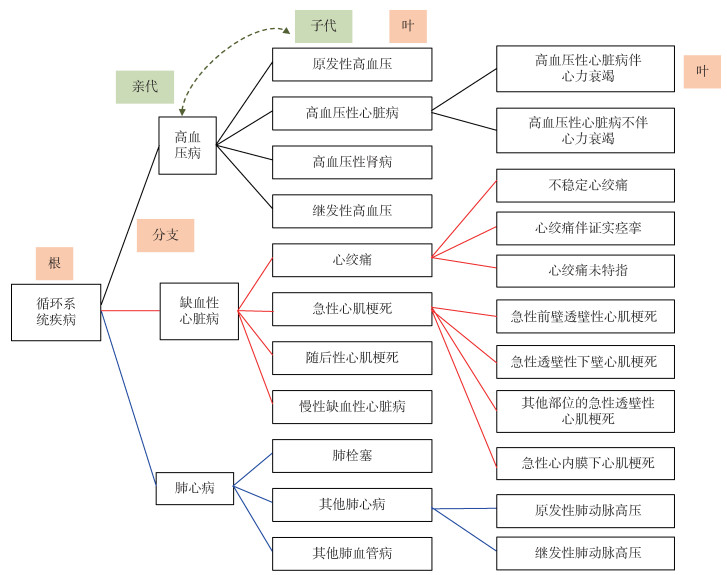
|
| 图 1 树状结构示意图(以循环系统疾病为例) |
扫描统计量用于检测时空数据中的异常簇(Cluster)。在使用时首先通过特定的位置集合和时间范围对簇进行限定。然后通过计算各个簇的观察数(如发病或死亡人数)和期望数来获得统计量,进而可以发现具有最大统计量的簇。扫描是指在不同的空间位置用不同大小的窗口去扫描。统计是指统计窗口内的对象分布数目和窗口外分布的差异,并对其随机性进行假设检验。已有研究对该方法的原理进行了生动的阐述:使用硬币这个简单的道具,先真实抛掷硬币并记录一个正反面排列顺序,再随意编造一个排列顺序,通过统计连续多次出现硬币正面朝上这个簇的分布,来推断两个排列顺序的真伪,这是对扫描统计量的原理的一次简单却经典的展示[25]。
3. TreeScan方法分类及应用:美国哈佛医学院和哈佛朝圣者医疗保健院的Martin Kulldorff教授为推动树状扫描统计量的推广应用做出了卓越的贡献。他将树状扫描统计量开发成便于使用的软件并免费使用。软件的下载地址为:https://www.treescan.org。该软件中涉及树状扫描统计量的分析方法有3种,可以根据研究目标和获取的数据特点进行选择(表 2)。上述3种模型在选择上主要需要考虑以下两点:拟开展研究在研究设计上是否需要预先设置对照;是否关注暴露出现与发生不良结局之间的风险期长度[24]。
第一种是泊松概率模型,该模型可以用于分析多种暴露或干预与不良结局的关联。暴露或干预可以是不同的职业、药物或疫苗。不良事件结局可以是死亡、疾病的发病或不良反应。分析时会对树状结构进行剪枝(在每个节点间进行剪切),分别计算被剪下来的枝上包含的节点,及其余树状结构中节点上发生研究结局的理论频数,进而根据对应的算法进行统计量计算,结合蒙特卡洛概率分析进行统计推断。基于韩国ADR自发报告数据库开展卡介苗的安全性信号监测研究采用的是该模型[19]。研究以数据库中所有疫苗接种者作为研究对象,以接种卡介苗人群出现的23种可能与疫苗接种相关的不良反应构建树状结构。按照模型分析要求准备数据导入文件(出现的特定不良反应、出现特定不良反应者接种卡介苗的人数和总人数),分析中数据导入文件与树状结构自动建立映射完成分析。在分析中模型会在每个树状结构的分支进行剪切,将剪切下来的分支(特定类型不良反应如发热)与剩余的不良反应(剩余的22种不良反应数据合集)相互形成对照,通过分析上述2个分组中卡介苗接种占各自分组中的比例差异进行统计推断。模型会输出树状结构中的每个分支(特定不良反应类型)的统计分析结果。采用该类型模型开展研究无需预先设立对照组,另外,由于上述模型未考虑暴露与发生不良结局的风险期间隔,可能存在一定的偏倚。
第二种为伯努利概率模型,该模型也可以分析暴露与多种不良事件结局的关联。此方法在构建树状结构上与泊松概率模型一致,但需要在研究设计时设置对照组。因此,结局数据导入文件与泊松概率模型不同(出现的特定不良反应、暴露组发生人数和对照组发生人数)。在设定对照时,对照可以是接受其他干预措施的人群,也可以是自身前后对照。在自身前后对照设计中需要规定暴露和对照的时长,如接种疫苗后的第一个28 d作为暴露期,可以将第二个28 d作为对照期。暴露的时长设置应根据研究的药物或疫苗出现不良反应的特点进行设定。设置对照人数时可以按1∶1设置,也可以按1∶n匹配。另外,在该研究设计中需要设定一个在接受干预或暴露之前的“洗脱”期,用于排除在接受干预或暴露前的特定时间内出现过研究终点事件的研究对象,以排除暴露或干预后出现的终点事件属于“旧病复发”的可能。Schachterle等[10]通过整合美国医疗保险数据、药品处方记录和就诊记录,采用该方法分析了抗真菌药物使用后可能出现的药物不良反应。上述研究以研究人群中出现的879个诊断构建树状结构,分析其中可能与抗真菌药物使用有关的不良反应类型,采用用药前后自身对照设计,同时,设置了一个180 d的洗脱期并设定了2个暴露时长(14 d和56 d)。
第三种为树状时空扫描统计,该方法的优势:不仅可以探讨暴露与多种不良事件结局的关联,还可以通过分析暴露后不同风险期时长中不良反应事件出现的概率大小,获得暴露或干预后某种特定不良反应集中出现的时点。时空中的“时”是暴露(如疫苗接种)后出现不良反应的时长,“空”是指树状结构中不同的分支。该方法在设计时需要设置对照(常用自身对照设计),且需要设定暴露的时长。分析所需的树状结构的构建与之前的2种方法相同,而结局数据导入文件不同(出现的特定不良反应、出现特定不良反应的人数和对应人数的时长),多用于疫苗的主动监测。2018年Yih等[17]基于此方法,利用美国食品药品监督管理局哨点数据合作伙伴系统中1 903 697名接种四价乳头状瘤病毒疫苗的人群数据,对该疫苗可能存在的不良反应信号进行了分析。在发现新的不良反应信号的同时,也验证了既往研究已经证实的该疫苗存在的不良反应信号——蜂窝织炎,发现该不良反应主要出现在接种后的第2~3天。
4. 探究TreeScan方法信号检出能力的研究进展:在目前的研究中,有两种方法用于探究TreeScan方法的信号检出能力与准确性。一种方法是同时使用其他不良反应信号挖掘方法进行分析,比较TreeScan方法与其他方法的检出结果,另一种方法为使用已知存在的不良反应,探究该方法能否检出这个不良反应信号。
Brown等[8]首次采用自身前后对照设计,运用TreeScan对2种抗真菌药物(特比萘芬和伊曲康唑)和2种降糖药物(吡格列酮和罗格列酮)的ADR信号进行了探讨。在该研究中,研究者通过使用健康维护组织网络(Health Maintenance Organization Research Network)中涵盖的340万人的门诊、急诊及住院医疗信息中提取的药物使用和患病记录,分析并发现了5个可能与服用特比萘芬相关的不良反应,2个可能与服用伊曲康唑相关的不良反应,其中有3个是上述药物已知的不良反应(肝脏疾病、过敏、恶心呕吐)。该研究还比较了ADR自发报告研究常用的伽玛泊松分布缩减法和TreeScan在检出ADR方面的一致性,结果显示TreeScan检出的ADR更多,2种方法检出ADR的一致率为67%。同时,该研究也提示研究存在适应症偏倚和通道偏倚,如:研究发现抗真菌药物使用与出现皮肤疾病诊断存在关联,降糖药物使用与出现肾脏疾病诊断存在关联(伴有肾脏疾病的糖尿病患者会因为使用其他药物存在禁忌,进而服用研究药物)。近年来我国的研究者也开始对TreeScan在药物流行病学研究中的应用进行了探索[12-13, 20]。研究者利用我国区域医疗信息平台,对TreeScan、伽玛泊松分布缩减法、贝叶斯置信传播神经网络、传统队列研究等4种方法检出他汀类药物已知ADR的能力进行了评价,结果显示TreeScan检出准确性高于其他3种方法,受试者工作特征曲线下面积为0.75(95%CI:0.62~0.89)[13]。
TreeScan方法在使用中的注意事项:本研究通过系统梳理既往研究,总结出TreeScan应用于药物或疫苗ADR研究时,在研究设计和数据处理时需要重点关注的注意事项:①需要收集并展示可以反映研究对象代表性的相关数据(如性别、年龄、种族和其他人口统计学信息),且描述研究数据来源的特征,注意暴露因素数据与研究结局数据之间链接的关键信息完整性;②如果使用疾病作为树状结构,从而探讨药物使用或疫苗接种相关ADR,需要对树状结构进行定义。应排除药物使用或疫苗接种不可能导致的疾病或状态,且在树状结构中排除拟研究药物的适应症或与用药目的相关的疾病;③在收集研究对象患病相关数据时,应尽量采集全面。可从住院、门诊和急诊就诊信息等多个渠道进行资料采集和汇总,以降低漏诊的可能性。在目前正式发表的研究中,只有2项研究在收集研究对象患病信息时涵盖了上述3个诊断信息源[5, 18];④研究中需要考虑以开始用药或接种为时间点向前设定一个“洗脱”期对研究对象进行筛选。对于洗脱期内已经出现了研究者所关注安全信号的研究对象,应在最终分析时进行剔除,因为上述时期内的病症与用药或接种无关。洗脱期的长短需要根据研究关注的不良事件的病程特点进行设置。同时,研究对象限定在首次接受暴露的人群范围为最佳[9, 17-18]。另外,建议在研究中关注与药物或疫苗使用有关的已知ADR,进而在研究中对该方法的检验效能进行验证。ADR的发生常与药物使用者或疫苗接种者的性别、年龄、种族或基础疾病等因素存在关联。目前已经正式发表的使用TreeScan监测药物或疫苗ADR的研究中,往往对研究人群的特征或代表性重视不足。有的研究仅介绍了研究对象的年龄特点[16-18, 21],另有4项研究仅介绍了数据来源,未对研究对象的特征进行描述[8-10, 19]。在未来的研究中应该加以重视。
TreeScan在分析中虽然可以通过间接标准化的方法对期望研究结局发生数量进行调整(条件分析),进而在一定程度上实现控制混杂偏倚的目的。但受到调整因素数量的限制,混杂控制可能不够充分[4]。如果研究设计中选取其他人群作为对照(接受其他类型干预的人群如使用其他药物的人群),则可以采用倾向性评分匹配的方法为研究组匹配可比的对照人群,进而能在研究中更好地控制偏倚。目前已经有研究者进行了评价,结果显示倾向性评分匹配后可以有效地降低一类错误(假阳性)的发生概率[9]。
TreeScan在应用中还有另外一个需要特别关注的问题,在目前探讨药物使用或疫苗接种相关ADR的研究中,常将研究对象在服药或接种疫苗后第一次出现的疾病或异常状况作为研究结局,而排除后续出现的病症[5,8-10,16-18]。这样可能会导致真实的不良反应信号信息被排除。因此,研究对象暴露期内的其他病症与药物或疫苗使用的关联应给予重视。
5. 小结:本研究通过系统综述TreeScan在药物流行病学研究领域中的应用,归纳了该方法在运用中的注意事项,总结了该方法的局限性以及改进情况的进展。旨在让更多的研究者了解TreeScan的正确使用方法,推动该方法的推广应用和不断完善。限于综述的目的和篇幅,本文没有对TreeScan方法原理和软件操作方法进行更细致的介绍,感兴趣的研究者可以从TreeScan网站(https://www.treescan.org)获得更多信息和资料。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Huang YL, Moon J, Segal JB. A comparison of active adverse event surveillance systems worldwide[J]. Drug Saf, 2014, 37(8): 581-596. DOI:10.1007/s40264-014-0194-3 |
| [2] |
Pariente A, Gregoire F, Fourrier-Reglat A, et al. Impact of safety alerts on measures of disproportionality in spontaneous reporting databases: the notoriety bias[J]. Drug Saf, 2007, 30(10): 891-898. DOI:10.2165/00002018-200730100-00007 |
| [3] |
Arnaud M, Bégaud B, Thurin N, et al. Methods for safety signal detection in healthcare databases: a literature review[J]. Expert Opin Drug Saf, 2017, 16(6): 721-732. DOI:10.1080/14740338.2017.1325463 |
| [4] |
Kulldorff M, Fang ZX, Walsh SJ. A tree-based scan statistic for database disease surveillance[J]. Biometrics, 2003, 59(2): 323-331. DOI:10.1111/1541-0420.00039 |
| [5] |
Kulldorff M, Dashevsky I, Avery TR, et al. Drug safety data mining with a tree-based scan statistic[J]. Pharmacoepidemiol Drug Saf, 2013, 22(5): 517-523. DOI:10.1002/pds.3423 |
| [6] |
李海龙, 赵厚宇, 周一帆, 等. 基于电子医疗数据库的药品不良反应信号挖掘方法概述[J]. 药物流行病学杂志, 2018, 27(8): 541-549. Li HL, Zhao HY, Zhou YF, et al. Data mining methods for adverse drug reaction signals detection in healthcare databases: a literature review[J]. Chin J Pharmacoepidemiol, 2018, 27(8): 541-549. |
| [7] |
Moher D, Liberati A, Tetzlaff J, et al. Preferred reporting items for systematic reviews and Meta-analyses: the PRISMA statement[J]. PLoS Med, 2009, 6(7): e1000097. DOI:10.1371/journal.pmed.1000097 |
| [8] |
Brown JS, Petronis KR, Bate A, et al. Drug adverse event detection in health plan data using the gamma Poisson shrinker and comparison to the tree-based scan statistic[J]. Pharmaceutics, 2013, 5(1): 179-200. DOI:10.3390/PHARMACEUTICS5010179 |
| [9] |
Wang SV, Maro JC, Baro E, et al. Data mining for adverse drug events with a propensity score-matched tree-based scan statistic[J]. Epidemiology, 2018, 29(6): 895-903. DOI:10.1097/EDE.0000000000000907 |
| [10] |
Schachterle SE, Hurley S, Liu Q, et al. An implementation and visualization of the tree-based scan statistic for safety event monitoring in longitudinal electronic health data[J]. Drug Saf, 2019, 42(6): 727-741. DOI:10.1007/s40264-018-00784-0 |
| [11] |
Maro JC, Nguyen MD, Izem R, et al. Signal detection using tree-temporal scan statistics in the sentinel system[J]. Pharmacoepidemiol Drug Saf, 2018, 27: 346-347. |
| [12] |
Li HL, Zhang LL, Zhan SY. Adverse reaction signal detection for statins in a Chinese regional healthcare database using tree based scan statistic method[J]. Pharmacoepidemiol Drug Saf, 2019, 28: 186-187. |
| [13] |
Li HL, Zhang LL, Zhan SY. Drug safety signal detection in a Chinese regional healthcare database using the tree-based scan statistic and comparison to 3 other statistical methods[J]. Pharmacoepidemiol Drug Saf, 2019, 28: 399. |
| [14] |
Brown JS, Petronis K, Bate A, et al. Comparing two methods for detecting adverse event signals in observational data: Empirical bayes gamma poisson shrinker vs. tree-based scan statistic[J]. Pharmacoepidemiol Drug Saf, 2011, 20: S144. |
| [15] |
Brown JS, Dashevsky I, Fireman B, et al. Data mining with a tree-based scan statistic[J]. Pharmacoepidemiol Drug Saf, 2011, 20: S331. DOI:10.1002/pds.2097 |
| [16] |
Yih WK, Kulldorff M, Dashevsky I, et al. Using the self-controlled tree-temporal scan statistic to assess the safety of live attenuated herpes zoster vaccine[J]. Am J Epidemiol, 2019, 188(7): 1383-1388. DOI:10.1093/aje/kwz104 |
| [17] |
Yih WK, Maro JC, Nguyen M, et al. Assessment of quadrivalent human papillomavirus vaccine safety using the self-controlled tree-temporal scan statistic signal-detection method in the sentinel system[J]. Am J Epidemiol, 2018, 187(6): 1269-1276. DOI:10.1093/aje/kwy023 |
| [18] |
Li RX, Weintraub E, Mcneil MM, et al. Meningococcal conjugate vaccine safety surveillance in the Vaccine Safety Datalink using a tree-temporal scan data mining method[J]. Pharmacoepidemiol Drug Saf, 2018, 27(4): 391-397. DOI:10.1002/pds.4397 |
| [19] |
Kim JH, Lee H, Shin JY. Bacillus Calmette-Guérin (BCG) vaccine safety surveillance in the Korea Adverse Event Reporting System using the tree-based scan statistic and conventional disproportionality-based algorithms[J]. Vaccine, 2020, 38(21): 3702-3710. DOI:10.1016/j.vaccine.2020.04.007 |
| [20] |
Liu CH, Juan YC, Yang YY, et al. Varicella vaccine safety surveillance using a tree-based scan statistic[J]. Pharmacoepidemiol Drug Saf, 2018, 27: 394. |
| [21] |
Kulldorff M, Nguyen M, Balsbaugh C, et al. Self-control tree scan data mining for vaccine adverse events[J]. Pharmacoepidemiol Drug Saf, 2013, 22: 384-385. |
| [22] |
李海龙, 赵厚宇, 詹思延. 树状扫描统计量在药品安全性监测中的应用[J]. 药物流行病学杂志, 2018, 27(3): 209-213. Li HL, Zhao HY, Zhan SY. The application of tree-based scan statistic in drug safety surveillance[J]. Chin J Pharmacoepidemiol, 2018, 27(3): 209-213. |
| [23] |
Kulldorff M. A spatial scan statistic[J]. Commun Stat Theory Methods, 1997, 26(6): 1481-1496. |
| [24] |
Kulldorff M. SaTScan user guide for version 9.7[EB/OL]. (2021-01-15)[2021-05-05]. https://www.satscan.org/cgi-bin/satscan/register.pl/SaTScan_Users_Guide.pdf?todo=process_userguide_download.
|
| [25] |
Glaz J, Naus J, Wallenstein S. Scan statistics[M]. New York: Springer-Verlag, 2001.
|
 2021, Vol. 42
2021, Vol. 42




