文章信息
- 王秋伟, 黄鹏, 岳明, 黄恩炯, 蔡怡珊, 梁培栋, 姚苹苹, 张云, 谭伟龙, 喻荣彬.
- Wang Qiuwei, Huang Peng, Yue Ming, Huang Enjiong, Cai Yishan, Liang Peidong, Yao Pingping, Zhang Yun, Tan Weilong, Yu Rongbin
- 福建省宁德港地区2020年夏季鼠形动物汉坦病毒基因特征分析
- Genetic characteristics of hantavirus from rodents in port area of Ningde, Fujian province in the summer of 2020
- 中华流行病学杂志, 2021, 42(7): 1266-1273
- Chinese Journal of Epidemiology, 2021, 42(7): 1266-1273
- http://dx.doi.org/10.3760/cma.j.cn112338-20210126-00065
-
文章历史
收稿日期: 2021-01-26
2. 江苏省人民医院, 南京 210029;
3. 福州市国际旅行卫生保健中心 350001;
4. 浙江省疾病预防控制中心, 杭州 310051;
5. 中国人民解放军东部战区疾病预防控制中心, 南京 210002
2. Jiangsu Province Hospital, Nanjing 210029, China;
3. Fuzhou International Travel Health Care Center, Fuzhou 350001, China;
4. Zhejiang Provincial Center for Disease Control and Prevention, Hangzhou 310051, China;
5. Eastern Theater Command Centers for Disease Control and Prevention, Chinese People's Liberation Army, Nanjing 210002, China
汉坦病毒(HV)为布尼亚病毒目HV科正HV属的单股负链RNA病毒。基因结构包括L、M、S 3个片段,分别编码RNA依赖的RNA聚合酶、病毒糖蛋白以及核蛋白[1]。该病毒主要通过啮齿动物感染人类,引起肾综合征出血热(HFRS)和HV肺综合征(HPS)。据最新统计,全球每年发生HV相关疾病超过50 000例,HFRS和HPS致死率分别为12%和40%,主要疫区在亚洲地区[2]。中国是全球HFRS发病率最高的国家,1950-2010年我国有29个省份报告HFRS病例超过140万例,对我国人民健康造成了巨大的疾病负担[3-4]。目前我国已发现约11种HV基因型/血清型,其中汉滩病毒(HTNV)和汉城病毒(SEOV)是主要基因型,主要动物宿主分别为黑线姬鼠和褐家鼠[5-6]。
福建省沿海地区鼠密度高,港口环境为HV的传播提供良好条件[7-8]。根据中国CDC法定传染病报告,2004-2017年福建省共报告3 802例HFRS病例,高发疫区主要集中在宁德市、福州市等地,基因型主要以SEOV为主,优势鼠种为褐家鼠[9];福建省宁德港地区(宁德港)HV及宿主鲜有报道,2020年夏季在宁德港开展鼠形动物及其携带HV的调查,分析该地区鼠形动物分布情况及其携带HV基因特征及遗传进化关系。
对象与方法1. 样本采集:2020年6-8月在宁德港进行夹夜法鼠形动物采集,采集后分类鉴定、计数,进行标本解剖,采集鼠肺并编号存放,样本保存于-80 ℃冰箱中待用。
2. HV检测:取30~50 mg鼠肺组织,使用PBS溶液充分研磨,使用QIAamp Viral RNA Mini Kit试剂盒(德国凯杰企业管理有限公司)提取鼠肺组织RNA;采用荧光定量PCR法和HVⅠ、Ⅱ型通用核酸测定试剂盒(中国上海之江生物科技有限公司)对特异性RNA片段扩增检测,每个反应管内的荧光信号到达设定的域值时所经历的循环数(Ct值)≤38报告为阳性,将HV阳性样本送往中国深圳华大基因科技有限公司进行全基因组测序,并对相应的阳性样本利用相应引物进行验证[10]。
3. 序列分析:选取不同地区HV株与宁德港HV阳性样本进行序利比对,采用Dnastar 7.1软件进行DNA分析,其中SeqMan模块进行序列拼接及编辑,MegAlign版块进行核苷酸及氨基酸相似性分析;采用DNAMAN 9.0软件进行核苷酸及氨基酸的变异位点分析,并根据相应的免疫表位数据库(Immune epitope database and analysis resource)进行抗原表位变异分析;采用MEGA-X-10.0.5软件确定最优替代模型,使用GTR+G及GTR+G+I最优替代模型通过最大似然法基于S、M、L片段开放阅读框进行系统发育分析,分析采用1 000个多序列复制组。
结果1. HV筛查结果:本研究共采集鼠形动物112只,包括褐家鼠40只、黄胸鼠16只、小家鼠7只和鼩鼱49只;HV阳性鼠7只,HV总阳性率为6.25%(7/112),包括5只褐家鼠和2只黄胸鼠,这2类鼠的HV阳性率为12.50%(7/56)。
2. 序列特征分析:阳性样本测序共得到2株HV全基因组序列(分别命名为FJ35和FJ36),将所得到的2株全基因组序列的L、M、S片段分别在美国国家生物技术信息中心(National Center for Biotechnology Information,NCBI)中进行blast比对,结果显示2株HV均为SEOV。FJ35和FJ36全长L片段分别由6 527和6 530个核苷酸组成,开放阅读框位置为37~6 492核苷酸,共计6 456个核苷酸,编码2 151个氨基酸;M片段分别由3 651和3 635个核苷酸组成,编码糖蛋白的核苷酸位于第47~3 448位,编码1 133个氨基酸;S片段分别包括1 629和1 769个核苷酸,开放阅读框位于第43~1 332位,共计1 290个核苷酸,编码429个氨基酸组成相应的核蛋白。
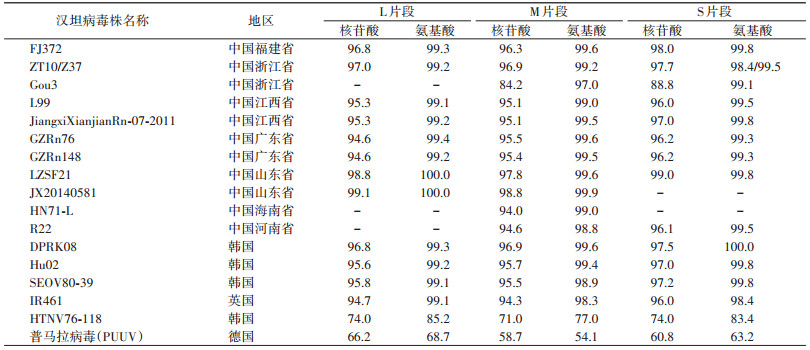
3. 核苷酸及氨基酸相似性分析:FJ35和FJ36 L、M、S片段核苷酸相似性分别为100.0%、99.9%、99.9%,推导的氨基酸序列完全相同。FJ35和FJ36与福建省2013年发现的病毒株FJ372/2013之间L、M、S片段核苷酸相似性分别为96.8%、96.3%、98.0%,氨基酸序列相似性为99.3%、99.6%、99.8%。与福建省其他HV L片段部分序列比较核苷酸相似性为91.3%~95.7%,氨基酸相似性为92.5%~95.7%。
2株阳性样本L、M和S片段核苷酸相似性与山东省病毒株JX20140581和LZSF21之间最高,核苷酸及氨基酸相似性分别为98.0%~99.0%,99.0%~100.0%;与浙江省、江西省、广东省等东南沿海省份3个节段核苷酸相似性分别为94.6%~97.0%、84.0%~97.0%、88.0%~97.0%,其氨基酸相似性除与S5亚型浙江省Gou株之间差异较大,与其他亚型之间约为99.0%;与国际标准株SEOV80-39之间核苷酸相似性为95.5%~97.0%,氨基酸相似性为98.9%~99.8%;与HTNV之间差异较大,核苷酸相似性仅为71.0%~74.0%,氨基酸相似性为76.0%~85.0%;与普马拉病毒之间核苷酸相似性为58.0%~66.0%,氨基酸相似性为54.0%~68.7%(表 1)。
4. 蛋白质二级结构预测分析:将推导的氨基酸进行分析发现,2株HV糖蛋白之间和核蛋白之间二级结构基本相似。糖蛋白主要以α螺旋和β折叠为主,而核蛋白主要以α螺旋为主。见图 1。

|
| 注:使用Garnier-Robson和Chou-Fasman两种方法进行蛋白质二级结构预测 图 1 2020年夏季福建省宁德港地区鼠形动物2株汉坦病毒(FJ35、FJ36)根据氨基酸序列推导的蛋白质 |
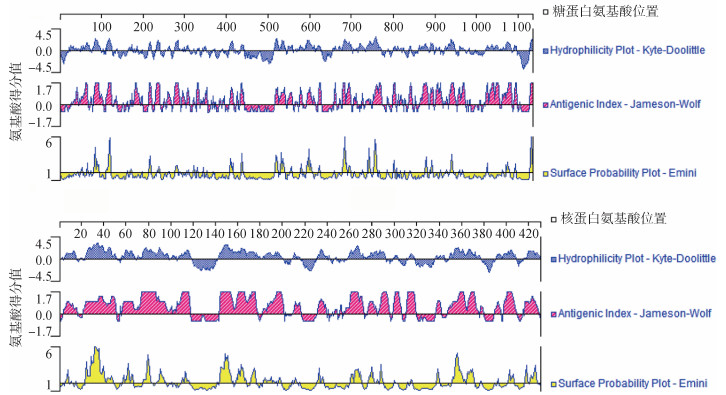
5. 蛋白质基本性质分析:FJ35和FJ36的糖蛋白N端有一明显的疏水区域且核蛋白的抗原性较强(图 2)。
|
| 注:2株汉坦病毒蛋白结构相同 图 2 2020年夏季福建省宁德港地区鼠形动物2株汉坦病毒(FJ35、FJ36)根据氨基酸序列推导的糖蛋白和核蛋白亲疏水性、抗原性和表面可能性分析 |
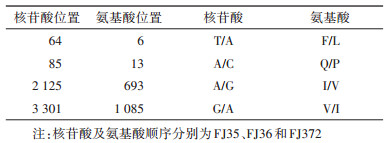
2株HV Gn糖蛋白的信号肽区域为第1~16位氨基酸,编码序列从第17位亮氨酸开始;Gc糖蛋白信号肽为第1~17位氨基酸,编码从第18位丙氨酸开始;对2株HV核蛋白分析未发现其信号肽区域。位于糖蛋白第642~646位氨基酸的WAASA基序为信号肽酶的识别位点,将M片段编码的糖蛋白切割成为含641个氨基酸的Gn糖蛋白和含487个氨基酸的Gc糖蛋白。
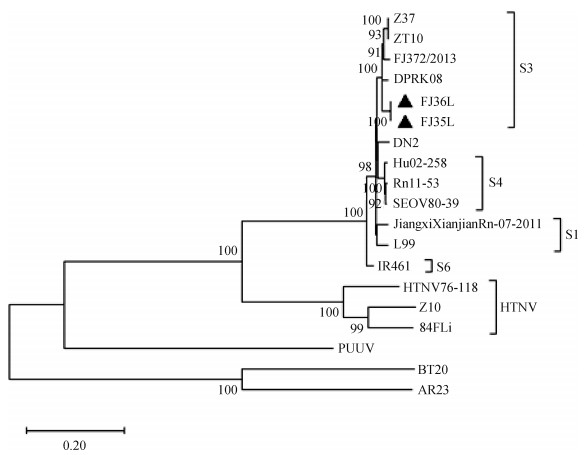
6. 系统发生分析:针对S、M、L片段开放阅读框运用GTR+G和GTR+G+I最优替代模型采用最大似然法构建系统发育树(图 3~5)。根据M片段构建的系统发育树发现FJ35和FJ36的HV阳性样本属于S3亚型,该亚型还包括其他地区HV分离株如中国山东省JX20140581和LZSF21、福建省FJ372、浙江省Z37和ZT10以及韩国DPRK08等。该样本与分离自中国山东省的JX20140581病毒株之间遗传距离最近,核苷酸相似性较高为98.95%。S片段构建的系统发生树与M片段的拓扑结构基本一致,根据S片段系统发生分析,FJ35和FJ36与山东省HV分离株LZSF21和SD201属于同一亚支,均为S3亚型,核苷酸相似性为99.00%。此外,L片段系统发生树也表明宁德港HV阳性样本属于S3亚型。

|
| 注:使用GTR+G最优替代模型根据最大似然法构建系统发生树 图 3 2020年夏季福建省宁德港地区鼠形动物2株汉坦病毒(FJ35、FJ36)与其他汉坦病毒代表株S片段构建系统发育树 |

|
| 注:S1~S6分别代表SEOV型汉坦病毒不同亚型,使用GTR+G最优替代模型构建系统发生树 图 4 2020年夏季福建省宁德港地区鼠形动物2株汉坦病毒(FJ35、FJ36)与其他汉坦病毒代表株M片段构建系统发育树 |
|
| 注:使用GTR+G+I最优替代模型利用最大似然法构建系统发生树 图 5 2020年夏季福建省宁德港地区鼠形动物2株汉坦病毒(FJ35、FJ36)与其他汉坦病毒代表株L片段构建系统发育树 |
7. 核苷酸及氨基酸变异位点分析:M片段为HV变异最快的节段,本研究新发现2株HV与福建省原病毒株FJ372相比,M片段编码的糖蛋白共发现4个突变位点(表 2),这些位置氨基酸的改变可能引起该病毒相关毒力的变化。
将2株HV与国际标准株SEOV80-39比较[11],发现8个在SEOV各亚型之间M片段突变频率超过40%的氨基酸位点,另外发现1个与抗原表位有关的氨基酸变异点,由SEOV80-39的第81位精氨酸R变为谷氨酰胺Q,因此与国际标准株相比,新发现HV阳性样本的糖蛋白抗原性可能发生改变。与疫苗株Z37和L99糖蛋白相比,福建省2株HV与Z37存在10个氨基酸变异位点,与L99存在11个变异位点,其中包括4个相同的变异位点(表 3)。
根据免疫抗原表位分析发现FJ35和FJ36样本编码的核蛋白抗原与FJ372之间存在差异,第259位氨基酸由天冬酰胺变异为丝氨酸,根据免疫表位抗原数据库第251~264位氨基酸区域为核蛋白的抗原表位,因此新发现的HV阳性样本的核蛋白抗原性可能与既往FJ372病毒株存在差异(图 6)。

|
| 图 6 2020年夏季福建省宁德港地区鼠形动物2株汉坦病毒(FJ35、FJ36)与汉城病毒亚型核蛋白的氨基酸变异分析 |
8. 疫源地变迁分析:研究表明S3亚型SEOV在全国各省份地区均有分布[12],根据基因相似性及系统发育分析,宁德港新发现的2株HV与S3亚型SEOV之间序列相似性高,且与山东省HV分离株LZSF21和JX20140581处于系统发育树的同一分支,推测该病毒沿渤海、黄海、东海、南海等海运方向传播扩散的可能性比较大,且可能经海运由山东省某疫源地HV相应宿主传入。此外,中国福建省宁德港新发现的HV与来自韩国的DPRK08在进化树上遗传距离相对较远,但同属于S3亚型,其核苷酸和氨基酸相似性分别为96.8%~97.5%和99.0%~100.0%,可能存在共同的祖先起源。
讨论20世纪60年代福建省首次发现HFRS,80年代发病数达到最高峰,此后逐渐下降并维持在较低水平,但近年来福建省HFRS发病数呈明显上升趋势,2004-2017年共报告HFRS病例3 644例,死亡20例[13]。既往监测结果显示,褐家鼠是福建省HFRS的主要宿主动物和传染源,疫区以家鼠型为主[14]。本研究在宁德港地区共采集鼠形动物112只,检测HV阳性7只,病毒携带率为6.25%,对阳性样品测序获得2条HV的全序列,经blast比对均为SEOV型。
对2株HV基因组糖蛋白及核蛋白二级结构分析发现,核蛋白具有较强的抗原性,可刺激机体产生相应的免疫反应,因此与FJ372相比其核蛋白N259S变异可能导致抗原性的改变;与国际标准株SEOV80-39相比,Q81R位氨基酸改变可能导致糖蛋白抗原性的改变。G1、G2片段编码的包膜糖蛋白对病毒的致病性起重要作用,是决定HV毒力、传染性、中和活性、血凝素和细胞融合等生物学特性的主要因素。该区基因变异如点突变、缺失、基因重排及糖基化方式的改变均有可能引起病毒毒力的改变,进而可能影响临床表现和病程转归[15-16]。本研究发现,宁德港2株HV M片段编码的糖蛋白与FJ372存在4个位点的变异,均可能引起病毒毒力、抗原性和细胞融合等改变。研究证实HTNV糖基化位点的改变可能影响相应蛋白的正确折叠以及对抗体的识别,本研究共发现6个糖基化位点与其他SEOV亚型一致,具有高度保守性[17]。与疫苗株Z37、L99 M片段所推导的氨基酸序列进行比对,发现新序列均存在明显的变异,这种现象可能导致现有疫苗对现阶段流行株保护作用降低甚至失去保护作用[12]。
为更准确地了解宁德港HV基因特征,对新发现的HV序列与不同地区的HV进行相似性及系统发生分析。核苷酸序列比对结果表明,2株HV之间基因相似性很高,核苷酸同源性为99.9%~100.0%,所推导的氨基酸序列完全相同,这种现象可能是因为宿主动物的主动或被动迁移导致。新发现的两序列与福建省从鼩鼱中分离得到的FJ372之间相似性较高,这可能与HV的宿主溢出现象有关[18]。通过系统发生分析发现该阳性样本所携带HV属于S3亚型,与韩国分离得到的DPRK08株属于同一亚型,可能存在共同的祖先起源[19];与中国浙江省Z37、山东省LZSF21、河北省93HBX12以及东北地区zy27、Gongzhuling415等也属于同一亚型,其中与山东省病毒株之间相似性最高为98.0%~99.0%,因此推测该阳性样本可能由山东省某疫源地传入,当然不排除其他疫源地入侵的可能。根据近年来HV的分子流行病学调查,SEOV的S3亚型在我国分布最为广泛,绝大部分病毒株的核苷酸及氨基酸序列具有较高的同源性,且无明显的地理聚集现象[20],这可能与其动物宿主褐家鼠有关,该动物在空间上比其他任何HV宿主更接近人类甚至可以通过轮船、火车等交通工具迁移到很远的地区[21]。
本研究存在不足。仅对HV糖蛋白及核蛋白氨基酸突变位点进行分析,未进行相应蛋白质及功能实验的验证,在今后的研究中,应结合相应的验证实验研究氨基酸变异对其功能的影响。
尽管宁德港新发现的HV阳性样本与东南沿海省份HV序列相似性较高,但仍存在某些氨基酸的变异从而引起相应抗原性的改变,因此应系统开展HV的流行病学及病原学检测,了解HV遗传进化特征,对预防控制HV具有重要的指导意义。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Avšič-Županc T, Saksida A, Korva M. Hantavirus infections[J]. Clin Microbiol Infect, 2019, 21S: e6-16. DOI:10.1111/1469-0691.12291 |
| [2] |
Watson DC, Sargianou M, Papa A, et al. Epidemiology of Hantavirus infections in humans: a comprehensive, global overview[J]. Crit Rev Microbiol, 2014, 40(3): 261-272. DOI:10.3109/1040841X.2013.783555 |
| [3] |
Tian HY, Hu SX, Cazelles B, et al. Urbanization prolongs hantavirus epidemics in cities[J]. Proc Natl Acad Sci USA, 2018, 115(18): 4707-4712. DOI:10.1073/pnas.1712767115 |
| [4] |
Zou LX, Chen MJ, Sun L. Haemorrhagic fever with renal syndrome: literature review and distribution analysis in China[J]. Int J Infect Dis, 2016, 43: 95-100. DOI:10.1016/j.ijid.2016.01.003 |
| [5] |
Kang YJ, Zhou DJ, Tian JH, et al. Dynamics of hantavirus infections in humans and animals in Wuhan city, Hubei, China[J]. Infect, Genet Evol, 2012, 12(8): 1614-1621. DOI:10.1016/j.meegid.2012.07.017 |
| [6] |
陈俊江, 国天赐, 宋姝璇, 等. 中国肾综合征出血热流行特征及时空分析模型进展[J]. 中华流行病学杂志, 2020, 41(10): 1735-1740. Chen JJ, Guo TC, Song SX, et al. Epidemiological characteristics and the development of spatiotemporal analysis models on hemorrhagic fever with renal syndrome in China[J]. Chin J Epidemiol, 2020, 41(10): 1735-1740. DOI:10.3760/cma.j.cn112338-20191108-00794 |
| [7] |
金达莱, 王进喜, 王光辉. 宁德口岸白马港鼠类及其寄生虫调查报告[J]. 中国国境卫生检疫杂志, 2000, 23(3): 154-156. Jin DL, Wang JX, Wang GH. Investigation report on rodents and their parasites in Baima Port at Ningde Port[J]. Chin Froniter Health Quarantine, 2000, 23(3): 154-156. DOI:10.3969/j.issn.1004-9770.2000.03.016 |
| [8] |
刘晓婷, 刘宝英, 吴济阳. 宁德港船厂鼠形动物及体表昆虫媒介调查[J]. 中国血吸虫病防治杂志, 2012, 24(2): 208-211. Liu XT, Liu BY, Wu JY. Investigation on rat-shaped animals and insect vectors on their body surface at Ningde Harbor[J]. Chin J Schisto Control, 2012, 24(2): 208-211. DOI:10.16250/j.32.1374.2012.02.005 |
| [9] |
何似, 陈阳, 王灵岚, 等. 福建省肾综合征出血热疫情及宿主动物监测分析[J]. 海峡预防医学杂志, 2011, 17(2): 8-10. He S, Chen Y, Wang LL, et al. Surveillance and analysis on HFRS epidemic situation and the host animals in Fujian province[J]. Strait J Prev Med, 2011, 17(2): 8-10. DOI:10.11853/j.issn.1003.4692.2014.02.025 |
| [10] |
王颖, 方志强, 李颖, 等. 2019年长白口岸鼠类携带汉坦病毒调查及其基因特征分析[J]. 中华卫生杀虫药械, 2020, 26(6): 546-548. Wang Y, Fang ZQ, Li Y, et al. Investigation and gene analysis of rodent-borne hantavirus at Changbai Port in 2019[J]. Chin J Hyg Insect Equip, 2020, 26(6): 546-548. DOI:10.19821/j.1671-2781.2020.06.011 |
| [11] |
Ning TT, Wang L, Liu S, et al. Monitoring neutralization property change of evolving hantaan and seoul viruses with a novel pseudovirus-based assay[J]. Virol Sin, 2020, 36(1): 304-112. DOI:10.1007/s12250-020-00237-y |
| [12] |
殷强玲. 中国流行性出血热重点流行区汉坦病毒遗传进化特征分析[D]. 北京: 中国疾病预防控制中心, 2019. Yin QL. Genetic characterization of hantavirus in key epidemic areas of hemorrhagic fever with renal syndrom in China[D]. Beijing: Chinese Center for Diease Control and Prevention, 2019. |
| [13] |
陈阳, 邓艳琴, 林代华, 等. 福建省部分地区首次确定肾综合征出血热疫源地的调查研究[J]. 中国媒介生物学及控制杂志, 2019, 30(6): 692-695. Chen Y, Deng YQ, Lin DH, et al. An investigation of firstly identified natural foci of hemorrhagic fever with renal syndrome in some areas of Fujian province, China[J]. Chin J Vector Biol Control, 2019, 30(6): 692-695. DOI:10.11853/j.issn.1003.8280.2019.06.023 |
| [14] |
陈阳, 林代华, 陈亮, 等. 福建省2013-2015年肾综合征出血热流行特征及宿主动物监测[J]. 中国媒介生物学及控制杂志, 2016, 27(6): 594-596. Chen Y, Lin DH, Chen L, et al. Surveillance of epidemic and host animals of hemorrhagic fever with renal syndrome in 2013-2015 in Fujian province, China[J]. Chin J Vector Biol Control, 2016, 27(6): 594-596. DOI:10.11853/j.issn.1003.8280.2016.06.019 |
| [15] |
Parrington MA, Lee PW, Kang CY. Molecular characterization of the prospect hill virus M RNA segment: a comparison with the M RNA segments of other hantaviruses[J]. J Gen Virol, 1991, 72(2): 1845-1854. DOI:10.1099/0022-1317-72-8-1845 |
| [16] |
杨其松, 张天琛, 刘晓青, 等. 汉坦病毒及其与宿主基因相互作用研究进展[J]. 中华疾病控制杂志, 2018, 22(7): 751-754. Yang QS, Zhang TC, Liu XQ, et al. Research progress of hantavirus and its interaction with host genes[J]. Chin J Dis Control Prev, 2018, 22(7): 751-754. DOI:10.16462/j.cnki.zhjbkz.2018.07.022 |
| [17] |
王文, 林献丹, 郭文平, 等. 褐家鼠携带2株汉坦病毒的遗传特征分析[J]. 中华流行病学杂志, 2010, 31(9): 1030-1034. Wang W, Lin XD, Guo WP, et al. Genetic analysis of hantaviruses carried by Rattus norvegicus collected from Henan and Neimenggu provinces, China[J]. Chin J Epidemiol, 2010, 31(9): 1030-1034. DOI:10.3760/cma.j.issn.0254-6450.2010.09.017 |
| [18] |
Wang YP, Zhang XL, Zhang JM, et al. Complete genome sequence of a novel mutation of seoul virus isolated from suncus murinus in the Fujian province of China[J]. Genome Announc, 2015, 3(2): e00075-15. DOI:10.1128/genomeA.00075-15 |
| [19] |
Plyusnina A, Laakkonen J, Niemimaa J, et al. Genetic analysis of hantaviruses carried by Myodes and Microtus rodents in Buryatia[J]. Virol J, 2008, 5: 4. DOI:10.1186/1743-422x-5-4 |
| [20] |
Li J, Zhao ZT, Wang ZQ, et al. Nucleotide sequence characterization and phylogenetic analysis of hantaviruses isolated in Shandong province, China[J]. Chin Med J (Engl), 2007, 120(9): 825-830. |
| [21] |
Plyusnin A, Morzunov SP. Virus evolution and genetic diversity of hantaviruses and their rodent hosts[J]. Curr Top Microbiol Immunol, 2001, 256: 47-75. DOI:10.1007/978-3-642-56753-7_4 |
 2021, Vol. 42
2021, Vol. 42