文章信息
- 曲梅, 黄瑛, 田祎, 张新, 贾蕾, 吕冰, 王全意.
- Qu Mei, Huang Ying, Tian Yi, Zhang Xin, Jia Lei, Lyu Bing, Wang Quanyi
- 北京市2010-2020年多重耐药肯塔基沙门菌流行特征分析
- Analysis on epidemiological characteristics of multidrug-resistant Salmonella Kentucky in Beijing, 2010-2020
- 中华流行病学杂志, 2021, 42(7): 1252-1259
- Chinese Journal of Epidemiology, 2021, 42(7): 1252-1259
- http://dx.doi.org/10.3760/cma.j.cn112338-20201206-01382
-
文章历史
收稿日期: 2020-12-06
沙门菌属是一种重要的人兽共患病原菌,也是引起感染性腹泻和食物中毒最常见的病原菌之一。每年全球因沙门菌感染引起的肠胃炎有9 400万例,死亡人数高达15万,导致不同程度的暴发和流行[1]。WHO最近将产生抗氟喹诺酮类药物的沙门菌优先列为对人类健康构成最大威胁的病原体[2],因此沙门菌感染及日趋严重的耐药问题已成为全世界关注的公共卫生问题。
肯塔基沙门菌是一种肠道沙门菌血清型。自1937年报道以来一直与家禽密切相关,之后相继在各种食源性动物家禽与腹泻患者体内检出。环丙沙星耐药的ST198肯塔基沙门菌,自2000年以来在全球范围内广泛流行,在食物链传播及进化过程中,产生多重耐药菌株,尤其是对喹诺酮类药物和三代头孢菌素产生的抗性引起高度关注[3-4]。在我国近年来也有人感染病例及污染食物的报道[5-7]。北京市自2010年以来开展肠道门诊腹泻病例沙门菌的监测,为了解肯塔基沙门菌在北京市的流行情况,本研究通过10年来沙门菌的回顾性调查,对腹泻患者中分离到的22株肯塔基沙门菌进行抗生素耐药和PFGE和全基因组测序分析,以掌握肯塔基沙门菌的分子流行病学特征和耐药特性,为北京市的非伤寒沙门菌监测、临床治疗用药以及风险预测提供依据。
材料与方法1. 菌株来源:肯塔基沙门菌共22株。其中13株来自2010-2020年北京市肠道门诊腹泻病例-沙门菌监测项目;另外9株来自2020年7月的一起腹泻聚集性疫情分离到的菌株。
2. 实验方法:
(1)粪便沙门菌的分离培养:参照卫生部行业标准WS 271-2008附录B进行,生化反应符合并且血清抗原式符合[8,20:i:z6]菌株,鉴定为肯塔基沙门菌。
(2)药敏试验:采用最小抑菌浓度(minimal inhibition concentration,MIC)法,选择28种抗生素:氨苄西林(AMP)、氨苄西林-舒巴坦(AMS)、四环素(TET)、氯霉素(CHL)、复方新诺明(SXT)、头孢唑林(CFZ)、头孢西丁(FOX)、头孢他啶(CAZ)、头孢噻肟(CTX)、头孢吡肟(FEP)、庆大霉素(GEN)、阿奇霉素(AZI)、环丙沙星(CIP)、链霉素(STR)、磺胺异噁唑(Sul)、萘啶酸(NAL)、阿莫西林-克拉维酸(AMC)、强力霉素(DOX)、氨曲南(AZM)、亚胺培南(IMI)、美罗培南(MEM)、卡那霉素(KAN)、阿米卡星(AMI)、左氧氟沙星(LEV)、吉米沙星(GMI)、米诺环素(MIN)、多粘菌素E(CT)以及多粘菌素B(PB)进行沙门菌耐药性检测。结果与美国临床实验室标准化协会(clinical and laboratory standards institute,CLSI)2018年标准比较,以大肠埃希菌(ATCC25922)为质控菌株。耐药种类≥3种抗生素,既为多重耐药(multidrug resistance,MDR)。
(3)超广谱β-内酰胺酶(extended-spetrum beta-lactamase,ESBL)表型确证试验:参照CLSI推荐的MIC法标准进行,在初筛实验中头孢他啶或头孢噻肟MIC≥2 μg/ml;在ESBL表型确证试验中,对两组中任何一个药物,在加克拉维酸后MIC值与不加克拉维酸的MIC相比,降低值≤3个倍比稀释浓度,确证该菌株产ESBLs。
3. 全基因组测序:细菌基因组DNA提取采用DNA mini Kit试剂盒,由德国Qiagen凯杰企业管理(上海)有限公司生产,按操作手册进行提取。核酸的浓度用Qubit DNA-HS进行定量,经测定所有样品的A260/A280比值均在1.8~2.0之间,基因组DNA的浓度 > 100 ng/μl,每个样品的最终体积是50 μl。委托中国北京诺禾致源生物公司基于Illumina PE150平台进行双末端高通量测序。对测序数据进行质量控制,去除低质量reads和接头序列,数据过滤后得到Clean Data,利用SPAdes(v3.13)软件进行denovo组装,得到基因组的contigs序列。上传至基因组流行病学中心数据平台(Center for Genomic Epidemiology,http://www.genomicepidemiology.org/)进行多位点序列分型(MLST)及耐药基因鉴定。并依据文献[8]在全基因组序列中筛查沙门基因组岛1(Salmonella genomic island,SGI1)及其变异体(SGI1-K、P和Q)。
4. PFGE分型:参照PulseNet制定的沙门菌PFGE标准分型方法。内切酶XbaⅠ(40 U),37 ℃酶切3 h。电泳条件:电压6 V/cm,脉冲时间从2.2~63.8 s,线性转换,转换角度120°,电泳时间19 h,电泳温度14 ℃。沙门菌标准株H9812作为分子量标记。采用BioNumerics 5.0软件对电泳图像进行处理和比对分析。聚类树状图根据非加权配对算术平均法(unweighted pair-group method using arithmetic averages,UPGMA)构建。聚类相似性系数采用距离法计算,得到菌株带型相似性的聚类分析树。
结果1. 流行情况:北京市自2010年开展肠道门诊腹泻病例的沙门菌监测,2016年之前从未分离到肯塔基沙门菌,2016年开始在散发腹泻病例中分离到肯塔基沙门菌,2016年(9株)、2018年(2株)、2019年(2株)、2020年(1株);另外2020年7月某区的一起10人的聚集性腹泻疫情分离到9株菌;共22株菌。这些菌株的病例年龄26~70岁,平均年龄42.2岁,男女性比例为2.1∶1(15∶7)。
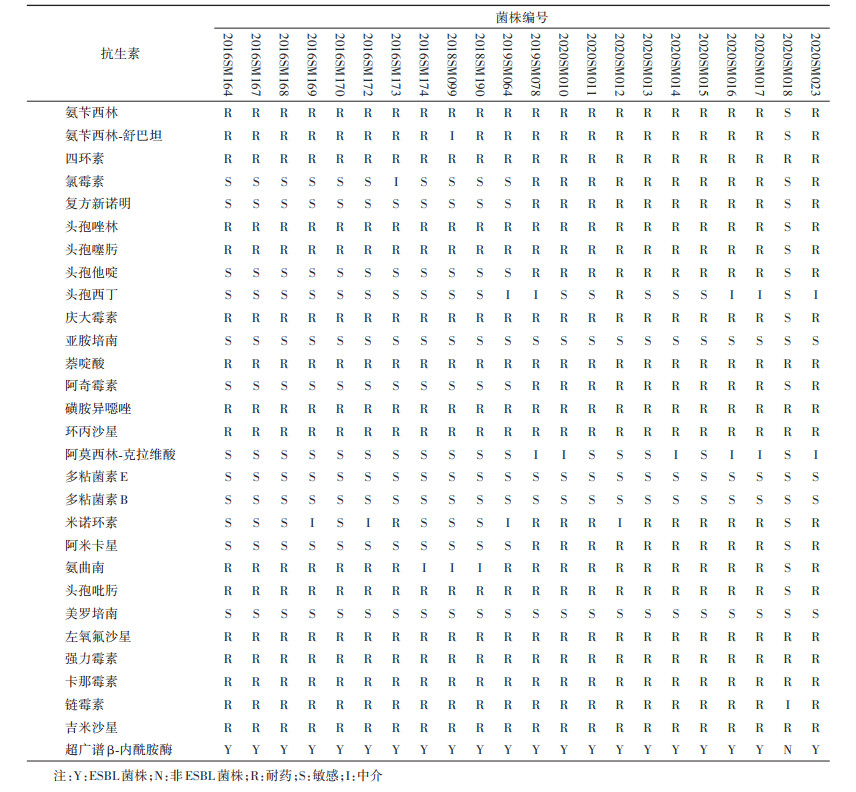
2. 耐药特征:药物敏感试验结果见表 1。肯塔基沙门菌对大部分β-内酰胺类、喹诺酮类、氨基糖甙类以及四环素类药物耐药严重。22株菌对TET、DOX、Sul、KAN、NAL、CIP、LEV和GMI全部耐药;对AMP、CFZ、CTX、FEP、GEN和STR 21株耐药(95.5%);对单环内酰胺类AZM 18株耐药(81.8%);对其他各类抗生素的耐药率也均在45.5%以上。除菌株2020SM018外,21株菌是ESBL阳性。22株菌对IMI、MEM、CT和PB完全敏感;对AMC的敏感率为72.7%,无耐药。多重耐药分析显示,全部菌株均对8种以上抗生素耐药,最多可达对22种抗生素的耐药,有多种耐药表型。其中9株菌(40.9%)表现为最长的耐药模式:AMP-AMS-TET-CHL-SXT-CFZ-CTX-CAZ-GEN-NAL-AZI-Sul-CIP-MIN-AMI-AZM-FEP-LEV-DOX-KAN-STR-GMI;7株菌(31.8%)对16种药物耐药,模式:AMP-AMS-TET-CFZ-CTX-GEN-NAL-Sul-CIP-AZM-FEP-LEV-DOX-KAN-STR-GMI。
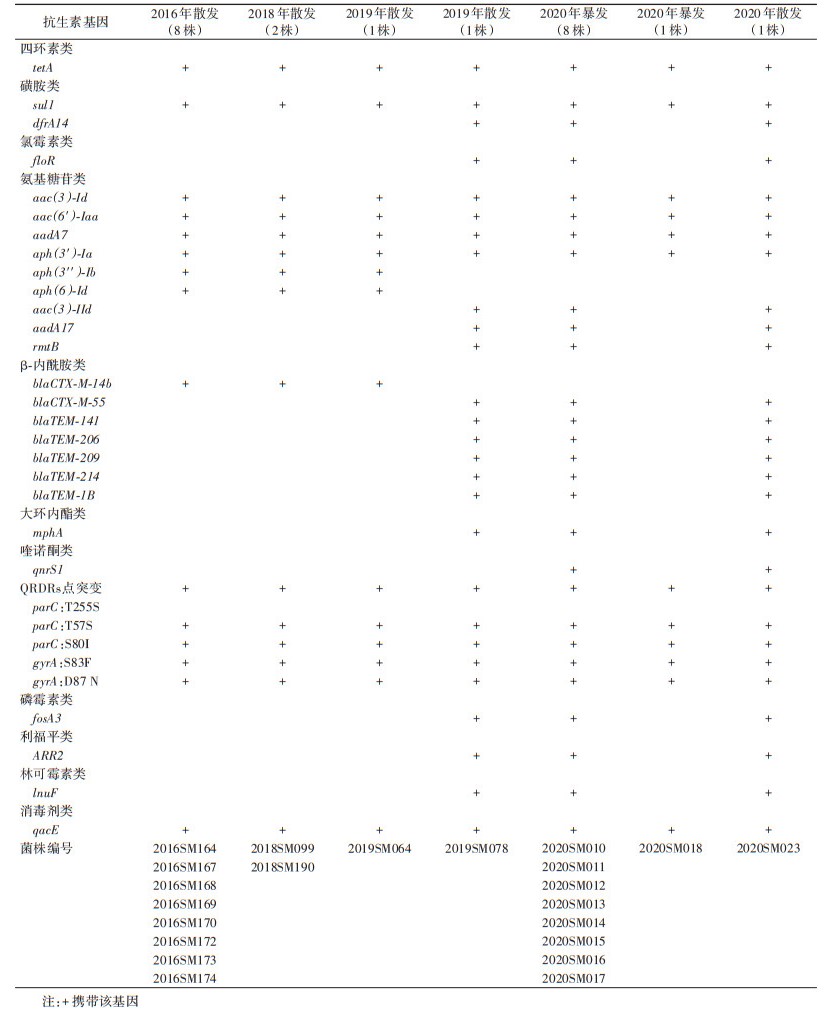
3. 全基因组测序分析MLST、SGI-1基因组岛和耐药基因携带情况:22株菌的全基因测序数据上传至CGE基因组流行病学中心,进行MLST分型和耐药基因筛查。22株菌均为相同的MLST序列型:ST198(aroC_76、dnaN_14、hemD_3、hisD_77、purE_64、sucA_64、thrA_67)。22株菌均携带SGI1-K基因组岛。耐药基因鉴定显示(表 2):22株均携带四环素类耐药基因tetA、磺胺类耐药基因sul1、消毒剂类耐药基因qacE、喹诺酮耐药决定区(quinolone resistance determining regions,QRDRs)gyrA基因2个位点突变(S83F、D87 N)、parC基因3个位点的突变(T255S、T57S、S80I)。另外,β-内酰胺类相关耐药基因7种(blaCTX-M-55、blaCTX-M-14b、blaTEM- 141、blaTEM-206、blaTEM-209、blaTEM-214、blaTEM- 1B)、氨基糖甙类耐药相关基因9种[aac(3)-Id、aac(3)-IId、aac(6')-Iaa、aadA7、aadA17、aph(3')-Ia、aph(3")-Ib、aph(6)-Id、rmtB]、氯霉素类(floR)、甲氧苄氨嘧啶类(dfrA14)、大环内酯类(mphA)、喹诺酮类(qnrS1)、磷霉素类(fosA3)、利福平类(ARR2)和林可霉素类(InuF),以上这些耐药基因,除菌株2020SM018外,在散发菌株和暴发菌株间存在明显差异。表现为自2019SM078以后的菌株获得耐药基因的数量和种类明显增多,相对应的抗生素耐药表型由敏感变成了耐药:如CHL、SXT、CAZ、AZI和AMI(表 1);而且多达10种药物对肯塔基沙门菌的MIC值也成倍数显著增高(表 3):如CHL的MIC值由原来4 μg/ml到64 μg/ml,提高16倍、SXT提高32倍、CAZ提高4倍、AZI提高8~16倍、氨基糖甙类GEN和AMI提高2~32倍、四环素类MIN提高4倍、氟喹诺酮类CIP和GMI提高2~4倍、AZM提高2倍。其中CIP MIC值> 32 μg/ml,CTX MIC值> 8 μg/ml,SXT MIC值> 8/152 μg/ml,AZI MIC值64 μg/ml,都表现为超高水平耐药。
4. PFGE聚类及遗传进化分析:PFGE聚类分析显示,22株之间存在0~4条带的差异,相似性 > 85%,亲缘关系相近,属于同一克隆来源,与全球广泛传播的CIP耐药的ST198-X1克隆株比较[8],带型高度相似,故属于该国际流行株。22株菌分为2大聚类簇(图 1)。2016年8株散发菌株与2018年2株散发菌株带型一致,属于Cluster B,菌株2019SM064与其有1条带的差异,相似度为96%,也属于Cluster B。在Cluster A中,2020年的9株暴发菌株中有7株带型完全一致,与其他2株菌2020SM014和2020SM018存在1~2条带的差异,相似度也 > 96%,由于这是一起聚集性腹泻疫情分离到的9株菌,存在流行病学关联,菌株高度同源,认为来自同一克隆株;另外Cluster A中还包括1株散发菌株2019SM078,虽然与上述菌株间亲缘关系稍远,但相似度也在90%以上。值得注意的是2020SM018株菌,相较于其他暴发菌株缺少了1条170 kb左右的大片段,造成耐药表型与耐药基因也与其他菌株存在明显差异。

|
| 注:AMP:氨苄西林;AMS:氨苄西林-舒巴坦;TET:四环素;CHL:氯霉素;SXT:复方新诺明;CFZ:头孢唑林;CTX:头孢噻肟;CAZ:头孢他啶;FOX:头孢西丁;GEN:庆大霉素;IMI:亚胺培南;NAL:萘啶酸;AZI:阿奇霉素;Sul:磺胺异噁唑;CIP:环丙沙星;AMC:阿莫西林-克拉维酸;CT:多粘菌素E;PB:多粘菌素B;MIN:米诺环素;AMI:阿米卡星;AZM:氨曲南;FEP:头孢吡肟;MEM:美罗培南;LEV:左氧氟沙星;DOX:强力霉素;KAN:卡那霉素;STR:链霉素;GMI:吉米沙星;ESBL:超广谱β-内酰胺酶;黑色方块表示菌株对抗生素有抗性,无色表示中介或敏感 图 1 22株肯塔基沙门菌聚类分析 |
ST198肯塔基沙门菌是20世纪90年代中期在埃及首先被发现,是肯塔基沙门菌多耐药流行株的共同祖先。2000年以后,在进化演变过程中,获得了携带多种抗药基因的沙门菌基因组岛(SGI1)及其突变体(SGI-1K)等[9],其包含复杂的Ⅰ类整合子、插入序列(IS26)和转座子等可移动遗传元件,而具有了对CIP、AMP、STR、GEN、Sul和TET等多种抗生素的高水平耐药[10],在全球广泛传播。有流行病学证据证实,该流行株已通过旅游者、动物性食品、外环境以及家禽、宠物、野生动物等多种感染来源,在印度次大陆、东南亚地区和欧洲地区等快速播散[3-4, 8, 11],成为当地非伤寒沙门菌重点监测的国际流行株。而我国关于此血清型流行株的报道相对较少。最早的文献记录是2009年7月在四川省的一起食物中毒事件中分离到肯塔基沙门菌[12],但因缺乏详细的实验室数据,无法确定是否为国际流行株。2016年,从浙江省[6]和广东省[7]的腹泻患者粪便和市售鸡肉中分离到肯塔基沙门菌,对环丙沙星和头孢菌素等抗生素表现为高水平耐药。这意味着多重耐药肯塔基沙门菌在我国人和动物之间可能发生了一定传播,对公共卫生造成威胁。
本研究通过对2010年以来沙门菌监测数据的回顾性分析发现,2016-2020年北京市腹泻患者中分离的22株肯塔基沙门菌,属于国际上流行的环丙沙星耐药的ST198-X1-SGI1-K克隆株[8, 10],在2016年传入北京市,当年从散发腹泻患者中分离到8株肯塔基沙门菌,由于时间间隔不到1个月,地点都集中在某一个区,流行病学上存在密切关联,而且耐药谱与PFGE图谱完全一致,所以推测该流行株在北京市造成小范围的局部流行,且与2016年浙江省腹泻患者和食品中分离到的肯塔基沙门菌PFGE图谱完全一致,说明该克隆株在2016年国内其他地区也有扩散,这一带型的克隆株在北京市2018年还持续存在,保持着低水平传播,但在扩散过程中发生累积的变异,可以看到2019年分离到的1株菌2019SM078和2020年以后的菌株在抗生素选择压力下,耐药谱和PFGE图谱都发生了一些变化,由Cluster B群演变成Cluster A群。PFGE图谱个别条带的变化,展现出ST198-X1亚型的高度多样性,可能与IS26转座活性和拷贝数变异引起的基因插入、缺失或转座,导致菌株出现独特的基因重排形式有关[10],这需要后续进一步的实验和对基因组学数据的深度挖掘来验证。
细菌在药物长期的选择压力作用下,耐药性会不断增加,耐药表型与耐药基因之间往往存在着密切联系。喹诺酮类药物的耐药性与QRDRs基因的点突变有关[13]。本研究通过全基因组序列分析,能准确鉴定出菌株所携带的全部耐药基因。所有菌株QRDRs的gyrA基因都发生了2个突变:S83F、D87 N;parC基因有3个突变:T57S、S80I、T255S,与环丙沙星耐药肯塔基沙门菌的相关报道一致[8, 11, 14]。值得注意的是,质粒介导的喹诺酮类耐药基因qnrS1,仅在2020年分离的菌株中有检出,而其他同类研究则未见报道。ESBL也是由质粒介导的酶,可以水解青霉素、AZM和头孢菌素等,对CTX的水解活性远高于CAZ,可被β-内酰胺酶抑制剂抑制。本研究除菌株2020SM018外,CTX-M型和/或TEM型ESBL耐药基因在所有菌株中均有检出,但不同年代菌株基因亚型不同,在聚类图上明显分为两大基因簇,而且这些耐药基因的分布情况与菌株所表现出的对AMP和CTX高度耐药,对CAZ部分耐药,对β-内酰胺酶抑制剂AMC高度敏感的表型特征一致。与2016年宁波市分离到的来自人源和食品菌株的耐药结果基本一致[6]。磷酸转移酶mphA基因,可灭活大环内酯类抗微生物药物[8],本研究在2019年以后菌株中检测到mphA基因,并表现出对AZI的高耐药(MIC为32~64 μg/ml),应引起高度关注。另外,还发现所有菌株均携带季胺类化合物消毒剂耐药基因qacE及磺胺类耐药基因sul1,qacE-sul1位于Ⅰ类整合子3'端[10],这反映出北京市肯塔基沙门菌流行株对环境消毒剂和防腐剂已出现耐药,今后对此流行株造成的院内感染和养殖业污染的防控将更为严峻。
氟喹诺酮类和三代头孢类抗生素是目前治疗沙门菌感染的首选用药[15]。AZI是治疗重症沙门菌感染的最佳用药[16]。本研究发现,北京市自2016年以来出现多重耐药ST198-X1肯塔基沙门菌的流行,该流行株不但对氟喹诺酮类中的CIP、LEV呈现高水平耐药,而且对Ⅲ、Ⅳ代头孢菌素和AZI也出现了严重耐药,对8种以上抗生素表现为多重耐药。这种超级耐药菌给临床用药带来极大挑战,结合本研究结果,临床治疗时可选用碳青霉烯类抗生素以及β-内酰胺酶抑制剂的复合剂AMC。ST198多重耐药肯塔基沙门菌在广泛传播过程中不断获得最新的耐药性,被视为全球高风险克隆株,应在加强系统监测的同时,规范抗生素的合理使用。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Majowicz SE, Musto J, Scallan E, et al. The global burden of nontyphoidal Salmonella gastroenteritis[J]. Clin Infect Dis, 2010, 50(6): 882-889. DOI:10.1086/650733 |
| [2] |
Tacconelli E, Carrara E, Savoldi A, et al. Discovery, research, and development of new antibiotics: the WHO priority list of antibiotic-resistant bacteria and tuberculosis[J]. Lancet Infect Dis, 2018, 18(3): 318-327. DOI:10.1016/S1473-3099(17)30753-3 |
| [3] |
Le Hello S, Hendriksen RS, Doublet B, et al. International spread of an epidemic population of Salmonella enterica serotype Kentucky ST198 resistant to ciprofloxacin[J]. J Infect Dis, 2011, 204(5): 675-684. DOI:10.1093/infdis/jir409 |
| [4] |
Antunes P, Mourão J, Campos J, et al. Salmonellosis: the role of poultry meat[J]. Clin Microbiol Infect, 2016, 22(2): 110-121. DOI:10.1016/j.cmi.2015.12.004 |
| [5] |
牛惠敏, 杨玲, 王艳芬. 一起肯塔基沙门氏菌食物中毒事件调查[J]. 医学动物防制, 2017, 33(8): 888-889. Niu HM, Yang L, Wang YF. Investigation on food poisoning incident of Salmonella kentucky[J]. J Med Pest Control, 2017, 33(8): 888-889. DOI:10.7629/yxdwfz201708024 |
| [6] |
杨元斌, 高红, 章丹阳, 等. 浙江省宁波市多重耐药肯塔基沙门菌的检出及病原学分析[J]. 疾病监测, 2018, 33(6): 510-514. Yang YB, Gao H, Zhang DY, et al. Etiology of multi-drug resistant Salmonella Kentucky isolates from retail chicken and a patient in Ningbo[J]. Dis Surveill, 2018, 33(6): 510-514. DOI:10.3784/j.issn.1003-9961.2018.06.015 |
| [7] |
王少君, 孙康泰, 熊智颖, 等. 广东省零售市场鸡肉中肯塔基沙门菌的流行情况及耐药性分析[J]. 畜牧兽医学报, 2019, 50(12): 2509-2517. Wang SJ, Sun KT, Xiong ZY, et al. Analysis of prevalence and drug resistance of Salmonella Kentucky in chicken from retail markets in Guangdong[J]. Acta Vet Zootechn Sin, 2019, 50(12): 2509-2517. DOI:10.11843/j.issn.0366-6964.2019.12.015 |
| [8] |
Le Hello S, Bekhit A, Granier SA, et al. The global establishment of a highly-fluoroquinolone resistant Salmonella enterica serotype Kentucky ST198 strain[J]. Front Microbiol, 2013, 4: 395. DOI:10.3389/fmicb.2013.00395 |
| [9] |
Le Hello S, Weill FX, Guibert V, et al. Early strains of multidrug-resistant Salmonella enterica serovar Kentucky sequence type 198 from Southeast Asia harbor Salmonella genomic island 1-J variants with a novel insertion sequence[J]. Antimicrob Agents Chemother, 2012, 56(10): 5096-5102. DOI:10.1128/AAC.00732-12 |
| [10] |
Hawkey J, Le Hello S, Doublet B, et al. Global phylogenomics of multidrug-resistant Salmonella enterica serotype Kentucky ST198[J]. Microb Genom, 2019, 5(7): e000269. DOI:10.1099/mgen.0.000269 |
| [11] |
Park AK, Shin E, Kim S, et al. Traveller-associated high-level ciprofloxacin-resistant Salmonella enterica Serovar Kentucky in the Republic of Korea[J]. J Glob Antimicrob Resist, 2020, 22: 190-194. DOI:10.1016/j.jgar.2019.12.014 |
| [12] |
王清, 代彦, 张勇. 首次在四川地区食物中毒中检出肯塔基沙门菌分析[J]. 现代预防医学, 2010, 37(20): 3914-3916. DOI: CNKI:SUN:XDYF.0.2010-20-043. Wang Q, Dai Y, Zhang Y. Detection Analysis on Salmonella Kentucky in food poisoning on the first time in the Sichuan region[J]. Mod Prev Med, 2010, 37(20): 3914-3916. DOI: CNKI:SUN:XDYF.0.2010-20-043. |
| [13] |
Jacoby GA, Strahilevitz J, Hooper DC. Plasmid-mediated quinolone resistance[J]. Microbiol Spectr, 2014, 2(5). DOI:10.1128/microbiolspec.PLAS-0006-2013 |
| [14] |
Shah DH, Paul NC, Guard J. Complete genome sequence of a ciprofloxacin-resistant Salmonella enterica subsp. enterica serovar kentucky sequence type 198 strain, PU131, isolated from a Human Patient in Washington State[J]. Genome Announc, 2018, 6(9): e00125-18. DOI:10.1128/genomeA.00125-18 |
| [15] |
Wen SC, Best E, Nourse C. Non-typhoidal Salmonella infections in children: Review of literature and recommendations for management[J]. J Paediatr Child Health, 2017, 53(10): 936-941. DOI:10.1111/jpc.13585 |
| [16] |
Hill DR, Beeching NJ. Travelers' diarrhea[J]. Curr Opin Infect Dis, 2010, 23(5): 481-487. DOI:10.1097/QCO.0b013e32833dfca5 |
 2021, Vol. 42
2021, Vol. 42