文章信息
- 刘亚倩, 汪天培, 颜财旺, 朱猛, 杨明, 王梦筠, 胡志斌, 沈洪兵, 靳光付.
- Liu Yaqian, Wang Tianpei, Yan Caiwang, Zhu Meng, Yang Ming, Wang Mengyun, Hu Zhibin, Shen Hongbing, Jin Guangfu
- 多基因遗传风险评分与胃癌发病年龄的关联分析
- Association between polygenic risk score and age at onset of gastric cancer
- 中华流行病学杂志, 2021, 42(6): 1092-1096
- Chinese Journal of Epidemiology, 2021, 42(6): 1092-1096
- http://dx.doi.org/10.3760/cma.j.cn112338-20201103-01303
-
文章历史
收稿日期: 2020-11-03
2. 山东省肿瘤防治研究院基础研究中心, 济南 250117;
3. 复旦大学附属肿瘤医院, 上海 200032
2. Basic Research Center, Shandong Provincial Cancer Research Institute, Ji'nan 250117, China;
3. Fudan University Shanghai Cancer Center, Shanghai 200032, China
胃癌是全球最常见的恶性肿瘤之一,其发病率和死亡率分别居第五位和第三位[1]。尽管我国胃癌的发病率和死亡率呈稳步下降趋势,但全球仍有40%以上的胃癌发病和死亡病例发生在我国[2],其中早发胃癌发病率和死亡率呈上升趋势[3-4]。既往研究发现,早发胃癌更易受遗传因素影响,胃癌家族史可显著增加胃癌早发风险[5-7]。目前已发现CDH1、CTNNA1等基因遗传突变与早发胃癌密切相关[8-9],已被建议纳入筛查指南[10]。
近年来,国内外多个团队通过全基因组关联研究(genome-wide association study,GWAS)发现并鉴定了10余个胃癌的遗传位点,部分揭示了影响胃癌发生的遗传背景[11]。最近,本团队基于中国人群胃癌GWAS(累计10 254例胃癌病例和10 914例对照),利用112个单核苷酸多态性(SNP)位点构建了中国人群胃癌多基因遗传风险评分(polygenic risk score,PRS),并应用中国慢性病前瞻性研究队列(China Kadoorie Biobank,CKB)的10余万例研究对象评估了PRS与胃癌发病风险的关系,发现PRS可前瞻性预测胃癌发病风险[12],为揭示胃癌的病因及遗传高危人群评估提供了可测量的遗传指标。然而,PRS是否与胃癌发病年龄相关目前尚不清楚。因此,本研究基于已有的GWAS数据,探讨PRS与胃癌发病年龄以及早发风险的关系,为胃癌遗传高危人群病因预防和早期筛查提供科学依据。
对象与方法1.研究对象:本研究纳入前期完成的5项胃癌GWAS(包括East-GWAS、North-GWAS、BJ-GWAS、NJ-GWAS和Onco-GWAS)中经病理明确诊断的8 629例胃癌新发病例[12]。所有胃癌确诊病例均来自医院,其中East-GWAS的4 872例病例来自上海市和江苏省,North-GWAS的1 611例病例来自山东省和河北省,BJ-GWAS的456例来自北京市,NJ-GWAS的550例和Onco-GWAS的1 140例均来自江苏省。纳入本研究的所有研究对象均为无亲属关系的中国汉族人群,且签署知情同意书。本研究已获得南京医科大学伦理委员会伦理审批。
2.研究方法:East-GWAS和North-GWAS样本采用Illumina Infinium® Global Screening Array(GSA)v1.0芯片、BJ-GWAS和NJ-GWAS样本采用Affymetrix Genome-Wide Human SNP Array 6.0芯片、Onco-GWAS样本采用Infinium OncoArray-500K BeadChip芯片进行基因分型。芯片质控与填补的具体流程参见文献[11-12]。前期研究在CKB队列中验证确定了112个SNP位点所构的PRS与胃癌发生风险关联最强,因此,本研究采用这112个SNP位点所构PRS进行后续分析。PRS为每个SNP的风险等位基因的个数乘以其各自的效应值,构建公式: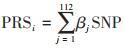
3.统计学分析:根据PRS数值将遗传风险五等分,以≤20%为低遗传风险组、20%~80%为中等遗传风险组、>80%为高遗传风险组。以病例首次确诊为原发胃癌的年龄作为胃癌发病年龄,根据发病年龄将胃癌病例分成五组(< 40、40~、50~、60~、≥70岁)。采用t检验比较两组间差异、方差分析比较多组间差异、Pearson相关性检验分析PRS与胃癌发病年龄的关系。将发病年龄 < 50岁的胃癌病例定义为早发胃癌病例[6, 13],采用Cox比例风险模型、以早发诊断年龄为时间变量估算胃癌早发的遗传风险比(hazard ratios,HR)值及其95%CI,调整性别、地区及前10个主成分。其中主成分基于EIGENSOFT软件的“smartpca”模块对芯片分型数据分析获得,在多因素分析模型中通过校正前10个主成分用于排除潜在人群遗传结构的混杂影响。所有的统计分析均由R 4.0.0软件完成。双侧检验,检验水准α=0.05。
结果1.基本特征:共纳入8 629例胃癌病例,男性6 284例(72.82%),女性2 345例(27.18%);其中6 365例病例有肿瘤部位信息,包括4 586例位于非贲门和1 779例位于贲门。本研究纳入的胃癌病例年龄范围为19~93岁,年龄为(60.61±10.80)岁;女性胃癌病例发病年龄[(58.19±12.33)岁]低于男性[(61.51±10.03)岁],差异有统计学意义(P < 0.001),非贲门癌病例发病年龄[(60.02±10.86)岁]低于贲门癌病例[(63.27±9.18)岁],差异有统计学意义(P < 0.001)。见表 1。
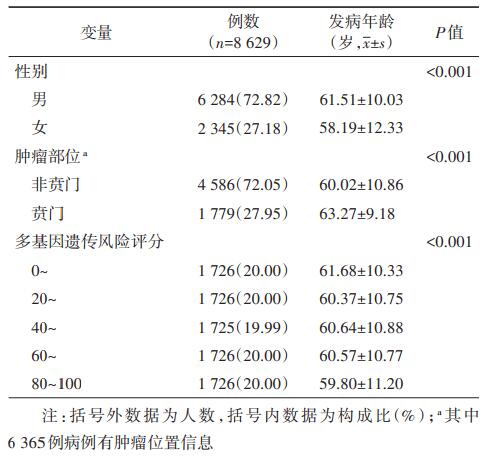
2.PRS与胃癌发病年龄的关联:胃癌病例PRS呈正态分布(图 1A),PRS范围为12.49~20.95,平均值为16.73±1.12,其与胃癌发病年龄呈显著负相关(r=-0.05,P < 0.001),且在贲门癌和非贲门癌中均呈显著负相关(贲门癌:r=-0.06,P=0.015;非贲门癌:r=-0.05,P < 0.001);以PRS数值将胃癌病例五等分后,发现随着PRS的增高,胃癌病例发病呈显著年轻化趋势(P < 0.001),见表 1;以PRS最低20%病例为低遗传风险组,PRS最高20%为高遗传风险组,低、中、高风险组胃癌发病年龄分别为(61.68±10.33)岁、(60.53±10.79)岁、(59.80±11.20)岁(图 1B);随着遗传风险的逐渐增加,低龄发病胃癌病例的比例呈显著上升趋势(P < 0.001,图 1C):低、中、高风险组 < 40岁胃癌病例的比例分别为2.78%、4.13%和5.50%,40~49岁胃癌病例比例分别为9.62%、10.51%和11.82%。

|
| 图 1 多基因遗传风险评分的分布及其与胃癌病例发病年龄的关系 |
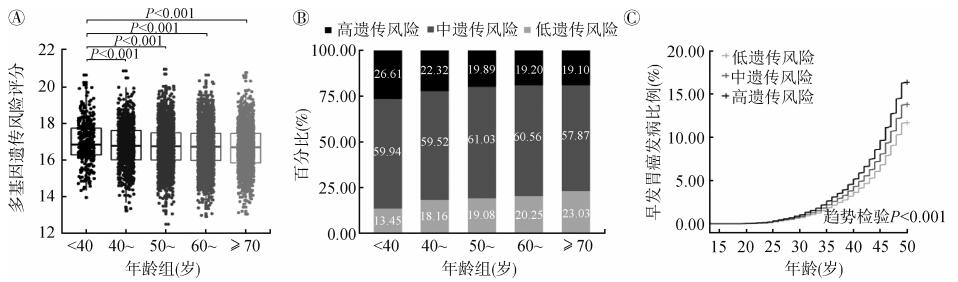
3.PRS与胃癌早发风险的关联:将胃癌病例以发病年龄分组后(< 40、40~、50~、60~、≥70岁)发现,发病年龄越小的病例其PRS越高(分别为:16.96±1.12、16.79±1.14、16.75±1.11、16.71±1.10、16.66±1.14,图 2A),具有高遗传风险病例的比例越高(分别为26.61%、22.32%、19.89%、19.20%、19.10%,图 2B)。将年龄 < 50岁的病例定义为早发胃癌,早发胃癌病例的比例为14.73%,早发非贲门癌和贲门癌的比例分别为15.83%和7.20%(表 2)。早发胃癌的PRS(16.84±1.14)显著高于非早发胃癌(16.71±1.11,P < 0.001);与低遗传风险组相比,中、高遗传风险组的早发胃癌病例比例显著增加(图 2C),多因素Cox回归分析显示,中、高遗传风险组胃癌早发风险分别增加了19%和44%(中风险:HR=1.19,95%CI:1.03~1.39,P=0.022;高风险:HR=1.44,95%CI:1.20~1.71,P < 0.001)。对不同肿瘤部位胃癌分析显示,高遗传风险显著增加非贲门癌早发风险(HR=1.43,95%CI:1.14~1.81,P=0.002),但与贲门癌早发风险不存在显著关联(HR=1.13,95%CI:0.63~2.02,P=0.683)。见表 2。
|
| 图 2 不同发病年龄胃癌病例的遗传风险分布 |
近年来,多项研究利用GWAS分析遗传位点累积效应与疾病早发的关联,发现高PRS可增加包括乳腺癌、前列腺癌、结直肠癌等在内的多种肿瘤早发风险[14-15],但PRS与胃癌发病年龄的关系尚不清楚。本研究基于本课题组前期大样本胃癌GWAS研究,探讨PRS与胃癌发病年龄之间的关联,研究结果显示,PRS越高胃癌发病越早,高PRS可显著增加胃癌早发风险,尤其是非贲门癌早发风险。
由于年轻病例接触环境暴露较少、暴露时间短,遗传因素在早发胃癌中可能发挥更重要的作用[16]。既往研究显示,青壮年人群胃癌发生可能易受到如胃癌家族史[5-7]、5p15 rs10052016的遗传变异[17]、CDH1基因大片段缺失[8]和微卫星不稳定(microsatellite instability,MSI)[18]等遗传改变的影响。本研究发现高PRS可显著增加早发胃癌发病风险,进一步证实了遗传因素在早发胃癌中的重要作用。
本课题组前期的大样本GWAS和前瞻性队列研究发现高PRS显著增加胃癌发生风险[12],本研究发现高PRS导致胃癌发病更早,早发胃癌风险更高。因此,PRS作为多基因遗传因素的可检测指标,不仅可预测胃癌发病风险,同时也是早发胃癌的独立危险因素。既往研究显示,青年人群中胃癌尤其是非贲门癌发病率显著上升[3, 19],12.5%的胃癌病例发病年龄 < 50岁[20],且早发胃癌病例中以女性为主[5],这与本研究结果一致。另外,Bergquist等[20]发现早发胃癌与传统胃癌危险因素如吸烟、饮酒、肥胖等均不相关,这也进一步表明PRS作为可检测指标对识别早发胃癌高危人群的重要价值。
本课题组前期已在大型前瞻性队列中证实遵循健康的生活方式可降低高遗传风险人群胃癌发生风险[12],指导遗传高危人群的病因预防。最近一项多中心研究利用年龄、性别、胃蛋白酶原Ⅰ/Ⅱ比值、胃泌素17浓度、幽门螺杆菌感染、腌制和油炸食品构建胃癌筛查模型[21],用于识别胃癌高风险人群指导内镜筛查,PRS将来也可能作为一项指标纳入筛查模型,指导胃癌甚至早发胃癌的二级预防。
本研究存在局限性。①本研究未能系统采集胃癌临床分期、病理类型等信息,难以分析PRS对不同类型胃癌发病年龄和早发风险的影响;②部分样本缺乏肿瘤部位信息,并且贲门癌的病例数较少,可能导致PRS与贲门癌的关联分析结果不够准确;③本研究PRS构建的遗传位点及其效应来源于胃癌GWAS,可能不能精确反映早发胃癌以及不同亚型胃癌的遗传风险;④本研究PRS全部来源于常见遗传变异构建,未考虑低频或罕见变异对胃癌早发的影响。
综上所述,本研究基于前期大样本胃癌GWAS研究构建的PRS,分析了PRS与胃癌发病年龄之间的关联,发现PRS越高胃癌发病越早,高PRS可显著增加胃癌早发风险,尤其是非贲门癌早发风险。PRS可作为遗传检测指标评估胃癌发病和早发风险,识别高危人群,对胃癌的一级和二级预防具有重要的指导意义。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Bray F, Ferlay J, Soerjomataram I, et al. Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J]. CA Cancer J Clin, 2018, 68(6): 394-424. DOI:10.3322/caac.21492 |
| [2] |
Chen WQ, Zheng RS, Baade PD, et al. Cancer statistics in China, 2015[J]. CA Cancer J Clin, 2016, 66(2): 115-132. DOI:10.3322/caac.21338 |
| [3] |
王少明, 郑荣寿, 张思维, 等. 2015年中国胃癌流行特征分析[J]. 中华流行病学杂志, 2019, 40(12): 1517-1521. Wang SM, Zheng RS, Zhang SW, et al. Epidemiological characteristics of gastric cancer in China, 2015[J]. Chin J Epidemiol, 2019, 40(12): 1517-1521. DOI:10.3760/cma.j.issn.0254-6450.2019.12.003 |
| [4] |
Yin J, Song JN, Bai ZG, et al. Gastric cancer mortality trends in China (2006-2013) reveal increasing mortality in Young Subjects[J]. Anticancer Res, 2017, 37(8): 4671-4679. DOI:10.21873/anticanres.11871 |
| [5] |
Milne AN, Sitarz R, Carvalho R, et al. Early onset gastric cancer: on the road to unraveling gastric carcinogenesis[J]. Curr Mol Med, 2007, 7(1): 15-28. DOI:10.2174/156652407779940503 |
| [6] |
Kwak HW, Choi IJ, Kim CG, et al. Individual having a parent with early-onset gastric cancer may need screening at younger age[J]. World J Gastroenterol, 2015, 21(15): 4592-4598. DOI:10.3748/wjg.v21.i15.4592 |
| [7] |
Shin CM, Kim N, Yang HJ, et al. Stomach cancer risk in gastric cancer relatives: interaction between Helicobacter pylori infection and family history of gastric cancer for the risk of stomach cancer[J]. J Clin Gastroenterol, 2010, 44(2): e34-39. DOI:10.1097/MCG.0b013e3181a159c4 |
| [8] |
Sugimoto S, Yamada H, Takahashi M, et al. Early-onset diffuse gastric cancer associated with a de novo large genomic deletion of CDH1 gene[J]. Gastric Cancer, 2014, 17(4): 745-749. DOI:10.1007/s10120-013-0278-2 |
| [9] |
Clark DF, Michalski ST, Tondon R, et al. Loss-of-function variants in CTNNA1 detected on multigene panel testing in individuals with gastric or breast cancer[J]. Genet Med, 2020, 22(5): 840-846. DOI:10.1038/s41436-020-0753-1 |
| [10] |
Blair VR, McLeod M, Carneiro F, et al. Hereditary diffuse gastric cancer: updated clinical practice guidelines[J]. Lancet Oncol, 2020, 21(8): e386-397. DOI:10.1016/S1470-2045(20)30219-9 |
| [11] |
Yan CW, Zhu M, Ding YB, et al. Meta-analysis of genome-wide association studies and functional assays decipher susceptibility genes for gastric cancer in Chinese populations[J]. Gut, 2020, 69(4): 641-651. DOI:10.1136/gutjnl-2019-318760 |
| [12] |
Jin GF, Lv J, Yang M, et al. Genetic risk, incident gastric cancer, and healthy lifestyle: a Meta-analysis of genome-wide association studies and prospective cohort study[J]. Lancet Oncol, 2020, 21(10): 1378-1386. DOI:10.1016/S1470-2045(20)30460-5 |
| [13] |
Ishikawa M, Hayashi H, Sakamoto N, et al. Cancer gene profiling explores the possible precision medicine for diffuse-type gastric adenocarcinoma[J]. Med Oncol, 2020, 37(1): 10. DOI:10.1007/s12032-019-1327-4 |
| [14] |
Mars N, Koskela JT, Ripatti P, et al. Polygenic and clinical risk scores and their impact on age at onset and prediction of cardiometabolic diseases and common cancers[J]. Nat Med, 2020, 26(4): 549-557. DOI:10.1038/s41591-020-0800-0 |
| [15] |
Archambault AN, Su YR, Jeon J, et al. Cumulative burden of colorectal cancer-associated genetic variants is more strongly associated with early-onset vs late-onset cancer[J]. Gastroenterology, 2020, 158(5): 1274-1286. e12. DOI:10.1053/j.gastro.2019.12.012 |
| [16] |
Skierucha M, Milne AN, Offerhaus GJA, et al. Molecular alterations in gastric cancer with special reference to the early-onset subtype[J]. World J Gastroenterol, 2016, 22(8): 2460-2474. DOI:10.3748/wjg.v22.i8.2460 |
| [17] |
Du JB, Xu YC, Dai JC, et al. Genetic variants at 5p15 are associated with risk and early onset of gastric cancer in Chinese populations[J]. Carcinogenesis, 2013, 34(11): 2539-2542. DOI:10.1093/carcin/bgt259 |
| [18] |
Bacani J, Zwingerman R, Di Nicola N, et al. Tumor microsatellite instability in early onset gastric cancer[J]. J Mol Diagn, 2005, 7(4): 465-477. DOI:10.1016/S1525-1578(10)60577-6 |
| [19] |
Sung H, Siegel RL, Rosenberg PS, et al. Emerging cancer trends among young adults in the USA: analysis of a population-based cancer registry[J]. Lancet Public Health, 2019, 4(3): e137-147. DOI:10.1016/S2468-2667(18)30267-6 |
| [20] |
Bergquist JR, Leiting JL, Habermann EB, et al. Early-onset gastric cancer is a distinct disease with worrisome trends and oncogenic features[J]. Surgery, 2019, 166(4): 547-555. DOI:10.1016/j.surg.2019.04.036 |
| [21] |
Cai QC, Zhu CP, Yuan Y, et al. Development and validation of a prediction rule for estimating gastric cancer risk in the Chinese high-risk population: a nationwide multicentre study[J]. Gut, 2019, 68(9): 1576-1587. DOI:10.1136/gutjnl-2018-317556 |
 2021, Vol. 42
2021, Vol. 42