文章信息
- 庞元捷, 吕筠, 余灿清, 孙点剑一, 李立明.
- Pang Yuanjie, Lyu Jun, Yu Canqing, Sun Dianjianyi, Li Liming
- 多组学在慢性病病因学研究中的应用及其进展
- A multi-omics approach to investigate the etiology of non-communicable diseases: recent advance and applications
- 中华流行病学杂志, 2021, 42(1): 1-9
- Chinese Journal of Epidemiology, 2021, 42(1): 1-9
- http://dx.doi.org/10.3760/cma.j.cn112338-20201201-01370
-
文章历史
收稿日期: 2020-12-01
多组学通常包括在脱氧核糖核酸(DNA)复制、转录、翻译、翻译后修饰的过程中,产生的全部基因(基因组学)、基因表达的广泛变化(表观遗传组学)、核糖核酸(RNA,转录组学)和蛋白质(蛋白质组学),以及下游的小分子代谢产物(代谢组学)。本文将首先介绍单一组学的概念、检测方法、在慢性病研究中的应用及研究进展,再从多组学层面介绍代表性研究与分析方法,以及多组学在慢性病病因学研究中的机遇与挑战,最后介绍多组学研究展望。
如图 1所示,多组学的影响因素包括年龄、社会经济因素、行为生活方式、环境等;统计分析方法主要为多变量模型和生物信息学分析;人群研究为多组学用于慢性病病因学研究的主要手段,其中的阳性结果需要在体内或体外实验中验证。多组学数据整合可以揭示分子间的相互作用网络,整合组学(integromics)的概念应运而生[1]。

|
| 图 1 多组学数据的整合 |
系统流行病学(systems epidemiology)将多组学、传统行为危险因素流行病学与健康环境决定因素、数据计算方法研究与流行病学研究进行整合,以深入了解疾病发生发展的分子途径、网络与交互,揭开暴露与结局之间的“黑箱子”,指导疾病的检测、临床诊断和预后,有助于精准预防和治疗[2]。系统流行病学的核心是整合,包括:①系统内不同性质的构成要素(DNA、信使RNA、蛋白质、生物小分子等);②多细胞生物从基因到细胞、器官、组织、个体的各个层次;③研究思路和方法;④生物学研究、暴露组学研究、临床、人群健康和疾病结局的研究。在系统流行病学领域,多组学数据的综合分析将为慢性病发病机制的拓展、生物标志物的发现和治疗靶标的识别提供证据。
二、基因组学1.基因组学的概念与检测方法:基因组学是对生物体全基因组的研究。基因组中的变异分为单核苷酸变异(simple nucleotide variation,SNV)和结构变异(structural variation,SV)。编码区的SNV和SV可能影响蛋白质序列,而非编码区的SNV和SV可能影响基因表达和剪接过程。一般人群中的频率 > 1%的SNV称为单核苷酸多态性(single nucleotide polymorphism,SNP)[1]。当前测定遗传变异的技术有Sanger测序、DNA微阵列(基因芯片)和二代测序(next generation sequencing,NGS)[3]。全基因组测序(whole genome sequencing,WGS)能够测定罕见变异,它们与侧翼变异(flanking variant)的连锁不平衡较低,对基因功能和表达的影响更大,具有更大的群体特异性,在复杂疾病的遗传中发挥独特作用[4]。全基因组关联分析(genome- wide association study,GWAS)的原理为微阵列测序。由于NGS费用比GWAS高1~2个数量级,GWAS仍广泛用于较大人群的基因型检测。
2.基因组学在慢性病研究中的应用:首先,GWAS和其他无假设的遗传分析方法为功能分析提供靶点,有助于识别未知的致病机制。由于功能丧失(loss-of-function,LoF)变异(如无意义突变、移码和剪接位点变异)有较为确定的功能,常利用LoF变异识别因果变异位点(causal variant)。第二,遗传变异与疾病的关联为药物研发提供新目标。利用遗传学数据预测新药的治疗效果与副作用,在减少费用的同时改善药物研发流程。具有遗传证据支持靶点的候选药物更有可能在Ⅱ和Ⅲ期临床试验中获得成功,前蛋白转化酶枯草溶菌素9(proprotein convertase subtilisin/kexin type 9,PCSK9)与胆固醇酯转移蛋白(cholesterol ester transfer protein,CETP)就是典型的例子[5]。第三,具有高疾病风险的遗传变异对精准医学尤为重要。少数复杂疾病是由某些基因中的单个致病变异引起的,例如遗传性乳腺癌和卵巢癌综合征中的BRCA1和BRCA2。针对变异携带者的预防措施包括双侧乳房切除术、输卵管卵巢切除术、乳腺磁共振成像等[4]。最后,遗传位点在遗传风险评分、孟德尔随机化、基因-环境交互等领域有广泛应用。多基因风险评分(polygenic risk score,PRS)旨在量化多个基因或位点的累积效应,利用数十、数百甚至更多的基因组变异信息构建衡量个体疾病易感性的分数值[6-7]。已在中国人群中开发的PRS有BMI、高血压、糖尿病、非小细胞性肺癌(non-small- cell lung cancer,NSCLC)、胃癌等[8-12]。
3.基因组学研究进展:近三年国际高水平医学期刊发表的近百篇代表性GWAS文章中,12篇纳入亚洲人群,4篇纳入中国人群[13-16];已发表的代表性WGS文章中,仅1篇纳入东亚人群,为日本人群[17];有3篇全外显子测序(whole exome sequencing,WES)文章纳入中国人群[18-20]。2020年Nature发表的东亚人群GWAS研究纳入77 418名2型糖尿病(type 2 diabetes,T2D)患者和356 122名健康对照,共发现与T2D易感性相关的遗传位点301个,其中61个为首次报道[16]。2019年发表的GWAS荟萃分析纳入27 120例NSCLC与27 355例对照(中国人群占50%),发现19个易感位点,包括6个新位点。利用这些位点构建中国人群的PRS,已在独立中国人群中得到验证,为中国慢性病前瞻性研究(China Kadoorie Biobank,CKB)中的95 408名研究对象[11]。2020年发表的中国人群胃癌的GWAS研究,纳入10 254名胃癌患者与10 914名健康对照,并利用112个位点构建PRS,研究基因-环境交互作用与胃癌发病风险。健康生活方式包括不吸烟、不饮酒、较少食用腌制食品、经常摄入新鲜水果和蔬菜。在独立中国人群中(即CKB 100 220名研究对象),PRS与胃癌发病风险显著相关;与采取不健康生活方式且具有高遗传风险的成年人相比,采取健康生活方式的成年人胃癌发病风险降低[12]。研究结果提示,可以利用PRS识别癌症高危人群,进行精准预防。
三、表观遗传组学1.表观遗传组学的概念与检测方法:表观遗传学是一门在不改变DNA序列情况下,研究基因的不同活动状态及其分子与机制的学科。表观遗传学变化包括DNA甲基化水平的变化、组蛋白修饰、染色质重塑(chromatin remodeling)、非编码RNA(non-coding RNA,ncRNA)功能的变化和表观遗传成分的突变。最著名的表观遗传DNA修饰是CG富集序列中胞嘧啶的DNA甲基化(CpG岛)。全基因组DNA甲基化有多种测定方法,例如全基因组亚硫酸氢盐测序、简化代表性亚硫酸氢盐测序、甲基化DNA免疫沉淀和甲基CpG免疫沉淀[21]。组蛋白为小分子的碱性蛋白质,末端可被甲基化、乙酰化、磷酸化和泛素化等,其中乙酰化最常见。组蛋白的翻译后修饰(post-translational modification,PTM)在基因转录的激活和抑制中起着至关重要的作用。染色质重塑是指真核基因组包裹成核小体染色质的过程。染色质免疫沉淀结合高通量测序可用于推断与DNA结合蛋白质的分布,例如具有特定PTM的核心组蛋白成分、转录因子和表观遗传酶[22]。
2.表观遗传组学在慢性病研究中的应用:表观遗传应用于衰老、疾病病因、药物研发等领域。DNA甲基化随年龄而变化,因此被用作反映生物学年龄与衰老的标志物。将机器学习应用于高通量DNA甲基化数据,已构建若干生物学年龄的甲基化指标。结合甲基化数据与测量表型年龄的临床方法,确定一组CpG基因组位点,可以更好地预测寿命和健康寿命。疾病病因方面,Barker最早提出疾病的胎儿起源假设,认为在子宫内胎儿发育的特定敏感时期或童年早期,暴露于环境因素(例如化学物质、药物、压力或感染),成年后易患某些疾病。后来的研究提出表观遗传可能介导其中部分作用。此外,大量体内与体外实验研究证据提示,表观遗传变化可能是癌症发生发展的主要驱动力。近年来,表观遗传组逐渐成为肿瘤治疗研究的新热点。具有临床治疗前景的药物有DNA甲基转移酶和组蛋白去乙酰化酶抑制剂等[23]。
3.表观遗传组学研究进展:近年有两项中国人群代表性的表观遗传学研究[24-25]。2017年发表的中国人群研究在102名急性冠状动脉综合征(acute coronary syndrome,ACS)患者与101名健康对照中测定全血的全基因组甲基化,并在100名ACS患者与102名对照中进行验证。研究发现47个与ACS相关的CpG位点,代表与动脉粥样硬化和炎症信号有关的路径,包括趋化性、冠状动脉血栓形成和T细胞介导的细胞毒性。发现的阳性关联归因于CD8+T细胞、CD4+T细胞和B细胞中CpG位点的差异甲基化。研究结果提示,在ACS中免疫信号和细胞功能可能在表观遗传水平上受到调节[24]。2018年一项研究测定989名中国成年人与160名欧洲成年人的全血甲基化数据,在中国人群中构建并验证了甲基化年龄。这一甲基化年龄为中国人和欧洲人提供准确的预测,甲基化年龄与实际年龄的相关系数为0.94~0.96。研究还发现暴露于多环芳烃可能对正常衰老和表观遗传改变产生不利影响[25]。
四、转录组学1.转录组学的概念与检测方法:转录组是细胞中基因转录所得产物RNA的集合,由占1%~4%的编码RNA(即信使RNA,mRNA)和占 > 95%的ncRNA组成。mRNA的数量相对固定,ncRNA的数量随生物体的进化逐渐增加[26]。RNA检测技术及其优缺点与DNA相似,主要有微阵列和测序技术(RNA sequencing,RNA-seq)[1]。传统的微阵列和大规模RNA-seq涉及细胞集合群,测定的为不同种类细胞基因表达的平均水平。单细胞RNA测序(single-cell RNA sequencing,scRNA-seq)技术对给定样本中每个细胞的转录组进行高分辨率和深度分析,可无偏差地评估细胞异质性,以超高分辨率和准确性阐明发育和分化过程中的动态细胞转变。scRNA-seq技术对心血管和癌症等研究领域产生了深远影响[27-28]。
2.转录组学在慢性病研究中的应用:
mRNA分析的应用包括测定转录子是否存在与进行定量分析;评价差异剪接以评估或预测蛋白异构体;使用表达数量性状位点(expression quantitative trait loci,eQTL,指控制mRNA表达水平的位点)或等位基因特异性表达(allele specific expression,ASE)定量评估基因型对基因表达的影响。这些信息对于了解细胞和组织代谢的动力学,对于了解转录组变化及其如何影响健康和疾病至关重要[1]。ncRNA存在于多种生物样本中,其测定简易且无侵袭性,可作为生物标志物用于疾病诊断、风险评估、治疗方法选择和治疗效果监测[26]。全转录组关联研究(transcriptome-wide association study,TWAS)对GWAS筛选出的潜在致病位点进行优先级排序,即筛选出可能与疾病具有因果关联的基因位点。TWAS利用带有基因型数据(通常为GWAS)与表达信息的eQTL队列,分析基因与表型的因果关联。TWAS使用表达面板(expression panel)中基因邻近区域遗传变异的等位基因计数,来预测GWAS队列中每个个体的基因表达,进一步分析预测基因表达与性状之间的统计关联[29]。
3.转录组学研究进展:近三年国际高水平医学期刊发表的代表性转录组学研究主要在欧美人群开展,在生物标志物筛选、风险预测、疾病预后、致病突变的识别等方面发挥重要作用。全基因组癌症分析(Pan-Cancer Analysis of the Whole Genomes consortium,PCAWG)是迄今为止最大的国际合作联盟,对38类肿瘤的2 658个全基因组进行测序。在以往研究检测癌症基因组蛋白编码区的工作基础上,该项目探索了编码区和非编码区中体细胞和种系变异,特别强调顺式调控位点、ncRNA和基因组SV。2020年初Nature发表了一系列文章,介绍了PCAWG关于驱动突变(cancer drivers)、非编码DNA遗传驱动因子(non-coding changes)、突变标签(mutational signatures,即独特性DNA序列或单核苷酸位点)、SV、肿瘤进化(cancer evolution)和转录组的最新发现[30]。美国国立卫生研究院于2010年启动基因型-组织表达项目(Genotype-Tissue Expression portal,GTEx),旨在识别和绘制eQTL以研究基因组遗传变异与不同组织内基因表达的关联。GTEx提供人类基因表达、eQTL、sQTL(splice quantitative trait loci,剪接数量性状位点)和ASE数据,以验证不同组织中的基因表达和基因表达调控模式[31]。
五、蛋白质组学1.蛋白质组学的概念与检测方法:蛋白质组是在生物体、系统或生物学环境中产生的蛋白质。在人体全部组织和器官中,能够被表达和加工为蛋白质的基因约有2万个。人类蛋白质组计划已收集的质谱(mass spectrometry,MS)数据约涵盖这2万种蛋白质的90%。转录翻译后的蛋白质通常会经历翻译后加工,可能产生7万余种蛋白质[32]。MS是蛋白质组学中最常用的技术,其他常见方法有基于免疫或亲和力(affinity)的检测方法与蛋白质测序法。MS或免疫测定法共检测到约5 000种循环蛋白,约占人类蛋白质组的25%[33]。MS中有两种互补的肽段测量方法:靶向MS使用稳定同位素标记肽作为标准,对样品中的肽进行绝对定量;非靶向MS利用肽离子强度进行蛋白质组的半定量测定[32]。
2.蛋白质组学在慢性病研究中的应用:蛋白质组学检测技术的发展为使用GWAS检测pQTL(protein quantitative trait loci,蛋白数量性状位点,指影响蛋白水平的位点)进行循环蛋白遗传调控研究铺平了道路,已报道数百个SNP与蛋白质水平存在关联。与蛋白质水平相关的遗传变异分两类:顺式pQTL(cis-pQTL)为靠近编码蛋白质的基因;反式pQTL(trans-pQTL)距离编码蛋白质的基因更远,通常位于不同染色体上。当前发现的蛋白质中有18%~25%具有至少一个cis-pQTL。如果cis-pQTL主要通过影响mRNA表达或周转起作用,则在相关组织或细胞类型中可能发现eQTL[33]。pQTL在生物医学和制药领域有广泛应用。pQTL为疾病通路上的中间表型,用于解释疾病GWAS发现的遗传位点。此外,pQTL为疾病致病基因提供线索,有助于发现临床生物标志物、发现现有药物新的适应症、评价开发中药物的潜在安全性,有助于蛋白质-蛋白质相互作用网络的研究[32]。
3.蛋白质组学研究进展:近三年发表于高水平期刊的代表性研究主要使用Olink、Somalogic与MS平台测定蛋白质组,测定100~1 000余种蛋白质,开展心血管代谢疾病的机制研究与药物研发,研究对象以欧洲人群为主。2019年一项发表于Nature的中国研究在110对病例与健康对照中,分析HBV感染所致早期肝细胞癌的肿瘤与非肿瘤样本的蛋白质组特征[34]。2020年CKB项目发表的最新研究探讨中国人群肥胖与蛋白质组学以及蛋白质组学与心血管疾病(cardiovascular disease,CVD)的关联。该研究纳入628名研究对象,发现BMI与27种蛋白质呈正相关,与3种蛋白质呈负相关。进一步孟德尔随机化分析发现,BMI与特定蛋白质的因果关联与观察性关联在方向上一致。在30种与BMI相关的蛋白质中,有10种(包括白介素6、白介素18、肝细胞生长因子)与CVD发病风险相关。研究结果提示,特定蛋白质可能介导肥胖与CVD之间的关联[35]。
六、代谢组学1.代谢组学的概念与检测方法:代谢组学是对机体中小分子成分的系统研究,通常涉及数百至数千种代谢物的测量。代谢标志物既代表基因组的下游输出,又代表环境的上游输入。因此,对代谢物和代谢组的研究能够探索基因与环境的相互作用[36]。代谢组常用的检测技术有核磁共振(nuclear magnetic resonance,NMR)技术、气相色谱质谱法(gas chromatography-mass spectrometry,GC- MS)和液相色谱质谱法MS(liquid chromatography- mass spectrometry,LC-MS),涵盖许多种类的有机化合物,包括脂质、氨基酸、糖、生物胺和有机酸等[36]。非靶向代谢组为对所有可测量分析物的半定量测定,是一种检测特定条件下代谢物水平变化的无偏方法。靶向代谢组通过化学定量和生化注释法,鉴定与特定表型相关或疾病通路上的关键代谢物,常应用于实验环境或大规模人群研究中[37]。
2.代谢组学在慢性病研究中的应用;与蛋白质和基因相比,代谢产物分子量较小、结构简单且容易获得。代谢组主要应用于如下领域:诊断先天性疾病;发现中间代谢的早期改变;鉴定既往未知的代谢产物;开发生物标志物,比如与行为生活方式相关和多种慢性病的诊断与治疗的标志物;预测疾病发病与死亡风险;定义干预研究的替代终点。
3.代谢组学研究进展:近三年发表于高水平期刊的代表性研究使用靶向与非靶向代谢组检测平台,测定数百种代谢产物,研究代谢组与慢性病的关联、生物标志物开发、以及药物对生物标志物的影响。2018年CKB项目发表的代谢组研究,利用NMR对4 660名研究对象血浆样本中225种代谢产物进行测定。研究发现脂蛋白和脂质与心肌梗死和缺血性脑卒中的关联相似,但与出血性脑卒中无关。在HDL-C颗粒中,胆固醇与心肌梗死呈负相关,而TG与心肌梗死呈正相关。乙酰基糖蛋白和几种非脂质代谢产物(如酮体、葡萄糖和二十二碳六烯酸)与上述3种疾病均相关[38]。在同一人群中,体力活动与100余个代谢产物相关,提示代谢产物可能解释体力活动与CVD风险的关联[39]。
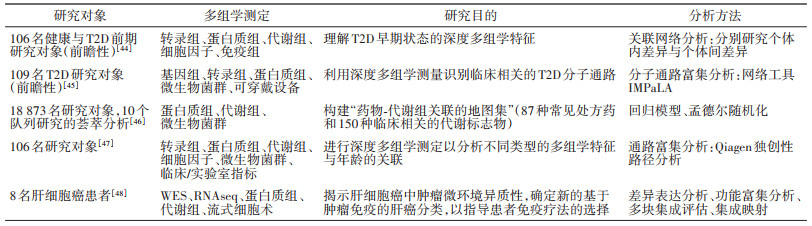
七、多组学1.多组学代表性研究及分析方法:单一组学层面的研究缺乏多水平、多层面的整合,对复杂疾病病因推断的价值有限。多组学因而被越来越多应用于慢性病病因学研究。近三年高水平期刊发表的代表性多组学文章有数十篇,部分代表性文章见表 1。研究对象多为西方人群,中国人群开展的相关研究较少。目前常见的多组学分析方法包括:①数据归一化和降维方法,例如主成分分析,该方法将数据分解为几个变量,以识别能最好解释表型差异的变量;②其他多变量分析方法,例如典型相关分析(canonical correlation analysis),用于分析变量集之间的整体相关性,并最终确定最能反映特定生物学状况的因素;③其他积分学框架法包括偏最小二乘(partial least square)回归分析或多因素分析,能够确定表型差异的主要来源[40-41];④生物信息学将多分子水平的实验数据、临床信息与计算模型相结合,将系统作为一个整体进行处理,应用于诊断、预后或治疗[42-43]。通路富集分析(pathway enrichment analysis)为多组学常用分析方法,该方法对待检验的基因数量、相对排序、注释基因路径中的基因数量进行了统计检验,以判断相应路径是否过度表达[42-43]。
2.多组学对慢性病流行病学研究的意义和价值:主要体现在因果推断、中介分析与风险预测三方面。第一,多组学从宏观与微观病因学层面进行因果推断,全面系统探索环境与遗传因素在慢性病病因学中的作用,揭示复杂疾病的致病因素与分子机制。第二,多组学有助于探索环境及行为生活方式与慢性病关联的中介通路,有望揭示复杂疾病的机制。第三,利用多组学标志物开发风险预测模型应用于精准医学,用于疾病风险预测以识别高危人群,用于筛选药物治疗对象使之受益最大化,用于监测药物疗效与不良反应。
3.多组学研究为大规模队列研究带来的机遇与挑战:首先,多组学研究拓展了病因学研究的深度。慢性病病因学研究从传统的危险因素分析、生物标志物识别、疾病风险模型开发,到借助多组学检测方法的微观病因学研究,应用系统流行病学的研究方法与思路,对疾病的分子机制进行探索[2]。第二,随着国际公开数据、分析平台与协作组的指数级增加,研究资源将更加丰富,研究成本也将大幅下降。生物银行为多组学研究的宝贵资源,然而其长期随访的维持与实验室检测费用十分可观。临床上病理样本获得难度较大,为某些罕见疾病的分子研究带来挑战。第三,由于多种因素综合作用和单个数据集的高变异性可能导致错误发现,这使得解读多组学分析结果,尤其是识别生物学相关的分子存在困难。第四,多组学带来跨国合作、跨学科合作的机会,对包括中国在内的中低收入国家研究尤为重要,尤其在研究设计与检测技术方面。然而,研究涉及的伦理与数据共享问题值得重点关注。
4.中国在多组学病因学研究的优势与问题:中国在多组学病因学研究的独特优势体现在不同于西方人群的疾病谱、行为生活方式与遗传背景方面。利用多组学研究探索疾病的发病机制在不同人种是否存在差异,研究特定危险因素与疾病关联的机制。中国人群出血性脑卒中比例较高,出血性与缺血性脑卒中的危险因素存在差异(如血脂);在特定BMI水平,中国人群中心性肥胖比例较高,中心性肥胖为心血管代谢疾病的重要危险因素;中国人有独特的生活方式,如食辣食与饮茶等,两者均为CVD的保护因素[49]。此外,中国人群的遗传背景不同于西方人群,特定等位基因频率不同导致关联研究统计学功效不同。中国人群还存在独特的LoF变异[50]。这些遗传变异为下一步的功能学研究、工具变量的选择和PRS的构建提供重要指导。
现有中国的多组学研究中,涉及3种及以上组学的检测通常是以医院为基础的临床组织样本研究(如肿瘤样本),规模相对较小,纳入研究对象通常为100人以下。由于基因组检测技术高通量、低成本的特点,GWAS已广泛应用于大规模队列。然而,代谢组与蛋白质组等检测技术尚未达到高通量、低成本。因此,大规模队列测定的组学种类相对较少,通常为基因组与另一种组学(如代谢组、蛋白质组)。
八、展望多组学研究慢性病病因将向着横向与纵向两方面发展。横向方面,国际上多组学协作组的成立拓展了样本量,纳入来自不同国家的人群,使得验证早期研究发现的阳性结果成为可能。2020年一项最新GWAS研究纳入3万余名欧洲成年人,报道了85种蛋白的451个pQTL[51]。该研究来自SCALLOP协作组,纳入20个利用Olink平台测定蛋白质组的队列。SCALLOP的目的是研究主要慢性病的蛋白质标志物及其影响因素,为蛋白质组用于精准医学研究提供了资源[52]。代谢组研究联盟(the Consortium of Metabolomics Studies,COMETS)为一项国际大规模协作研究,成立于2014年,旨在分析代谢组及其与疾病病因、诊断和预后的关系。COMETS包括来自亚洲、欧洲、北美和南美的47个队列,纳入136.5万名研究对象,包括1985-2017年收集血液样本的代谢组数据。代谢组数据由17个不同的平台测定[53]。考虑到数据整合需求日益增多,COMETS的发展为队列研究在代谢组平台选择上提供指导。由于以往协作组纳入的人群以欧洲人群为主,研究者在解读以往研究结果、进行研究设计时,应充分考虑中国人群行为生活方式、疾病谱、遗传与生物标志物水平的差异,以及研究结果在中国人群中的验证。此外,考虑到测定平台存在的差异与不同研究结果之间的可比性,在研究设计阶段选择测定平台尤为重要。
纵向方面,经典的多组学定义正在向全组学拓展,包括生命历程的多组学动态变化、微生物菌群(microbiome)、基于医疗大数据的表型组和临床影像学检查的放射组(radiomics)。研究者应根据研究问题与经费,决定多组学所检测组学的种类,选择研究样本(血液、组织等)与研究设计,比如是进行大规模单次检测的横断面或前瞻性研究,还是进行小规模多次检测的纵向研究。在研究方法上将基础-预防-临床相结合,多组学发现的阳性标志物正逐渐在细胞和动物实验中得到验证。例如,基于前瞻性队列研究发现的支链氨基酸与胰腺癌风险的关联,已在由突变Kras表达驱动的早期胰腺癌小鼠模型中得到验证,提示蛋白质分解增加是胰腺癌发展中的早期事件[54]。然而,已报道的胰腺癌风险预测模型尚未评价支链氨基酸的作用。这提示多学科合作在多组学病因研究方面的重要性。
综上所述,多组学为传统的观察性流行病学研究进行慢性病病因推断提供新思路,为系统流行病学整合探索疾病机制提供宝贵资源,为后续进一步的实验性验证研究提供重要参考。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Manzoni C, Kia DA, Vandrovcova J, et al. Genome, transcriptome and proteome:the rise of omics data and their integration in biomedical sciences[J]. Brief Bioinform, 2018, 19(2): 286-302. DOI:10.1093/bib/bbw114 |
| [2] |
黄涛, 李立明. 系统流行病学[J]. 中华流行病学杂志, 2018, 39(5): 694-699. Huang T, Li LM. Systems epidemiology[J]. Chin J Epidemiol, 2018, 39(5): 694-699. DOI:10.3760/cma.j.issn.0254-6450.2018.05.031 |
| [3] |
Prokop JW, May T, Strong K, et al. Genome sequencing in the clinic:the past, present, and future of genomic medicine[J]. Physiol Genomics, 2018, 50(8): 563-579. DOI:10.1152/physiolgenomics.00046.2018 |
| [4] |
Momozawa Y, Mizukami K. Unique roles of rare variants in the genetics of complex diseases in humans[J]. J Hum Genet, 2020. DOI:10.1038/s10038-020-00845-2 |
| [5] |
King EA, Davis JW, Degner JF. Are drug targets with genetic support twice as likely to be approved? Revised estimates of the impact of genetic support for drug mechanisms on the probability of drug approval[J]. PLoS Genet, 2019, 15(12): e1008489. DOI:10.1371/journal.pgen.1008489 |
| [6] |
杭栋, 沈红兵. 多基因风险评分与复杂性疾病风险预测和精准预防:机遇和挑战[J]. 中华流行病学杂志, 2019, 40(9): 1027-1030. Hang D, Shen HB. Application of polygenic risk scores in risk prediction and precision prevention of complex diseases:opportunities and challenges[J]. Chin J Epidemiol, 2019, 40(9): 1027-1030. DOI:10.3760/cma.j.issn.0254-6450.2019.09.001 |
| [7] |
林丽娟, 魏永越, 张汝阳, 等. 孟德尔随机化方法在观察性研究因果推断中的应用[J]. 中华预防医学杂志, 2019, 53(6): 619-624. Lin LJ, Wei YY, Zhang R, et al. Application of mendelian randomization methods in causal inference of observational study[J]. Chin J Prev Med, 2019, 53(6): 619-624. DOI:10.3760/cma.j.issn.0253-9624.2019.06.015 |
| [8] |
Pang Y, Kartsonaki C, Lv J, et al. Observational and genetic associations of body mass index and hepatobiliary diseases in a relatively lean Chinese population[J]. JAMA Netw Open, 2020, 3(10): e2018721. DOI:10.1001/jamanetworkopen.2020.18721 |
| [9] |
Lu X, Huang J, Wang L, et al. Genetic predisposition to higher blood pressure increases risk of incident hypertension and cardiovascular diseases in Chinese[J]. Hypertension, 2015, 66(4): 786-792. DOI:10.1161/HYPERTENSIONAHA.115.05961 |
| [10] |
Gan W, Bragg F, Walters RG, et al. Genetic predisposition to type 2 diabetes and risk of subclinical atherosclerosis and cardiovascular diseases among 160, 000 Chinese adults[J]. Diabetes, 2019, 68(11): 2155-2164. DOI:10.2337/db19-0224 |
| [11] |
Dai J, Lv J, Zhu M, et al. Identification of risk loci and a polygenic risk score for lung cancer:a large-scale prospective cohort study in Chinese populations[J]. Lancet Respir Med, 2019, 7(10): 881-891. DOI:10.1016/S2213-2600(19)30144-4 |
| [12] |
Jin G, Lv J, Yang M, et al. Genetic risk, incident gastric cancer, and healthy lifestyle:a meta-analysis of genome-wide association studies and prospective cohort study[J]. Lancet Oncol, 2020, 21(10): 1378-1386. DOI:10.1016/S1470-2045(20)30460-5 |
| [13] |
Tin A, Marten J, Halperin Kuhns VL, et al. Target genes, variants, tissues and transcriptional pathways influencing human serum urate levels[J]. Nat Genet, 2019, 51(10): 1459-1474. DOI:10.1038/s41588-019-0504-x |
| [14] |
Wuttke M, Li Y, Li M, et al. A catalog of genetic loci associated with kidney function from analyses of a million individuals[J]. Nat Genet, 2019, 51(6): 957-972. DOI:10.1038/s41588-019-0407-x |
| [15] |
Lin GW, Xu C, Chen K, et al. Genetic risk of extranodal natural killer T-cell lymphoma:a genome-wide association study in multiple populations[J]. Lancet Oncol, 2020, 21(2): 306-316. DOI:10.1016/S1470-2045(19)30799-5 |
| [16] |
Spracklen CN, Horikoshi M, Kim YJ, et al. Identification of type 2 diabetes loci in 433, 540 East Asian individuals[J]. Nature, 2020, 582(7811): 240-245. DOI:10.1038/s41586-020-2263-3 |
| [17] |
Hirata J, Hosomichi K, Sakaue S, et al. Genetic and phenotypic landscape of the major histocompatibilty complex region in the Japanese population[J]. Nat Genet, 2019, 51(3): 470-480. DOI:10.1038/s41588-018-0336-0 |
| [18] |
Sun Y, Chen Y, Li Y, et al. Association of TSR1 variants and spontaneous coronary artery dissection[J]. J Am Coll Cardiol, 2019, 74(2): 167-176. DOI:10.1016/j.jacc.2019.04.062 |
| [19] |
Chang J, Zhong R, Tian J, et al. Exome-wide analyses identify low-frequency variant in CYP26B1 and additional coding variants associated with esophageal squamous cell carcinoma[J]. Nat Genet, 2018, 50(3): 338-343. DOI:10.1038/s41588-018-0045-8 |
| [20] |
Chen J, Yang H, Teo ASM, et al. Genomic landscape of lung adenocarcinoma in East Asians[J]. Nat Genet, 2020, 52(2): 177-186. DOI:10.1038/s41588-019-0569-6 |
| [21] |
Cavalli G, Heard E. Advances in epigenetics link genetics to the environment and disease[J]. Nature, 2019, 571(7766): 489-499. DOI:10.1038/s41586-019-1411-0 |
| [22] |
Zhang W, Song M, Qu J, et al. Epigenetic modifications in cardiovascular aging and diseases[J]. Circ Res, 2018, 123(7): 773-786. DOI:10.1161/CIRCRESAHA.118.312497 |
| [23] |
Salameh Y, Bejaoui Y, El Hajj N. DNA methylation biomarkers in aging and age-related diseases[J]. Front Genet, 2020, 11: 171. DOI:10.3389/fgene.2020.00171 |
| [24] |
Li J, Zhu X, Yu K, et al. Genome-wide analysis of DNA methylation and acute coronary syndrome[J]. Circ Res, 2017, 120(11): 1754-1767. DOI:10.3892/ijmm.2017.3220 |
| [25] |
Li J, Zhu X, Yu K, et al. Exposure to polycyclic aromatic hydrocarbons and accelerated DNA methylation aging[J]. Environ Health Perspect, 2018, 126(6): 067005. DOI:10.1289/EHP2773 |
| [26] |
de Gonzalo-Calvo D, Vea A, Bar C, et al. Circulating non-coding RNAs in biomarker-guided cardiovascular therapy:a novel tool for personalized medicine?[J]. Eur Heart J, 2019, 40(20): 1643-1650. DOI:10.1093/eurheartj/ehy234 |
| [27] |
Kashima Y, Sakamoto Y, Kaneko K, et al. Single-cell sequencing techniques from individual to multiomics analyses[J]. Exp Mol Med, 2020, 52(9): 1419-1427. DOI:10.1038/s12276-020-00499-2 |
| [28] |
Paik DT, Cho S, Tian L, et al. Single-cell RNA sequencing in cardiovascular development, disease and medicine[J]. Nat Rev Cardiol, 2020, 17(8): 457-473. DOI:10.1038/s41569-020-0359-y |
| [29] |
Wainberg M, Sinnott-Armstrong N, Mancuso N, et al. Opportunities and challenges for transcriptome-wide association studies[J]. Nat Genet, 2019, 51(4): 592-599. DOI:10.1038/s41588-019-0385-z |
| [30] |
Alexandrov LB, Kim J, Haradhvala NJ, et al. The repertoire of mutational signatures in human cancer[J]. Nature, 2020, 578(7793): 94-101. DOI:10.1038/s41586-020-1943-3 |
| [31] |
Consortium GT. The GTEx Consortium atlas of genetic regulatory effects across human tissues[J]. Science, 2020, 369(6509): 1318-1330. DOI:10.1126/science.aaz1776 |
| [32] |
Suhre K, McCarthy MI, Schwenk JM. Genetics meets proteomics:perspectives for large population-based studies[J]. Nat Rev Genet, 2020. DOI:10.1038/s41576-020-0268-2 |
| [33] |
Schubert OT, Rost HL, Collins BC, et al. Quantitative proteomics:challenges and opportunities in basic and applied research[J]. Nat Protoc, 2017, 12(7): 1289-1294. DOI:10.1038/nprot.2017.040 |
| [34] |
Jiang Y, Sun A, Zhao Y, et al. Proteomics identifies new therapeutic targets of early-stage hepatocellular carcinoma[J]. Nature, 2019, 567(7747): 257-261. DOI:10.1038/s41586-019-0987-8 |
| [35] |
Pang Y, Kartsonaki C, Lv J, et al. Adiposity, circulating protein biomarkers and risk of major vascular diseases[J]. JAMA Cardiol, 2020, Dec 2: e206041. DOI:10.1001/jamacardio.2020.6041 |
| [36] |
Wishart DS. Emerging applications of metabolomics in drug discovery and precision medicine[J]. Nat Rev Drug Discov, 2016, 15(7): 473-484. DOI:10.1038/nrd.2016.32 |
| [37] |
Muller MJ, Bosy-Westphal A. From a "Metabolomics fashion" to a sound application of metabolomics in research on human nutrition[J]. Eur J Clin Nutr, 2020. DOI:10.1038/s41430-020-00781-6 |
| [38] |
Holmes MV, Millwood IY, Kartsonaki C, et al. Lipids, lipoproteins, and metabolites and risk of myocardial infarction and stroke[J]. J Am Coll Cardiol, 2018, 71(6): 620-632. DOI:10.1016/j.jacc.2017.12.006 |
| [39] |
Pang Y, Kartsonaki C, Du H, et al. Physical activity, sedentary leisure time, circulating metabolic markers, and risk of major vascular diseases[J]. Circ Genom Precis Med, 2019, 12(9): 386-396. DOI:10.1161/CIRCGEN.118.002527 |
| [40] |
de Tayrac M, Le S, Aubry M, et al. Simultaneous analysis of distinct Omics data sets with integration of biological knowledge:Multiple Factor Analysis approach[J]. BMC Genomics, 2009, 10: 32. DOI:10.1186/1471-2164-10-32 |
| [41] |
Cisek K, Krochmal M, Klein J, et al. The application of multi-omics and systems biology to identify therapeutic targets in chronic kidney disease[J]. Nephrol Dial Transplant, 2016, 31(12): 2003-2011. DOI:10.1093/ndt/gfv364 |
| [42] |
Reimand J, Isserlin R, Voisin V, et al. Pathway enrichment analysis and visualization of omics data using g:Profiler, GSEA, Cytoscape and Enrichment Map[J]. Nat Protoc, 2019, 14(2): 482-517. DOI:10.1038/s41596-018-0103-9 |
| [43] |
Paczkowska M, Barenboim J, Sintupisut N, et al. Integrative pathway enrichment analysis of multivariate omics data[J]. Nat Commun, 2020, 11(1): 735. DOI:10.1038/s41467-019-13983-9 |
| [44] |
Zhou W, Sailani MR, Contrepois K, et al. Longitudinal multi-omics of host-microbe dynamics in prediabetes[J]. Nature, 2019, 569(7758): 663-671. DOI:10.1038/s41586-019-1236-x |
| [45] |
Schussler-Fiorenza Rose SM, Contrepois K, Moneghetti KJ, et al. A longitudinal big data approach for precision health[J]. Nat Med, 2019, 25(5): 792-804. DOI:10.1038/s41591-019-0414-6 |
| [46] |
Liu J, Lahousse L, Nivard MG, et al. Integration of epidemiologic, pharmacologic, genetic and gut microbiome data in a drug-metabolite atlas[J]. Nat Med, 2020, 26(1): 110-117. DOI:10.1038/s41591-019-0722-x |
| [47] |
Ahadi S, Zhou W, Schussler-Fiorenza Rose SM, et al. Personal aging markers and ageotypes revealed by deep longitudinal profiling[J]. Nat Med, 2020, 26(1): 83-90. DOI:10.1038/s41591-019-0719-5 |
| [48] |
Zhang Q, Lou Y, Yang J, et al. Integrated multiomic analysis reveals comprehensive tumour heterogeneity and novel immunophenotypic classification in hepatocellular carcinomas[J]. Gut, 2019, 68(11): 2019-2031. DOI:10.1136/gutjnl-2019-318912 |
| [49] |
Pang YJ, Lyu J, Yu CQ, et al. Risk factors for cardiovascular disease in the Chinese population:recent progress and implications[J]. Global Health Journal, 2020, 4(3): 65-71. DOI:10.1016/j.glohj.2020.08.004 |
| [50] |
Millwood IY, Bennett DA, Holmes MV, et al. Association of CETP gene variants with risk for vascular and nonvascular diseases among Chinese adults[J]. JAMA Cardiol, 2018, 3(1): 34-43. DOI:10.1001/jamacavdio.2017.4177 |
| [51] |
Folkersen L, Gustafsson S, Wang Q, et al. Genomic and drug target evaluation of 90 cardiovascular proteins in 30, 931 individuals[J]. Nat Metab, 2020, 2(10): 1135-1148. DOI:10.1038/s42255-020-00287-2 |
| [52] |
The SCALLOP Consortium[EB/OL].[2020-11-20].https://www.olink.com/scallop/.
|
| [53] |
Yu B, Zanetti KA, Temprosa M, et al. The Consortium of Metabolomics Studies (COMETS):metabolomics in 47 prospective cohort studies[J]. Am J Epidemiol, 2019, 188(6): 991-1012. DOI:10.1093/aje/kwz028 |
| [54] |
Mayers JR, Wu C, Clish CB, et al. Elevation of circulating branched-chain amino acids is an early event in human pancreatic adenocarcinoma development[J]. Nat Med, 2014, 20(10): 1193-1198. DOI:10.1038/nm.3686 |
 2021, Vol. 42
2021, Vol. 42