文章信息
- 谢彤, 吕莉琨, 谭昭麟, 李力, 吕杰, 李晓燕.
- Xie Tong, Lyu Likun, Tan Zhaolin, Li Li, Lyu Jie, Li Xiaoyan
- 中国天津市一例缅甸输入基孔肯雅热病例的病毒基因分型
- Genotyping on one case with Chikungunya infection introduced into Tianjin in China from Myanmar
- 中华流行病学杂志, 2020, 41(12): 2131-2134
- Chinese Journal of Epidemiology, 2020, 41(12): 2131-2134
- http://dx.doi.org/10.3760/cma.j.cn112338-20200131-00062
-
文章历史
收稿日期: 2020-01-31
基孔肯雅热是由于被伊蚊叮咬而感染基孔肯雅病毒(CHIKV)所引起的急性发热性传染病。感染者在急性期主要临床表现为发热、头痛、肌肉痛、多发性关节炎等症状,约25%的感染者中会出现持续6个月以上慢性关节炎[1]。CHIKV于1953年在坦桑尼亚首次被分离出来,为有包膜的,正链RNA病毒,属于披膜病毒科甲病毒属。CHIKV的长度约为11.8 kb,其读码框架分为两个部分,包括4个非结构蛋白编码区nsP1~nsP4和5个结构蛋白C-E3- E2-6K-E1编码区。
病毒进化研究表明CHIKV主要被分为3个基因型,包括西非型(West African)、亚洲型(Asian)和东/中/南非型(East/Central/South African,ECSA),从ECSA基因型毒株中又分化出印度洋(India Ocean lineage,IOL)亚型[2]。基孔肯雅热被认为是再现性传染性疾病,在过去15年中全球范围内至少有两次基孔肯雅热大规模流行。2004年由ECSA基因型CHIKV引发的基孔肯雅热疫情在非洲地区肯尼亚暴发,至2006年传播至南亚、东南亚地区,引发约600万人感染的大流行[3]。2007年基孔肯雅热通过输入性病例传入意大利并扩散至欧洲地区其他国家,导致基孔肯雅热首次在欧洲地区当地人群中暴发流行[4]。第二次大规模流行是2013年亚洲基因型CHIKV传入加勒比海岛屿和美洲地区,在近50个美洲国家中传播流行[5]。2019年11月天津市检出1例输入性基孔肯雅热病例,本研究对患者感染的CHIKV开展基因分型研究。
材料与方法1.样本采集:血标本采自从事导游工作、自缅甸回国的中国天津市本地居民。患者入境后开始发热(39 ℃),并伴有膝关节、肘关节疼痛、结膜充血等症状。在境外停留8 d期间有被蚊虫叮咬史。患者血清中登革病毒IgG抗体、IgM抗体和抗-NS1抗体检测阴性,登革病毒核酸检测亦为阴性。
2.病毒RNA提取:采用天隆科技RNA/DNA病毒核酸提取试剂盒(磁珠法)及其配套的核酸提取仪(型号:NP968-C)提取患者血清中的RNA,操作按照说明书进行。吸取血清的量为200 μl,病毒RNA溶于80 μl洗脱液中,-40 ℃储存。
3. CHIKV检测:采用上海之江CHIKV核酸测定试剂盒,每个反应体系中加入5 μl提取的RNA。实时荧光定量PCR反应在ABI公司7500 fast荧光定量PCR仪上进行,反应条件根据试剂盒操作说明进行设定。
4. CHIKV基因组E1编码区的扩增:病毒RNA反转录采用天根生物FastKing第一链合成试剂盒。向上述提取的8 μl病毒RNA中加入2 μl含有gDNase的缓冲液(试剂盒提供),通过42 ℃,3 min去除基因组DNA;去除基因组DNA后,加入2 μl 10×缓冲液、1 μl FastKing反转录酶、2 μl随机六聚引物混合物,补充去离子水5 μl使反转录的反应体系至20 μl;42 ℃,孵育15 min,合成第一链互补DNA。以第一链互补DNA为模板,使用两对引物分别扩增CHIKV E1编码区的不同区域[6]。50 μl PCR反应体系中包括25 μl Q5 2×Master Mix Taq酶(NEB Biolabs公司)、正反向引物(10 μmol/L)各2.5 μl、5 μl第一链互补DNA和15 μl去离子水。扩增条件为94 ℃ 5 min;94 ℃ 30 s,54 ℃ 60 s,72 ℃ 30 s,35个循环;72 ℃ 10 min。PCR反应完成后,经1.5%琼脂糖电泳,以确定扩增产物的长度是否符合预期。
5. CHIKV E1序列测定:PCR扩增产物的测序工作委托英骏生物技术有限公司完成。每个扩增片段使用正反向引物双向测序以保证测序结果的准确性。
6. CHIKV系统发生树分析:采用DNAStar 7.1软件包中EditSeq对测序结果进行复核、编辑。从GenBank获得CHIKV不同基因型序列以构建系统发生树,比较不同病毒株之间的同源性。用于构建系统发生树毒株的GenBank编号,流行的国家或地区。所有的序列经Clustal W软件进行排列比对,使用MEGA 10分析软件,通过邻接法(neighbor-joining,NJ)构建系统发生树,并用Bootstraping检验系统发生树的可信度,Bootstrap值设置为1 000。
结果1. CHIKV核酸检测:检测患者血清标本提取RNA经实时定量PCR检测,其Ct值为22.8,小于试剂盒操作说明设定Ct≤38的阈值,且反应曲线呈“S”形,因此判定为CHIKV核酸检测阳性,患者为CHIKV感染。
2. RT-PCR扩增CHIKV E1基因:扩增E1基因的两对引物分别对应从南美地区分离的CHIKV毒株(GenBank序列编号:MH359142)的10 209~10 554和10 356~10 950碱基,其扩增产物分别为346 bp和595 bp,琼脂糖电泳中扩增产物长度与预期长度相符(图 1)。
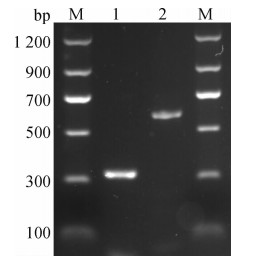
|
| 注:M:分子量标记Marker Ⅱ(天根生化);1:346 bp;2:595 bp 图 1 基孔肯雅病毒E1基因2个片段经RT-PCR反应后扩增产物凝胶电泳结果 |
3. CHIKV系统发生树构建与基因分型:为揭示天津市输入的CHIKV与其他地区流行毒株的关系,从GenBank中下载了33株在不同时期一些地区流行的不同基因型CHIKV序列,与本研究的测序结果一同构建系统发生树。结果表明,天津市输入型CHIKV基因分型属于能够感染白纹伊蚊并在其体内繁殖的ECSA基因型印度洋亚型(ECSA-IOL)(图 2)。系统发生树分析表明,此次输入的CHIKV与2016-2017年在巴基斯坦、意大利、孟加拉国等国家流行的CHIKV毒株高度同源,序列的同源性高达99.4%,并与这些国家流行的CHIKV毒株共同组成1个基因分型簇。
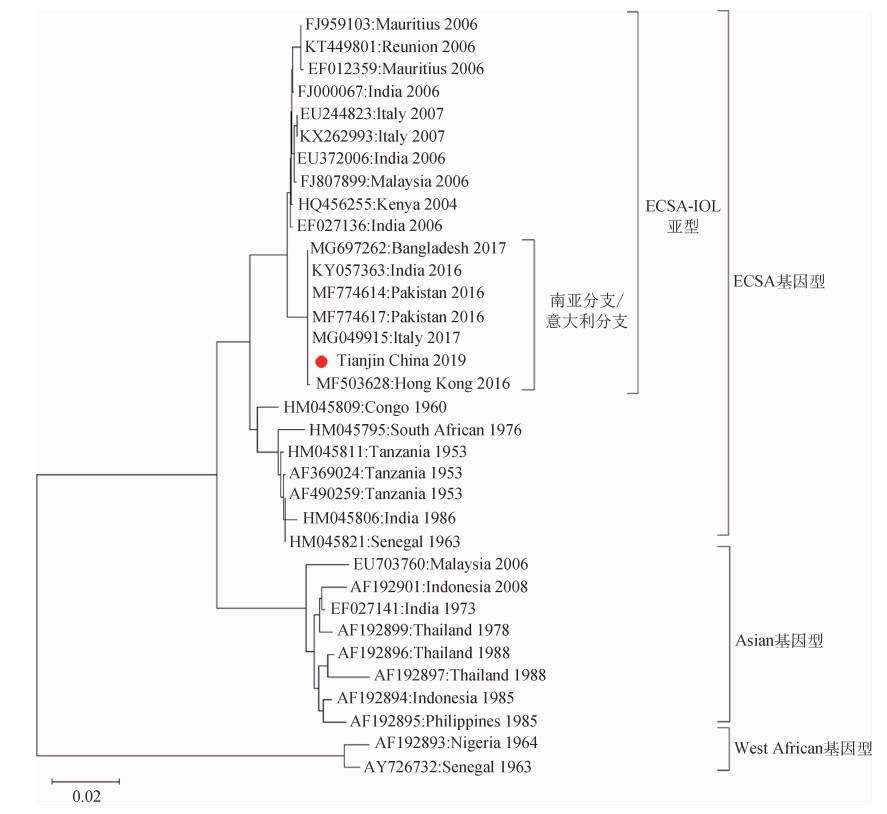
|
| 图 2 基于天津市输入型基孔肯雅病毒E1序列的系统发生树分析 |
近年来我国广东、浙江等省份均发现基孔肯雅热输入性病例[7],在广东省东莞市暴发了我国首起基孔肯雅热社区聚集性疫情[8]。我国报道基孔肯雅热病例主要发生在南方省份,而本次天津市出现的基孔肯雅热说明随着我国到境外疫区旅行人数逐年增长,北方地区同样会出现输入性病例,应加强针对入境人员的监测,防止疫情在本地人群中出现。
既往研究表明CHIKV发生适应性突变可引发基孔肯雅热疫情在全球的暴发流行。基孔肯雅热在非洲传播的蚊媒主要为埃及伊蚊,而导致2004年在南亚、东南亚地区大流行的ECSA型CHIKV能够在白纹伊蚊体内生存繁殖,特别是在病毒基因组编码的E1基因中存在A226V突变的毒株[9]。对CHIKV E1基因序列分析证实本次天津市输入性基孔肯雅热的毒株属于ECSA基因型IOL亚型。通过系统发生树构建显示此次输入型CHIKV与近期引发多个国家和地区基孔肯雅热流行的毒株高度同源,与这些国家和地区分离的毒株同属一个基因分型簇。该分型簇的毒株起源于印度,随后传播至巴基斯坦(2016)[10]、意大利(2017)[11]、孟加拉国(2017)[12]和伊朗(2019)[13]并引发基孔肯雅热在当地暴发流行,此分型簇的毒株还通过访问印度的旅行者输入至中国香港地区(2018)[14]和澳大利亚[15]。值得关注的是CHIKV在意大利只能以白纹伊蚊为媒介传播,说明该分型簇中的毒株在白纹伊蚊体内具有较强的生存和繁殖能力。这一基因分型簇中的毒株被认为是IOL亚型毒株经适应性突变进化而形成的一个新的分支,不同研究将其命名为巴基斯坦/意大利分支[11]或南亚分支[12]。本研究发现,该入境人员感染的CHIKV属于这一分支,尚属国内第一次发现该分支的毒株输入。IOL亚型中南亚分支CHIKV毒株广泛传播的机制尚未阐明,对其病原学特征的初步研究发现这一分支的CHIKV毒株在E1基因并未发生A226V突变,但该分支毒株的nsP2、nsP4等编码区的一些位点与其他毒株相比发生了特异性突变,这些突变对增强CHIKV传播能力的机制还需要进一步研究[11]。
对基孔肯雅热全球流行的监测中发现从ECSA基因型IOL亚型毒株中最新进化出来的南亚分支在短时间内造成全球多个国家基孔肯雅热暴发流行和病例输入,提示该分支的CHIKV毒株具有较强的传播能力。除本次输入病例外,2019年多个国家报告的基孔肯雅热输入病例均来自缅甸[16]。虽然未见有关基孔肯雅热在缅甸的分子流行病学研究,但本次从缅甸输入的毒株属于南亚分支,提示这一分支CHIKV已经传播至缅甸,并在当地人群中流行。缅甸与我国相邻且人员交往频繁,同时白纹伊蚊在我国大部分地区均有分布,因此本次输入病例为我国可能出现的基孔肯雅热本地传播疫情提供了预警。此外,对输入的基孔肯雅热病例开展分子流行病学研究可以及时掌握病毒的基因特征和变异情况,为研究可能出现的本地传播病例的起源、传播链追踪以及传播动力学提供数据支持。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Pathak H, Mohan MC, Ravindran V. Chikungunya arthritis[J]. Clin Med (Lond), 2019, 19(5): 381-385. DOI:10.7861/clinmed.2019-0035 |
| [2] |
Schneider ADB, Ochsenreiter R, Hostager R, et al. Updated phylogeny of chikungunya virus suggests lineage-specific RNA architecture[J]. Viruses, 2019, 11(9): 798. DOI:10.3390/v11090798 |
| [3] |
Silva LA, Dermody TS. Chikungunya virus:epidemiology, replication, disease mechanisms, and prospective intervention strategies[J]. J Clin Invest, 2017, 127(3): 737-749. DOI:10.1172/JCI84417 |
| [4] |
Rezza G, Nicoletti L, Angelini R, et al. Infection with chikungunya virus in Italy:anoutbreak in a temperate region[J]. Lancet, 2007, 370(9602): 1840-1846. DOI:10.1016/S0140-6736(07)61779-6 |
| [5] |
Leparc-Goffart I, Nougairede A, Cassadou S, et al. Chikungunya in the Americas[J]. Lancet, 2014, 383(9916): 514. DOI:10.1016/S0140-6736(14)60185-9 |
| [6] |
Cardoso FD, de Rezende IM, Barros ELT, et al. Circulation of Chikungunya virus East-Central-South Africa genotype during an outbreak in 2016-17 in Piaui State, Northeast Brazil[J]. Rev Inst Med Trop Sao Paulo, 2019, 61(10): e57. DOI:10.1590/S1678-9946201961057 |
| [7] |
Wang YL, Wang X, Liu XB, et al. Epidemiology of imported infectious diseases, China, 2005-2016[J]. Emerg Infect Dis, 2019, 25(1): 33-41. DOI:10.3201/eid2501.180178 |
| [8] |
Zhang QL, He JF, Wu D, et al. Maiden outbreak of chikungunya in Dongguan city, Guangdong province, China:epidemiological characteristics[J]. PLoS One, 2012, 7(8): e42830. DOI:10.1371/journal.pone.0042830 |
| [9] |
Schuffenecker I, Iteman I, Michault A, et al. Genome microevolution of chikungunya viruses causing the Indian Ocean outbreak[J]. PLoS Med, 2006, 3(7): e263. DOI:10.1371/journal.pmed.0030263 |
| [10] |
Shi JM, Su ZY, Fan ZJ, et al. Extensive evolution analysis of the global chikungunya virus strains revealed the origination of CHIKV epidemics in Pakistan in 2016[J]. Virol Sin, 2017, 32(6): 520-532. DOI:10.1007/s12250-017-4077-5 |
| [11] |
Lindh E, Argentini C, Remoli ME, et al. The Italian 2017 Outbreak chikungunya virus belongs to an emerging Aedes albopictus-adapted virus cluster introduced from the Indian subcontinent[J]. Open Forum Infect Dis, 2019, 6(1): ofy321. DOI:10.1093/ofid/ofy32 |
| [12] |
Melan A, Aung MS, Khanam F, et al. Molecular characterization of chikungunya virus causing the 2017 outbreak in Dhaka, Bangladesh[J]. New Microbes New Infect, 2018, 24(7): 14-16. DOI:10.1016/j.nmni.2018.03.007 |
| [13] |
Pouriayevali MH, Rezaei F, Jalali T, et al. Imported cases of Chikungunya virus in Iran[J]. BMC Infect Dis, 2019, 19: 1004. DOI:10.1186/s12879-019-4637-4 |
| [14] |
Ho DTW, Chan DPC, Lam CY, et al. At the advancing front of Chikungunya fever in Asia:Two imported cases in Hong Kong with novel amino acid changes[J]. J Microbiol Immunol Infect, 2018, 51(3): 419-421. DOI:10.1016/j.jmii.2017.08.006 |
| [15] |
Huang BX, Pyke AT, McMahon J, et al. Complete coding sequence of a case of chikungunya virus imported into Australia[J]. Genome Announc, 2017, 5(19): e00310-17. DOI:10.1128/genomeA.00310-17 |
| [16] |
Díaz-Menéndez M, Esteban ET, Ujiie M, et al. Travel-associated chikungunya acquired in Myanmar in 2019[J]. Euro Surveill, 2020, 25(1): 190072. DOI:10.2807/1560-7917.ES.2020.25.1.1900721 |
 2020, Vol. 41
2020, Vol. 41


