文章信息
- 苏永强, 郭丽民, 葛亚俊, 席进孝, 王宇萌, 苗克军, 吴斌, 徐大琴.
- Su Yongqiang, Guo Limin, Ge Yajun, Xi Jinxiao, Wang Yumeng, Miao Kejun, Wu Bin, Xu Daqin
- 甘肃省鼠疫耶尔森菌规律成簇间隔短回文重复序列位点多态性分析及地区分布
- Analysis on clustered regularly interspaced short palindromic repeats loci polymorphism of Yersinia pestis and its area distribution in Gansu province
- 中华流行病学杂志, 2020, 41(12): 2125-2130
- Chinese Journal of Epidemiology, 2020, 41(12): 2125-2130
- http://dx.doi.org/10.3760/cma.j.cn112338-20200107-00017
-
文章历史
收稿日期: 2020-01-07
2. 上海市(复旦大学附属)公共卫生临床中心 201058;
3. 中国疾病预防控制中心传染病预防控制所, 北京 102206
2. Shanghai Public Health Clinical Center, Fudan University, Shanghai 201058, China;
3. National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
甘肃省存在青藏高原喜马拉雅旱獭和甘宁黄土高原阿拉善黄鼠2种类型鼠疫自然疫源地。依据地理景观可继续分为阿尔金山、大雪山、祁连山北麓东段区、甘南高原旱獭鼠疫疫源地和陇中黄土高原阿拉善黄鼠鼠疫疫源地。甘肃省鼠疫疫源地分离的菌株分为阿尔金型、青藏型、祁连型和甘宁黄鼠型。不同生态型的鼠疫耶尔森菌(鼠疫菌)均有一块各自相对独立的疫源地分布区,而且各疫源地动物间鼠疫流行强度的差异,使得菌株在适应环境的变化过程中遗传特征发生变化,这种变异被保存下来,造成不同疫源地间菌株在基因上的差异。分析鼠疫菌基因组的方法主要有多位点串联重复序列分析、差异区段分析、单核苷酸多态性分析、规律成簇的间隔短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)等方法。CRISPR是由一段同向重复序列(direct repeat sequences,DR)和将其分隔开的间区序列(spacers)构成特殊结构的重复序列,广泛分布于原核生物基因组中。CRISPR位点可作为细菌分型和进化分析的理想分子靶标。鼠疫菌基因组中存在3个CRISPR位点,Cui等[1]、葛亚俊等[2]基于CRISPR分型方法将甘肃省30株鼠疫菌分成2大类群,4个CRISPR基因型。本研究利用CRISPR方法对甘肃省203株鼠疫菌进行多态性分析及地区分布研究,为甘肃省鼠疫防控提供技术依据。
对象与方法1.菌株来源及DNA制备:选取1962-2014年甘肃省203株鼠疫菌,其中甘宁黄鼠型菌株9株、青藏型菌株129株、祁连型菌株47株、阿尔金型菌株18株。菌株保藏于甘肃省CDC鼠疫防制科。鼠疫菌DNA提取参照文献[3]进行,置-20 ℃保存。
2. CRISPR位点PCR扩增及测序:3个CRISPR位点引物设计参照文献[1, 4]。PCR反应体系:Premix Ex Taq 25 μl,10 μmol/L正反向引物各1 μl,去离子水21 μl,模板DNA 2 μl。扩增条件:95 ℃预变性5 min;95 ℃变性40 s,58 ℃退火40 s,72 ℃延伸40 s,30个循环;72 ℃延伸5 min。PCR扩增产物经由1.5%琼脂糖凝胶电泳鉴定后,由赛默飞生物技术有限公司进行序列测定。
3. CRISPR位点序列分析:将测序结果通过在线工具CRISPR Finder(http://crispr.u-psud.fr/crispr)搜索并鉴定CRISPR DR-spacer。按照DR和spacers序列库确定每一个CRISPR位点的间区序列种类和排列情况,采用Cui等[1]提出的方法命名,并将每株菌CRISPR位点的排列模式整理在Excel表格中。应用BioNumerics 5.10软件进行聚类分析。甘肃省1:25万矢量化县(区)地图由中国CDC提供。采用ArcGIS 10.3软件进行鼠疫菌CRISPR位点地区分布特征分析。
结果1. CRISPR间区序列聚类分析:203株鼠疫菌分成4个大群,分别为Cb2群、Ca7和Ca7′群、Ca35′群和CaΔ5′群。Cb2群只有甘宁黄鼠菌株6株和1株祁连型菌株。Ca7和Ca7′只有一个位点的差别,聚类分析时成为一个大群。Ca7和Ca7′群、Ca35′群和CaΔ5′群的菌株数量多,生态型复杂。Ca7′群菌株均为青藏型,而Ca7群含有青藏型(40株)、阿尔金型(12株)、祁连型(1株)和甘宁黄鼠型(1株)。Ca35′群含有青藏型(58株)、阿尔金型(6株)和甘宁黄鼠型(1株)。CaΔ5′群含有祁连型(45株)、青藏型(25株)和甘宁黄鼠型(1株)。见图 1。

|
| 图 1 203株鼠疫菌间隔短回文重复序列间区序列聚类图 |
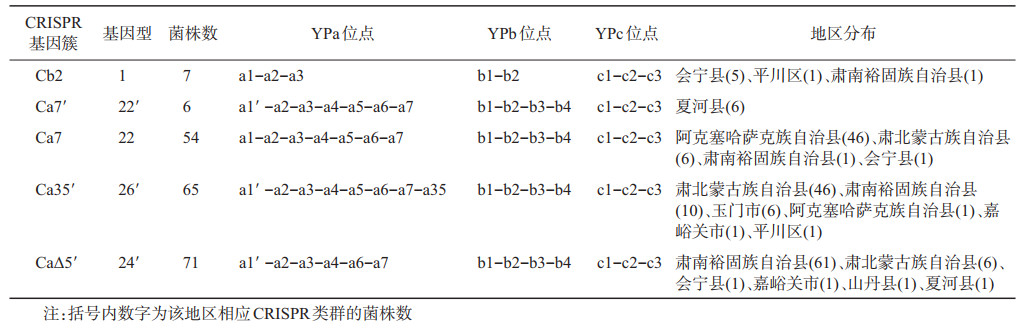
2. CRISPR间区序列种类:甘肃省203株鼠疫菌中共有16种间区序列,排列成5种基因簇(Cb2、Ca7、Ca7′、CaΔ5′、Ca35′)。其中YPa位点有9种,分别是a1、a1′、a2、a3、a4、a5、a6、a7、a35,新的间区序列a1′与鼠疫菌CRISPR位点中已知的a1有极高的同源相似性,a1′与a1在5′→3′方向的第27位存在1个单核苷酸碱基变异(a1′为T,a1为C);YPb位点有4种,分别是b1、b2、b3、b4;YPc位点有3种,分别是c1、c2、c3。各间区序列信息,见表 1。
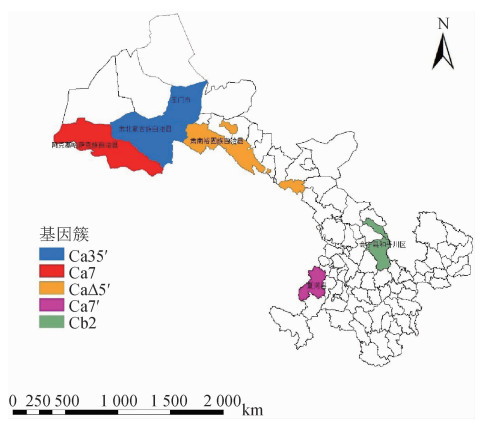
3. CRISPR基因簇及地区分布:203株鼠疫菌被分成5个基因簇,其中Cb2为阿拉善黄鼠疫源地的主要基因簇(66.7%,6/9),集中在会宁县、平川区;Ca7′为甘南高原疫源地的主要基因簇(85.7%,6/7),主要分布在夏河县;Ca7为阿尔金山疫源地的主要基因簇(97.9%,46/47),集中在阿克塞哈萨克族自治县;Ca35′为大雪山疫源地的主要基因簇(79.3%,46/58),主要分布在肃北蒙古族自治县、玉门市;CaΔ5′为祁连山北麓东段区疫源地的主要基因簇(81.3%,61/75),主要分布在肃南裕固族自治县。见表 1和图 2。
|
| 注:甘肃省1:25万矢量化县(区)地图由中国CDC提供 图 2 甘肃省鼠疫菌间隔短回文重复序列基因簇的地区分布 |
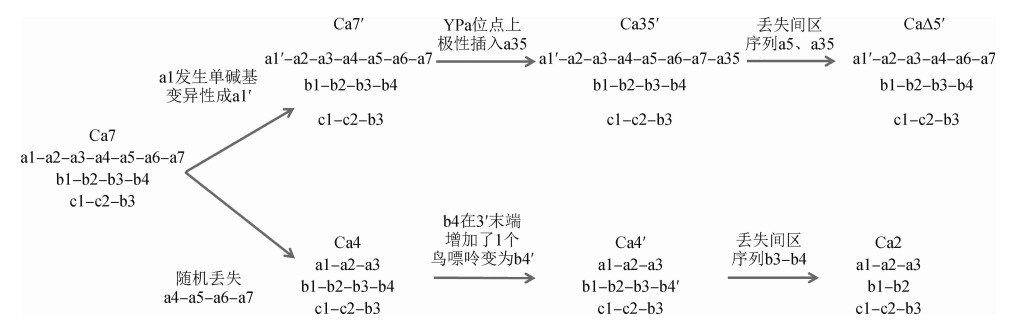
4.甘肃省鼠疫菌进化途径:Ca7类群菌株最为古老,在Ca7间区序列组合的基础上,首先在YPa位点的间区序列,形成Ca7′类群,然后在YPa位点上极性插入a35,形成Ca35′。Ca35′类群菌株适应环境变化,随机丢失间区序列a5、a35,形成a1′- a2-a3-a4-a6-a7间区序列组合,形成CaΔ5′类群。在Ca7间区序列组合的基础上,至此Ca7′、Ca35′、CaΔ5′成为阿尔金山-祁连山北麓东段区疫源地的优势CRISPR类群。Cb2类群菌株可能的形成过程:Ca7类群菌株在YPa位点上随机丢失a4-a5-a6-a7,形成Cb4类群,Cb4类群菌株为进一步适应环境变化,使得其YPb位点的间区序列b4在核酸序列的3′末端增加了1个鸟嘌呤,从而形成Cb4′类群。Cb4′类群在YPb上位点丢失间区序列b3-b4形成Cb2类群,由此Cb2类群适应当地的生态环境成为阿拉善黄鼠疫疫源地主要CRISPR类群。见图 3。
|
| 图 3 甘肃省鼠疫菌进化途径 |
本研究对甘肃省203株鼠疫菌CRISPR特征进行分析,共发现5个CRISPR基因簇,其中2个基因簇与前期报道相同[1],Ca7′、CaΔ5′、Ca35′为新发现的基因簇。阿拉善黄鼠疫源地的CRISPR基因簇为Cb2,基因型为1型。Ca7′为甘南高原疫源地的主要基因簇,基因型为22′型,与此疫源地接壤的青海省同仁县喜马拉雅旱獭鼠疫疫源地CRISPR类群相同[5]。阿尔金山-祁连山北麓东段区鼠疫疫源地存在Ca7、Ca35′、CaΔ5′3种主要类群,此块疫源地地理分布上从西向东分别属于阿尔金山疫源地(阿克塞哈萨克族自治县)、大雪山疫源地(肃北蒙古族自治县和玉门市)和祁连山北麓东段区(肃南裕固族自治县),CRISPR能够从地理分布区分鼠疫菌株。阿克塞哈萨克族自治县菌株主要为Ca7,肃北蒙古族自治县主要是Ca35′,玉门市主要为Ca35′,肃南裕固族自治县主要为CaΔ5′。这3种类群占所试菌株总数>95%,是阿尔金山-祁连山流行最广泛的菌群,而且与此疫源地接壤的青海省鼠疫疫源地(德令哈市、天峻县、祁连县和门源县)CRISPR主要类群一致[5]。
203株鼠疫菌的CRISPR间区序列聚类分析显示,阿拉善黄鼠疫源地的6株甘宁黄鼠型鼠疫菌和1株祁连型鼠疫菌聚在一起成为Cb2群,Ca7和Ca7′只有一个位点的差别,聚类分析时成为一个大群。Ca7′群菌株均为青藏型,而Ca7中以青藏型和阿尔金型菌株最多,Ca35′中青藏型菌株数量最多,CaΔ5′群中祁连型和青藏型菌株最多,CRISPR类群与菌株的生态型较好吻合。研究发现Ca7为阿克塞哈萨克族自治县菌株的主要类群,群中还含有肃北蒙古族自治县和肃南裕固族自治县菌株;Ca35′为肃北蒙古族自治县菌株的主要类群,群中含有肃南裕固族自治县和阿克塞哈萨克族自治县菌株;CaΔ5′为肃南裕固族自治县菌株的主要类群,群中含有肃北蒙古族自治县菌株。这种群内菌株交叉存在的结果,与前期实验室采用DFR基因分型的结果一致[6]。不同群内菌株的交叉存在,表明阿尔金山-祁连山北麓东段区疫源地鼠疫菌基因组之间存在交流,菌株在自然选择压力作用下适应不同生态景观成为某地区的主要基因类群。主要基因类群和次要基因类群的共同存在证实了该地区鼠疫在动物间持续流行的态势。
根据CRISPR位点spacer的随机丢失和极性化添加的规律,以及鼠疫菌CRISPR可能的进化模式,推测甘肃省鼠疫菌可能由相同的祖先菌进化而来[7-8]。前期研究表明Ca7是青藏高原和东祁连山鼠疫疫源地的主要种群[9],阿尔金山-祁连山和甘南高原鼠疫疫源地均为青藏高原鼠疫疫源地的一部分,而且本次实验证实甘南高原疫源地主要类群也是Ca7′,阿尔金山-祁连山疫源地主要存在Ca7、Ca35′、CaΔ5′ 3种类群,此块疫源地西部与东昆仑山旱獭疫源地发现的Ca7类群相一致[10],提示该地区的鼠疫菌可能是由东昆仑山旱獭疫源地传入。鼠疫菌株为适应环境变化进一步进化形成Ca35′、CaΔ5′类群。阿拉善黄鼠疫源地主要类群为Cb2,但是也存在阿尔金山-祁连山疫源地的种群Ca7、CaΔ5′、Ca35′菌株,表明Ca7类群菌株适应环境变化逐渐演变成Cb2类群。通过与其他疫源地菌株CRISPR基因类群相比较,阿尔金山-祁连山旱獭疫源地发现阿拉善黄鼠疫源地的种群Cb2,阿拉善黄鼠疫源地发现阿尔金山-祁连山疫源地的种群Ca7、CaΔ5′、Ca35′菌株,不同类群菌株的出现表明鼠疫菌可能经历了跨疫源地的远距离传播。鼠疫菌的远距离传播,促进了各疫源地菌株之间基因交流的频率,增加了疫源地鼠疫菌株基因组的多样性,给疫源地内鼠疫菌株基因分型和溯源分析带来困难,提示以后在人间疫情与鼠间疫情的溯源分析上要更加重视。甘肃省动物间疫情持续活跃,近几年人间鼠疫发生频率高。从近年的鼠疫分子流行病特征来看,肃北蒙古族自治县、阿克塞哈萨克族自治县等人间疫情以CaΔ5′、Ca35′、Ca7为主,人间疫情病例发病迅速,一般都是在当地进行疫情处置,未造成疫情扩散到其他省市,但是不排除鼠疫远距离传播的风险。阿尔金山-祁连山北麓东段区疫源地紧靠交通干线,更容易导致鼠疫远距离传播,需要加强对该地区鼠疫远距离传播的防控和鼠疫菌基因变异分析。
总之,CRISPR能够区分甘肃省不同疫源地来源的菌株,不同生态型的菌株存在较好的聚集性,能够较好地阐明疫源地之间的联系,演示了甘肃省鼠疫菌株可能进化路线,可为鼠疫监测和菌株溯源分析提供依据。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Cui YJ, Li YJ, Gorgé O, et al. Insight into microevolution of Yersinia pestis by clustered regularly interspaced short palindromic repeats[J]. PLoS One, 2008, 3(7): e2652. DOI:10.1371/journal.pone.0002652 |
| [2] |
葛亚俊, 格鹏飞, 席进孝, 等. 规律成簇的间隔短回文重复序列对甘肃省鼠疫菌株的基因分型研究[J]. 中华地方病杂志, 2017, 36(6): 404-407. Ge YJ, Ge PF, Xi JX, et al. Genotyping of Yersinia pestis isolated from Gansu province by clustered regularly interspaced short palindromic repeats[J]. Chin J Endemiol, 2017, 36(6): 404-407. DOI:10.3760/cma.j.issn.2095-4255.2017.06.004 |
| [3] |
戴二黑.鼠疫耶尔森氏菌基因分型与适应性微进化研究[D].北京: 中国人民解放军军事医学科学院, 2005. Dai EH. Genotyping and adaptive microevolutionary analysis of Yersinia pestis[D]. Beijing: Academy of Military Medical Science People's Liberation Army of China, 2005. |
| [4] |
Pourcel C, Salvignol G, Vergnaud G. CRISPR elements in Yersinia pestis acquire new repeats by preferential uptake of bacteriophage DNA, and provide additional tools for evolutionary studies[J]. Microbiology, 2005, 51(Pt 3): 653-663. DOI:10.1099/mic.0.27437-0 |
| [5] |
徐小青, 辛有全, 李翔, 等. 1954-2011年青海高原鼠疫耶尔森菌CRISPR基因分型及地区分布[J]. 中华预防医学杂志, 2017, 51(3): 237-242. Xu XQ, Xin YQ, Li X, et al. Genotyping by CRISPR and regional distribution of Yersinia pestis in Qinghai-plateau from 1954 to 2011[J]. Chin J Prev Med, 2017, 51(3): 237-242. DOI:10.3760/cma.j.issn.0253-9624.2017.03.009 |
| [6] |
王新华, 张宏, 郭丽民, 等. 甘肃省202株鼠疫耶尔森菌基因型分布及流行特征[J]. 中华流行病学杂志, 2013, 34(5): 433-437. Wang XH, Zhang H, Guo LM, et al. Analysis on genotype distributions and epidemiological characteristics of Yersinia pestis plague foci in Gansu province[J]. Chin J Epidemiol, 2013, 34(5): 433-437. DOI:10.3760/cmaj.issn.0254-6450.2014.02.005 |
| [7] |
杨光璨, 石丽媛, 郭英, 等. 云南省鼠疫耶尔森菌规律成簇间隔短回文重复序列分型[J]. 中华流行病学杂志, 2014, 35(8): 974. Yang GC, Shi LY, Guo Y, et al. Genotyping on Yersinia pestis isolated from Yunnan province by clustered-regularly-interspaced-short palindromic-repeats[J]. Chin J Epidemiol, 2014, 35(8): 974. DOI:10.3760/cma.j.issn.0254-6450.2014.08.022 |
| [8] |
祁腾, 梁莹, 李伟, 等. 四川省鼠疫耶尔森菌CRISPR基因分型及地区分布研究[J]. 中国人兽共患病学报, 2018, 34(9): 801-804, 810. Qi T, Liang Y, Li W, et al. CRISPR genotyping and regional distribution of Yersinia pestis in Sichuan Province, China[J]. Chin J Zoonoses, 2018, 34(9): 801-804, 810. DOI:10.3969/j.issn.1002-2694.2018.00.168 |
| [9] |
崔玉军.鼠疫耶尔森氏菌基因组多态性数据库及鉴定溯源系统的研究[D].北京: 中国人民解放军军事医学科学院, 2008. Cui YJ. Development of genomic polymorphism database and source-tracking system for Yersinia pestis[D]. Beijing: Academy of Military Medical Science People's Liberation Army of China, 2008. |
| [10] |
贺金荣, 张渝疆, 王宇萌, 等. 新疆地区58株鼠疫耶尔森菌规律聚集的间隔短回文重复位点多态性分析[J]. 中国媒介生物学及控制杂志, 2017, 28(3): 233-237. He JR, Zhang YJ, Wang YM, et al. Polymorphism analysis of clustered regularly interspaced short palindromic repeat (CRISPR) loci of 58 Yersinia pestis strains isolated from Xinjiang, China[J]. Chin J Vector Biol Control, 2017, 28(3): 233-237. DOI:10.11853/j.issn.1003.8280.2017.03.009 |
 2020, Vol. 41
2020, Vol. 41