文章信息
- 沈玲羽, 姜博, 严寒秋, 李伟红, 高志勇, 王全意.
- Shen Lingyu, Jiang Bo, Yan Hanqiu, Li Weihong, Gao Zhiyong, Wang Quanyi
- 诺如病毒基因分型研究进展
- Progress on genotyping of norovirus
- 中华流行病学杂志, 2020, 41(11): 1927-1932
- Chinese Journal of Epidemiology, 2020, 41(11): 1927-1932
- http://dx.doi.org/10.3760/cma.j.cn112338-20200302-00234
-
文章历史
收稿日期: 2020-03-02
2. 首都医科大学公共卫生学院, 北京 100069
2. School of Public Health, Capital Medical University, Beijing 100069, China
诺如病毒属杯状病毒科(Caliciviridae)诺如病毒属[1],宿主广泛,可感染人类、犬科动物、啮齿动物、家畜(猪、羊、牛)、海狮及蝙蝠等[2]。诺如病毒是引起人类非细菌性急性胃肠炎和食源性急性胃肠炎暴发的主要病原体之一,是成年人散发腹泻的第一大病原和≤5岁婴幼儿腹泻的第二大病原[3-7],各月份均有散发病例,冬春季节高发,全世界约20%的急性胃肠炎病例由诺如病毒引起[5, 8-10]。
诺如病毒是单股正链RNA病毒,极易变异,包括多个基因组及多种基因型[11]。如何准确鉴定诺如病毒基因型,对疫情溯源、分子流行病学监测和疫苗设计至关重要。研究早期主要基于衣壳蛋白编码区氨基酸或核苷酸序列的差异对诺如病毒分型[12]。随着在全球范围内的广泛传播和不断暴发,发现处在或靠近聚合酶区和衣壳VP1区重叠区易发生突变及重组,因此国际上推荐使用聚合酶区及VP1区双区域分型系统[4, 13]。基因分型标准早期采用未校正的遗传距离,诺如病毒型别和序列的增多导致原有的分型方法将型别错误分类,研究者在原有方法的基础上,优化遗传距离分类标准,提出了2倍标准差(2 s)校正后遗传距离的分型方法。依据此方法,将基因型重新划分,鉴定出新的基因组和基因型,并发现聚合酶区2个暂定基因组及10余种暂定基因型,VP1区2个暂定基因组及5种暂定基因型[13]。暂定基因组和基因型有待进一步型别鉴定和基因组划分。对诺如病毒分子特征、不同分型方法的原理、序列扩增方法、在线分型工具及最新的基因分型研究进展进行综述,旨在阐明如何开展诺如病毒分型,推动国内诺如病毒分型方法改进及标准化,提高我国诺如病毒监测水平。
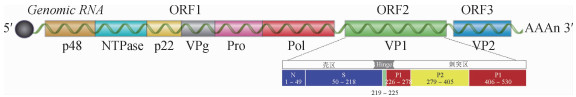
一、分子特征诺如病毒基因组核酸全长约7 000~7 700 bp,分为3个开放阅读框(open reading frames,ORFs)[1, 14-15]。其中ORF1编码6种非结构蛋白,分别是氨基末端蛋白(p48)、核苷三磷酸酶(NTPase)、3a样蛋白(p22)、基因组连接病毒蛋白(VPg)、半胱氨酸蛋白酶(Pro)和RNA依赖性RNA聚合酶(Pol)。诺如病毒衣壳由主要衣壳蛋白VP1和次要结构蛋白VP2组成,分别由ORF2和ORF3编码,衣壳蛋白VP1长539 aa,主要分为壳区(N、S区)和刺突区(P区),S区和P区通过铰链区(H区)连接,其中P区由P1亚区和P2亚区组成,见图 1[1,16]。
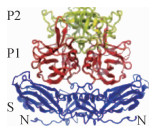
诺如病毒重组病毒样颗粒(recombinant virus-like particles,rVLPs)三维结构X射线晶体学研究显示,VP1区衣壳蛋白的P1及P2区形成拱形结构,P2结构域位于拱形顶部,由两个P1结构域“臂”支撑,S结构域形成围绕RNA基因组的病毒离子的内部支架,并将拱形定位到病毒离子表面,见图 2[17-19]。研究显示,P2结构域内存在组织血型抗原(HBGA)碳水化合物结合位点,为诺如病毒与宿主细胞相互作用和免疫识别发生的主要部位,为诺如病毒基因组中变异最活跃的区域,结合HBGAs的不同将直接影响病毒感染种群的发病率[17, 20]。通过病毒抗原变异,从而易于与不同的HBGAs结合以及逃避机体免疫应答[21-22]。VP2结构蛋白对诺如病毒颗粒重组有影响,其对VP1表达的上调和病毒结构中VP1的稳定性有关[23]。研究显示重组主要发生在ORF1~ORF2重叠区附近,少数发生在ORF2~ORF3重叠区[24-27]。
二、基因型分型方法1.基于未校正配对遗传距离的VP1区氨基酸序列分型方法:2006年Zheng等[12]报道基于未校正配对遗传距离的VP1区氨基酸序列分型方法。通过对GenBank收录的诺如病毒VP1区序列进行比对、氨基酸翻译及贝叶斯系统发育分析,计算系统发育树不同分支之间的未校正配对遗传距离,获得诺如病毒基因组别和基因型别的判定标准:如果毒株遗传距离 < 15%,属于同一基因型;如果遗传距离在15%~45%之间,则属于同一基因组;如果遗传距离>45%,则属于不同基因组。研究结果显示,诺如病毒被分为5组GⅠ~GⅤ,GⅠ组有8种基因型(GⅠ.1~8),GⅡ组有17种基因型(GⅡ.1~17),GⅢ组有2种基因型(GⅢ.1~2),GⅣ及GⅤ组均有1种基因型(GⅣ.1、GⅤ.1),其中新分型6种基因型,包括GⅠ.8及GⅡ.13~17。
2.基于2 s标准的聚合酶区和VP1区的双区域分型方法:2013年Kroneman等[28]对GenBank收录的诺如病毒VP1区和聚合酶区的氨基酸、核苷酸序列分别进行比对,运用最佳模型构建贝叶斯树、最大似然树,输出进化树中不同基因型的遗传距离,计算同种基因型和同种基因组的平均遗传距离及其标准差,得出基因型判定标准:当与同源性最高的基因型的遗传距离在该基因型的平均遗传距离±2 s范围内,则属于该基因型;当与已知同源性最高的基因型的遗传距离超过该基因型的平均遗传距离±2 s,则为新基因型,该分型标准即2 s标准。据此,VP1区分型:GⅠ组有9种基因型(GⅠ.1~9),GⅡ组有22种基因型(GⅡ.1~22),其中GⅠ及GⅡ组共鉴定4种新基因型,为GⅠ.9、GⅡ.20、22。聚合酶区分型结果显示,GⅡ组共分为18种基因型(GⅡ.1~4、GⅡ.6~8、GⅡ.11~13、GⅡ.16、GⅡ.18、GⅡ.20~22、GⅡ.c、GⅡ.e及GⅡ.g),与VP1鉴定的基因型相符合[28]。
三、诺如病毒基因型近年来,诺如病毒序列的增多及新型诺如病毒的出现促使更新基因分型。2019年Chhabra等[13]依据2 s标准分型方法将GenBank收录的VP1区和聚合酶区序列重新计算和分型。
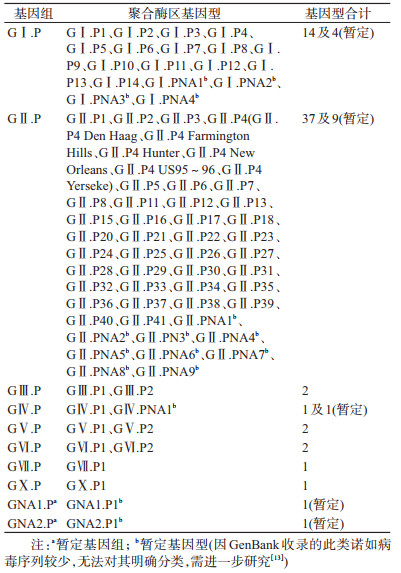
1. VP1区基因型:诺如病毒VP1区基因组共分为12组,包括明确分类的基因组10组,即GⅠ~GⅩ,以及新暂定基因组2组,即GNA1、GNA2,共分为48种确定基因型及5种新暂定基因型,见表 1[13, 28-33]。48种明确分类的基因型,其中9种为GⅠ组(GⅠ.1~9)、26种为GⅡ组(GⅡ.1~14、16~27)、3种为GⅢ组(GⅢ.1~3),GⅣ组、GⅤ组、GⅥ组各2种(GⅣ.1~2、GⅤ.1~2、GⅥ.1~2),GⅦ.1、GⅧ.1、GⅨ.1及GⅩ各1种(GⅦ.1、GⅧ.1、GⅨ.1和GⅩ.1)[13, 28-33]。5种新暂定基因型包括GⅡ组2种(GⅡ.NA1、GⅡ.NA2)、GⅣ及暂定基因组GNA1和GNA2各1种(GⅣ.NA1、GNA1.1、GNA2.1)[13, 28-33]。其中GⅡ.4又被分为不同基因亚型,包括GⅡ.4 Asia、GⅡ.4 Den Haag、GⅡ.4 Farmington Hills、GⅡ.4 Hong Kong、GⅡ.4 Hunter、GⅡ.4 New Orleans、GⅡ.4 Osaka、GⅡ.4 Sydney、GⅡ.4 US95~96、GⅡ.4 Yerseke[29-33]。
可感染人的诺如病毒VP1区基因组共5组,为GⅠ、GⅡ、GⅣ、GⅧ及GⅨ,包括38种可感染人的基因型,其中9种为GⅠ组(GⅠ.1~9)、25种为GⅡ组(GⅡ.1~10、GⅡ.12~14、GⅡ.16、GⅡ.17、GⅡ.20~27及暂定基因型GⅡ.NA1~GⅡ.NA2)、2种为GⅣ组(GⅣ.1及GⅣ.NA1)、GⅧ、GⅨ组各1种(GⅧ.1、GⅨ.1)[13, 31, 33]。
可感染动物的诺如病毒VP1区基因组共8组,为GⅡ、GⅢ、GⅣ、GⅤ、GⅥ、GⅦ、GNA1及GNA2,包括15种可感染动物的基因型[13, 29-32, 34]。其中感染猪的基因型为GⅡ.11、GⅡ.18、GⅡ.19,属于GⅡ组;感染牛的基因型为GⅢ.1、GⅢ.2,属于GⅢ组;感染羊的基因型为GⅢ.3,属于GⅢ组;感染鼠的基因型为GⅤ.1、GⅤ.2,属于GⅤ组;感染犬的基因型为GⅣ.2、GⅥ.1、GⅥ.2、GⅦ.1,分别属于GⅣ、GⅥ及GⅦ组;感染菊头蝠的基因型为GⅩ.1,属于GⅩ组;感染港湾鼠海豚的基因型为GNA1.1,属于GNA1组;感染海狮的基因型为GNA2.1,属于GNA2组[13, 29-32, 34]。
2.聚合酶区基因型:诺如病毒的聚合酶区基因组共分为10组,其中明确分类的基因组共8组,即GⅠ.P~GⅩ.P,新暂定基因组为2组,即GNA1.P、GNA2.P,共分为60种确定基因型及16种新暂定基因型,见表 2[13, 29-30, 32-34]。60种确定基因型,其中14种为GⅠ.P组(GⅠ.P1~14)、37种为GⅡ.P组(GⅡ.P1~8、11~13、15~18、20~41),GⅢ.P、GⅤ.P及GⅥ.P组各2种(GⅢ.P1~2、GⅤ.P1~2、GⅥ.P1~2),GⅣ.P、GⅦ.P及GⅩ.P组各1种(GⅣ.P1、GⅦ.P1、GⅩ.P1)[12-13, 28-32]。16种新暂定基因型包括GⅠ.P组4种(GⅠ.PNA1~4)、GⅡ组9种(GⅡ.PNA1~9),GⅣ、GNA1.P及GNA2.P各1种(GⅣ.PNA、GNA1.P、GNA2.P1)[32-33, 35]。其中GⅡ.P4又被分为不同基因亚型,包括GⅡ.P4 Den Haag、GⅡ.P4 Farmington Hills、GⅡ.P4 Hunter、GⅡ.P4 New Orleans、GⅡ.P4 US95~96、GⅡ.P4 Yerseke[13, 29-30, 32-34]。
可感染人的诺如病毒聚合酶区基因组共3组,为GⅠ.P、GⅡ.P、GⅣ.P,包括63种可感染人的基因型,其中18种为GⅠ组(GⅠ.P1~14、GⅠ.PNA1~4)、43种为GⅡ组(GⅡ.P1~8、GⅡ.P12~13、GⅡ.P15~17、GⅡ.P20~41、GⅡ.PNA1~3、GⅡ.PNA5~9)、2种为GⅣ组(GⅣ.P1及GⅣ.PNA1)[12-13, 31, 33]。
感染动物的诺如病毒聚合酶区基因组共8组,为GⅡ.P、GⅢ.P、GⅤ.P、GⅥ.P、GⅩ.P、GNA.P1及GNA.P2,包括13种可感染动物的基因型[12-13, 29-32, 34]。其中感染猪的基因型为GⅡ.P11、GⅡ.P18、GⅡ.PNA4,属于GⅡ.P组;感染牛的基因型为GⅢ.P1、GⅢ.P2,属于GⅢ.P组;感染鼠的基因型为GⅤ.P1、GⅤ.P2,属于GⅤ.P组;感染犬的基因型为GⅥ.P1、GⅥ.P2、GⅦ.P1,分别属于GⅥ.P及GⅦ.P组;感染菊头蝠的基因型是GⅩ.P1,属于GⅩ.P组;感染港湾鼠海豚的基因型是GNA1.P1,属于GNA1.P组;感染海狮的基因型是GNA2.P1,属于GNA2.P基因组[29-32, 34]。
3.国内外优势基因型:虽然诺如病毒分型众多,但引起人急性胃肠炎的诺如病毒主要为GⅡ组,部分为GⅠ组,少数由其他组引起[32-33]。人群中不同HBGAs表型的个体对诺如病毒结合力不同[22]。“分泌型”个体,即FUT2基因未失活,可分泌转移酶,在HBGAs上加一个α(1,2)-盐藻糖,使之成为ABO抗原的前体H抗原。分泌型个体对大多数诺如病毒易感,尤其是GⅡ.4,“非分泌型”个体,即FUT2基因失活,不能形成H抗原,对大部分诺如病毒耐受[36]。近期研究显示,GⅡ.4也可表现出结合非分泌型HBGAs Lea抗原的能力[37-38]。
因此,诺如病毒中最主要优势株是GⅡ.4,不同变异株及重组株的检出率占全球因诺如病毒引发疫情的55%~85%,其易于发生抗原漂移,通过与HBGAs之间相互作用,导致适应性变异的长期积累,形成新的亚型,发生HBGAs结合特异性变化,导致人群大流行[32, 39]。自20世纪90年代中期报道GⅡ.P4-GⅡ.4成为世界诺如病毒流行株之后,每隔几年就会产生新的基因型,引起全球的诺如病毒感染的暴发流行,截至目前,共引起6次大流行,分别由US95~96(1995年)、Farmington Hills(2002年)、Hunter(2004年)、Yerseke和Den Haag(2006年)、Neworlean(2009年)及Sydney(2012年)引起。2012年后GⅡ.P31(GⅡ.Pe)-GⅡ.4 Sydney 2012株成为国内外优势流行株[40]。
GⅡ.P17-GⅡ.17于1978年首次发现,2012年以前流行率低,2012-2013年,GⅡ.P17-GⅡ.17占肯尼亚河流沿岸农村和城市地区水环境检测到的诺如病毒的76%[41]。2014-2015年冬季,GⅡ.P17-GⅡ.17新亚型出现,在韩国、日本及中国等亚洲地区引起急性胃肠炎暴发,造成了严重的疾病负担[42]。在中国,报道显示占广东省和江苏省的暴发疫情中的82%和70%,在中国香港地区取代了GⅡ.4 Sydney 2012成为优势株,感染85%发生于学校,其次是工厂与幼儿园,易感人群主要为大龄儿童和成年人[43-44]。GⅡ.P17-GⅡ.17被分为4个进化分支(A~D),C、D支为在中国、韩国和日本引起暴发的主要毒株,在欧洲、美国和澳大利亚仅偶有发现[42]。
GⅡ.P16-GⅡ.2于2006-2007年冬季突然出现,并迅速成为优势流行株,在德国、日本、法国、美国和澳大利亚等均检测到这种病毒引起的暴发[45]。2016年冬季,GⅡ.P16-GⅡ.2新进化分支出现,引起的胃肠炎病例于既往同期相比大幅度激增[46]。研究推断,GⅡ.P16聚合酶区影响聚合酶功能和病毒传播的位点发生碱基突变,故而增强了病毒复制和传播的能力,提高了病毒适应性[46]。
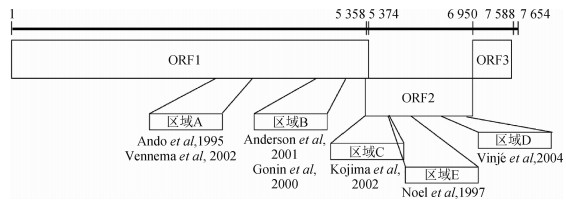
四、常用基因分型区域基于诺如病毒基因型和序列的多样性,扩增聚合酶区和VP1区序列全长较为困难,研究显示聚合酶区和VP1区中的不同基因区域可用于基因分型,从而减少扩增难度[29, 34]。聚合酶区分型区域有A和B,衣壳区分型为C、D及E,其中常用的聚合酶区为B、衣壳区C,见图 3[27,31,34,47-48]。诺如病毒ORF2区C及D的基因分型方案比较,发现D分型实验退火温度较低,为40 ℃,增加非特定序列的扩增,并且D位于ORF2的更可变部分,故C分型成功率高于D,分型成功率分别为78%、52%,但D更助于鉴定GⅡ.4变异株的序列遗传多样性。基于整体性能,研究者推荐诺如病毒的ORF2基因分型区域为C,D可用于识别GⅡ.4变异[27, 31]。
目前GⅠ组常用的VP1区分型引物,正向引物G1SKF(5′- CTGCCCGAATTYGTAAATGA-3′,5 342~5 361 bp)及反向引物G1SKR(5′-CCAACCCARCCATTRTACA-3′,5 653~5 671 bp),获得片段长度为330 bp[33, 35]。以及正向引物COG1F(5′-CGYTGGATGCGNTTYCATGA-3′,5 291~5 310 bp),与反向引物G1SKR,获得片段长度为381 bp[29, 30, 37]。GⅠ组可同时进行聚合酶区和VP1区分型的正向引物为MON432(5′-TGGACICGYGGICCYAAYCA- 3′,4 811~4 830 bp),反向引物G1SKR,获得片段长度为579 bp[32-33, 35]。
目前GⅡ组常用的VP1区分型引物,正向引物为G2SKF(5′-CNTGGGAGGGCGATCGCAA-3′,5 058~5 076 bp)与反向引物G2SKR(5′- CCRCCNGCATRHCCRTTRTACA-3′,5 379~5 401 bp),获得片段长度为344 bp[33, 35],以及COG2F(5′-CARGARBCNATGTTYAGRTGGATGAG-3′,5 003~5 028 bp)与G2SKR,获得片段长度为387 bp[30, 34, 36]。GⅡ组可同时进行聚合酶区和VP1区分型的正向引物为MON431(5′-TGGACIAGRGGICCYAAYCA-3′,4 820~4 838 bp),反向引物G2SKR,获得片段长度为570 bp[32, 35]。
五、诺如病毒命名基于诺如病毒聚合酶区和VP1区双区域分型,为了区分混合感染和重组,提出诺如病毒聚合酶区和VP1区双区域双命名系统,其聚合酶区基因型由大写P指定,如GⅡ.P4,然后是基因型指定,如GⅡ.4,即GⅡ.P4-GⅡ.4。命名系统包括诺如病毒/基因组别/宿主/国家/检出时间/基因型/毒株编号,例:诺如病毒已获得聚合酶区和VP1区双分型结果,则毒株名称应为:norovirus GⅡ/Hu/US/2010/GⅡ.P12-GⅡ. 12/HS206[28]。仅VP1序列已知,名称应为:norovirus/GⅡ/Hu/AU/2012/GⅡ.4 Sydney/Melbourne456[12, 28]。
六、在线分型软件1.荷兰在线分型软件:Kroneman等[49]开发诺如病毒在线基因分型工具,其分型原理共分为3步:①纳入感染人的杯状病毒科病毒的基因序列作为参考序列,对目的序列进行BLAST比对,确定其是否为诺如病毒及其不同基因组,当BLAST分析期望值(E) < 10-5时,可给出目的序列的片段长度和基因组片段位置;②对已鉴定成功的诺如病毒序列,且序列长度VP1区>100 bp或聚合酶区在100~800 bp之间,通过序列比对及系统发育分析,对诺如病毒的聚合酶区和VP1区序列进行分型;③通过对GⅡ.4序列比对,运用邻连法和HKY85-36替代模型构建系统发育树,进行基因亚型及变异株分型。目前该方法已通过验证并广泛使用,目前软件已更新为Norovirus Typing Tool Version 2.0,其网址为https://www.rivm.nl/mpf/typingtool/norovirus/。
2.美国在线分型软件:美国CaliciNet实验室推出诺如病毒在线分型软件[50],其原理是使用类似于BLAST算法,根据不同基因型别的参考序列,对GⅠ、GⅡ、GⅣ、GⅧ和GⅨ组的聚合酶区和衣壳区序列进行分型,并对GⅠ、GⅡ、GⅣ和GⅤ的札如病毒衣壳区进行分型[26]。基因组命名会根据NoroNet命名工作组的建议进行更新,分型参考序列分别依据最新的研究进展进行更新[13, 28, 39, 51]。目前该方法已通过验证,其网址为https://norovirus.phiresearchlab.org/bctyping.html。
七、展望随着GenBank中诺如病毒收录序列增多,诺如病毒基因分型更加明确和细化,分型方法从2006年未校正配对遗传距离的VP1区氨基酸序列分型方法,发展和更新为2013年基于2 s标准的诺如病毒聚合酶区和VP1区的双区域分型方法。运用该方法2019年诺如病毒基因分型细化到聚合酶区已确定60种基因型及16种暂定基因型,VP1区已确定48种基因型及5种暂定基因型。诺如病毒极易变异,新型诺如病毒以及重组诺如病毒不断出现,新的变异株因突变或重组从而提高病毒适应性和感染性,常常导致急性胃肠炎暴发流行。今后需进一步探究诺如病毒分型方法,推动国内诺如病毒分型方法改进及标准化,提高我国诺如病毒监测水平,为新型诺如病毒以及重组诺如病毒的发现和鉴定提供方法支持,对预防诺如病毒引起的急性胃肠炎暴发流行奠定基础。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Inns T, Harris J, Vivancos R, et al. Community-based surveillance of norovirus disease:a systematic review[J]. BMC Infect Dis, 2017, 17(1): 657-667. DOI:10.1186/s12879-017-2758-1 |
| [2] |
Lindesmith LC, Brewer-Jensen PD, Mallory ML, et al. Virus-host interactions between nonsecretors and human norovirus[J]. Cell Mol Gastroenterol Hepatol, 2020, 10(2): 245-267. DOI:10.1016/j.jcmgh.2020.03.006 |
| [3] |
Queiros-Reis L, Lopes-João A, Mesquita JR, et al. Norovirus gastroenteritis outbreaks in military units: a systematic review[J]. BMJ Mil Health, May 2020. DOI: 10.1136/bmjmilitary-2019-001341.
|
| [4] |
Kapikian AZ, Wyatt RG, Dolin R, et al. Visualization by immune electron microscopy of a 27-nm particle associated with acute infectious nonbacterial gastroenteritis[J]. J Virol, 1972, 10(5): 1075-1081. DOI:10.1128/JVI.10.5.1075-1081.1972 |
| [5] |
Hoa Tran TN, Trainor E, Nakagomi T, et al. Molecular epidemiology of noroviruses associated with acute sporadic gastroenteritis in children:global distribution of genogroups, genotypes and GⅡ.4 variants[J]. J Clin Virol, 2013, 56(3): 185-193. DOI:10.1016/j.jcv.2012.11.011 |
| [6] |
王宇, 贾蕾, 高志勇, 等. 综合指数法建立北京市诺如病毒急性胃肠炎流行水平分级标准[J]. 国际病毒学杂志, 2019, 26(4): 225-228. Wang Y, Jia L, Gao ZY, et al. Establishment of grading thresholds for epidemic intensity of norovirus acute gastroenteritis in Beijing by synthetic index method[J]. Int J Virol, 2019, 26(4): 225-228. DOI:10.3760/cma.j.issn.1673-4092.2019.04.004 |
| [7] |
Hall AJ, Wikswo ME, Pringle K, et al. Vital signs:foodborne norovirus outbreaks-United States, 2009-2012[J]. MMWR Morb Mortal Wkly Rep, 2014, 63(22): 491-495. |
| [8] |
Hall AJ, Eisenbart VG, Etingüe AL, et al. Epidemiology of foodborne norovirus outbreaks, United States, 2001-2008[J]. Emerg Infect Dis, 2012, 18(10): 1566-1573. DOI:10.3201/eid1810.120833 |
| [9] |
Ahmed SM, Hall AJ, Robinson AE, et al. Global prevalence of norovirus in cases of gastroenteritis:a systematic review and Meta-analysis[J]. Lancet Infect Dis, 2014, 14(8): 725-730. DOI:10.1016/S1473-3099(14)70767-4 |
| [10] |
Matsui SM, Kim JP, Greenberg HB, et al. The isolation and characterization of a Norwalk virus-specific cDNA[J]. J Clin Invest, 1991, 87(4): 1456-1461. DOI:10.1172/JCI115152 |
| [11] |
Xi JN, Graham DY, Wang KN, et al. Norwalk virus genome cloning and characterization[J]. Science, 1990, 250(4987): 1580-1583. DOI:10.1126/science.2177224 |
| [12] |
Zheng DP, Ando T, Fankhauser RL, et al. Norovirus classification and proposed strain nomenclature[J]. Virology, 2006, 346(2): 312-323. DOI:10.1016/j.virol.2005.11.015 |
| [13] |
Chhabra P, de Graaf M, Parra GI, et al. Updated classification of norovirus genogroups and genotypes[J]. J Gen Virol, 2019, 100(10): 1393-1406. DOI:10.1099/jgv.0.001318 |
| [14] |
Thorne LG, Goodfellow IG. Norovirus gene expression and replication[J]. J Gen Virol, 2014, 95(2): 278-291. DOI:10.1099/vir.0.059634-0 |
| [15] |
Prasad BV, Crawford S, Lawton JA, et al. Structural studies on gastroenteritis viruses[J]. Novartis Found Symp, 2001, 238: 26-37. DOI:10.1002/0470846534.ch3 |
| [16] |
Prasad BVV, Hardy ME, Dokland T, et al. X-ray crystallographic structure of the Norwalk virus capsid[J]. Science, 1999, 286(5438): 287-290. DOI:10.1126/science.286.5438.287 |
| [17] |
Cao S, Lou ZY, Tan M, et al. Structural basis for the recognition of blood group trisaccharides by norovirus[J]. J Virol, 2007, 81(11): 5949-5957. DOI:10.1128/JVI.00219-07 |
| [18] |
Choi JM, Hutson AM, Estes MK, et al. Atomic resolution structural characterization of recognition of histo-blood group antigens by Norwalk virus[J]. Proc Natl Acad Sci USA, 2008, 105(27): 9175-9180. DOI:10.1073/pnas.0803275105 |
| [19] |
Verhoef LPB, Kroneman A, van Duynhoven Y, et al. Selection tool for foodborne norovirus outbreaks[J]. Emerg Infect Dis, 2009, 15(1): 31-38. DOI:10.3201/eid1501.080673 |
| [20] |
Robilotti E, Deresinski S, Pinsky BA. Norovirus[J]. Clin Microbiol Rev, 2015, 28(1): 134-164. DOI:10.1128/cmr.00075-14 |
| [21] |
Graziano VR, Wei J, Wilen CB. Norovirus attachment and entry[J]. Viruses, 2019, 11(6): 495. DOI:10.3390/v11060495 |
| [22] |
Zhang DS, Tan M, Zhong WM, et al. Human intestinal organoids express histo-blood group antigens, bind norovirus VLPs, and support limited norovirus replication[J]. Sci Rep, 2017, 7: 12621. DOI:10.1038/s41598-017-12736-2 |
| [23] |
Donaldson EF, Lindesmith LC, Lobue AD, et al. Viral shape-shifting:norovirus evasion of the human immune system[J]. Nat Rev Microbiol, 2010, 8(3): 231-241. DOI:10.1038/nrmicro2296 |
| [24] |
蒋翠莲, 曹雪, 艾静, 等. GⅡ.P21/GⅡ.3型重组诺如病毒的分子特征分析[J]. 国际病毒学杂志, 2019, 26(4): 233-236. Jiang CL, Cao X, Ai J, et al. Analysis on the molecular characteristics of GⅡ.P21/GⅡ.3 recombinant norovirus[J]. Int J Virol, 2019, 26(4): 233-236. DOI:10.3760/cma.j.issn.1673-4092.2019.04.006 |
| [25] |
Fu J, Ai J, Jin M, et al. Emergence of a new GⅡ.17 norovirus variant in patients with acute gastroenteritis in Jiangsu, China, September 2014 to March 2015[J]. Euro Surveill, 2015, 20(24): 21157. DOI:10.2807/1560-7917.es2015.20.24.21157 |
| [26] |
Matsushima Y, Shimizu T, Ishikawa M, et al. Complete genome sequence of a recombinant GⅡ.P16-GⅡ.4 norovirus detected in Kawasaki City, Japan, in 2016[J]. Genome Announc, 2016, 4(5): e01099-16. DOI:10.1128/genomeA.01099-16 |
| [27] |
Mattison K, Grudeski E, Auk B, et al. Multicenter comparison of two norovirus ORF2-based genotyping protocols[J]. J Clin Microbiol, 2009, 47(12): 3927-3932. DOI:10.1128/JCM.00497-09 |
| [28] |
Kroneman A, Vega E, Vennema H, et al. Proposal for a unified norovirus nomenclature and genotyping[J]. Arch Virol, 2013, 158(10): 2059-2068. DOI:10.1007/s00705-013-1708-5 |
| [29] |
Ando T, Noel JS, Fankhauser RL. Genetic classification of "Norwalk-like viruses"[J]. J Infect Dis, 2000, 181(Suppl 2): S336-348. DOI:10.1086/315589 |
| [30] |
Gao ZY, Liu BW, Huo D, et al. Increased norovirus activity was associated with a novel norovirus GⅡ.17 variant in Beijing, China during winter 2014-2015[J]. BMC Infect Dis, 2015, 15: 574. DOI:10.1186/s12879-015-1315-z |
| [31] |
Vinjé J. Advances in laboratory methods for detection and typing of norovirus[J]. J Clin Microbiol, 2015, 53(2): 373-381. DOI:10.1128/JCM.01535-14 |
| [32] |
Cannon JL, Barclay L, Collins NR, et al. Genetic and epidemiologic trends of Norovirus outbreaks in the United States from 2013 to 2016 demonstrated emergence of Novel GⅡ.4 recombinant viruses[J]. J Clin Microbiol, 2017, 55(7): 2208-2221. DOI:10.1128/JCM.00455-17 |
| [33] |
Gao ZY, Liu BW, Yan HQ, et al. Norovirus outbreaks in Beijing, China, from 2014 to 2017[J]. J Infect, 2019, 79(2): 159-166. DOI:10.1016/j.jinf.2019.05.019 |
| [34] |
Vinjé J, Hamidjaja RA, Sobsey MD. Development and application of a capsid VP1(region D) based reverse transcription PCR assay for genotyping of genogroup Ⅰ and Ⅱ noroviruses[J]. J Virol Methods, 2004, 116(2): 109-117. DOI:10.1016/j.jviromet.2003.11.001 |
| [35] |
Kojima S, Kageyama T, Fukushi S, et al. Genogroup-specific PCR primers for detection of Norwalk-like viruses[J]. J Virol Methods, 2002, 100(1/2): 107-114. DOI:10.1016/s0166-0934(01)00404-9 |
| [36] |
Ferrer-Admetlla A, Sikora M, Laayouni H, et al. A natural history of FUT2 polymorphism in humans[J]. Mol Biol Evol, 2009, 26(9): 1993-2003. DOI:10.1093/molbev/msp108 |
| [37] |
Jin M, He YQ, Li HY, et al. Two gastroenteritis outbreaks caused by GⅡ Noroviruses:host susceptibility and HBGA phenotypes[J]. PLoS One, 2013, 8(3): e58605. DOI:10.1371/journal.pone.0058605 |
| [38] |
Nordgren J, Kindberg E, Lindgren PE, et al. Norovirus gastroenteritis outbreak with a secretor-independent susceptibility pattern, Sweden[J]. Emerg Infect Dis, 2010, 16(1): 81-87. DOI:10.3201/eid1601.090633 |
| [39] |
Yu YX, Yan SL, Li BL, et al. Genetic diversity and distribution of human norovirus in China (1999-2011)[J]. Biomed Res Int, 2014, 2014: 196169. DOI:10.1155/2014/196169 |
| [40] |
Farsi M, Roodbari F, Nejati B, et al. Prevalence and genetic diversity of norovirus genogroup Ⅱ in children less than 5 years of age with acute gastroenteritis in Tehran, Iran[J]. Med Microbiol Immunol, 2018, 207(3/4): 201-210. DOI:10.1007/s00430-018-0541-6 |
| [41] |
Kiulia NM, Mans J, Mwenda JM, et al. Norovirus GⅡ.17 Predominates in selected surface water sources in Kenya[J]. Food Environ Virol, 2014, 6(4): 221-231. DOI:10.1007/s12560-014-9160-6 |
| [42] |
Sang SW, Yang XY. Evolutionary dynamics of GⅡ.17 norovirus[J]. Peer J, 2018, 6: e4333. DOI:10.7717/peerj.4333 |
| [43] |
Lu J, Sun LM, Fang L, et al. Gastroenteritis outbreaks caused by norovirus GⅡ.17, Guangdong province, China, 2014-2015[J]. Emerg Infect Dis, 2015, 21(7): 1240-1242. DOI:10.3201/eid2107.150226 |
| [44] |
Han JK, Ji L, Shen YH, et al. Emergence and predominance of norovirus GⅡ.17 in Huzhou, China, 2014-2015[J]. Virol J, 2015, 12: 139. DOI:10.1186/s12985-015-0370-9 |
| [45] |
Niendorf S, Jacobsen S, Faber M, et al. Steep rise in norovirus cases and emergence of a new recombinant strain GⅡ.P16-GⅡ.2, Germany, winter 2016[J]. Euro Surveill, 2017, 22(4): 30447. DOI:10.2807/1560-7917.ES.2017.22.4.30447 |
| [46] |
Ao YY, Cong X, Jin M, et al. Genetic analysis of reemerging GⅡ.P16-GⅡ.2 noroviruses in 2016-2017 in China[J]. J Infect Dis, 2018, 218(1): 133-143. DOI:10.1093/infdis/jiy182 |
| [47] |
靳淼, 孙军玲, 常昭瑞, 等. 中国2006-2007年诺如病毒胃肠炎暴发及其病原学特征分析[J]. 中华流行病学杂志, 2010, 31(5): 549-553. Jin M, Sun JL, Chang ZR, et al. Outbreaks of noroviral gastroenteritis and their molecular characteristics in China, 2006-2007[J]. Chin J Epidemiol, 2010, 31(5): 549-553. DOI:10.3760/cma.j.issn.0254-6450.2010.05.017 |
| [48] |
Vinjé J, Vennema H, Maunula L, et al. International collaborative study to compare reverse transcriptase PCR assays for detection and genotyping of noroviruses[J]. J Clin Microbiol, 2003, 41(4): 1423-1433. DOI:10.1128/jcm.41.4.1423-1433.2003 |
| [49] |
Kroneman A, Vennema H, Deforche K, et al. An automated genotyping tool for enteroviruses and noroviruses[J]. J Clin Virol, 2011, 51(2): 121-125. DOI:10.1016/j.jcv.2011.03.006 |
| [50] |
Oka T, Wang QH, Katayama K, et al. Comprehensive review of human sapoviruses[J]. Clin Microbiol Rev, 2015, 28(1): 32-53. DOI:10.1128/CMR.00011-14 |
| [51] |
Vinjé J, Estes MK, Esteves P, et al. ICTV virus taxonomy profile:Caliciviridae[J]. J Gen Virol, 2019, 100(11): 1469-1470. DOI:10.1099/jgv.0.001332 |
 2020, Vol. 41
2020, Vol. 41