文章信息
- 陈涛, 肖梦, 禇堃, 唐晓君, 杨静, 舒跃龙.
- Chen Tao, Xiao Meng, Chu Kun, Tang Xiaojun, Yang Jing, Shu Yuelong
- TNF-α及CCR5Δ32基因多态性与A(H1N1)pdm09相关性的Meta分析
- Meta-analysis on association between TNF-α and CCR5Δ32 gene polymorphisms and influenza A(H1N1)pdm09
- 中华流行病学杂志, 2020, 41(11): 1909-1914
- Chinese Journal of Epidemiology, 2020, 41(11): 1909-1914
- http://dx.doi.org/10.3760/cma.j.cn112338-20200209-00082
-
文章历史
收稿日期: 2020-02-09
2. 重庆医科大学公共卫生与管理学院 400016;
3. 中山大学公共卫生学院(深圳), 广州 510275
2. School of Public Health and Management, Chongqing Medical University, Chongqing 400016, China;
3. School of Public Health(Shenzhen), Sun Yat-sen University, Guangzhou 510275, China
肿瘤坏死因子-α(tumor necrosis factor-α,TNF-α)是一种参与各种炎症和免疫应答的细胞因子,被认为是宿主防御感染的一部分,在许多自身免疫疾病和传染病的发病机制中起作用[1]。其中有研究发现其与流感病毒感染的关联。TNF-α常见单核苷酸多态性(single nucleotide polymorphisms,SNPs)位点包括rs361525(TNF-α-238G>A)、rs1800629(TNF-α-308G>A),rs1800750(TNF-α-376G>A)等[2-3],既往研究发现在TNF-α基因多态性与一些人类疾病之间存在联系[4-5],但TNF-α不同SNP与A(H1N1)pdm09之间的关联并不确定。CCR5则是一种在T细胞、大噬细胞和树突状细胞中表达的趋化因子受体,是白细胞趋化因子的重要中介,在CCR5的编码序列中,有一个32 bp的多态缺失,即CCR5Δ32。研究表明,CCR5基因中的32 bp的缺失通过CCL3、CCL4和CCL5干扰了免疫细胞的反应,影响病毒清除[6-7]。尽管目前有研究表明CCR5Δ32与流感严重程度的相关性,但是各项研究结果并不完全一致。
因此我们针对TNF-α-238G>A(rs361525)、308G>A(rs1800629)、376G>A(rs1800750)及CCR5Δ32(rs333)的多态性进行了Meta分析,以分别探讨TNF-α、CCR5细胞因子基因多态性与A(H1N1)pdm09患病风险、严重程度之间的关系。
材料与方法1.检索策略:在PubMed、Cochrane Library、OVID、EBSCO、Web of Science全面检索2019年2月7日及以前发表的相关文献。针对TNF和CCR5细胞因子的检索策略为“TNF OR tumor necrosis factor OR rs361525 OR rs1800629 OR rs180075 OR CCR OR Delta32 OR rs333”AND“flu OR influenza OR grippe”,具体检索流程见图 1。

|
| 图 1 文献检索流程图 |
2.排除和纳入标准:本研究的纳入标准:①人类研究;②描述了流感与TNF-α或CCR5之间的关系;③观察性研究;④能够获取全文,并从原文提取到可用于计算OR值以及95%CI值的数据;⑤英文文献。排除标准:①病理报告或摘要等;②重复发表的文献。
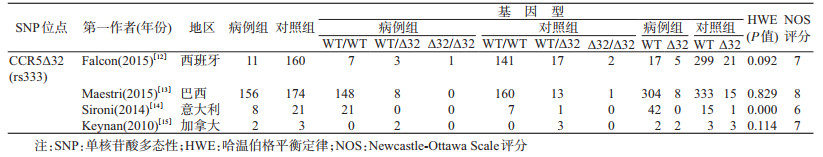
3.数据提取及文献质量评价:两位作者根据已经确定好的排除和纳入标准,独立地对文献进行筛选。意见不一致时,进行讨论,不能解决则需第3位作者对文献进行评价。主要提取基因位点、第一作者姓名及年份、地区、病例组和对照组基因型和等位基因的频率、哈温伯格平衡定律的P值和NOS(Newcastle-Ottawa Scale)评分。NOS量表评分满分为9分,包括研究对象的选择、病例组与对照组研究对象的可比性和结果评价3个方面,共8个条目9个得分点。
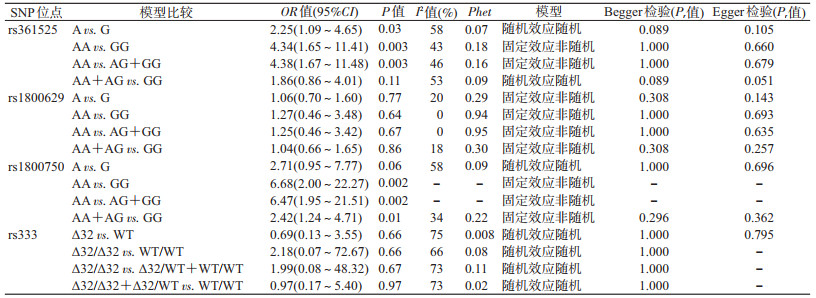
4.统计学分析:采用Revman 5.0软件进行数据统计分析,使用4种遗传模型(A vs. G、AA vs. GG、AA vs. AG+GG、AA+AG vs. GG)的OR值和95%CI,对TNF-α细胞因子的3个不同基因位点(rs361525、rs1800629、rs1800750)以及CCR5Δ32(rs333)与流行性感冒(流感)的关联性进行分析。异质性的计算使用基于Q检验、Thompson和Higgins分类指数(I2)计算的P值。如果P < 0.05或I2>50%,则使用随机效应模型,否则选择固定效应模型。发表偏倚的检测采用Stata软件的Begg’s和Egger’s检验,若P>0.05,提示无发表偏倚;反之,说明存在发表偏倚。
结果1.基本特征:在相关数据库中共检索到3 628篇相关文章,剔除重复文献后,剩余1 868篇文章;阅读题目和摘要后,剔除1 163篇与主题无关的文章,初步保留了705篇文献;阅读全文后,最后保留8篇英文文献[8-15],NOS评分为6~8分,见表 1。涉及的TNF-α相关位点共3个,均为核苷酸G→A的变异,分别是rs361525、rs1800629、rs1800750;3个基因位点的相关文献分别有4、4、3篇;发表时间2012-2015年;1篇是关于希腊人种,另外3篇均是墨西哥人种;病例组(患H1N1)分别纳入371、363、298例;对照组(健康人群)690、690、644例。涉及rs333的文献4篇,发表时间2010-2015年;共含病例组(严重或死亡病例)177例,对照组(轻症或存活病例)358例。纳入原始文献的NOS量表评分以及各位点的基因分布(TNF-α为GG、GA、AA;CCR5为WT/WT、WT/Δ32、Δ32/Δ32)等基本特征见表 1,2。
2. TNF-α各位点Meta分析:
(1)rs361525与A(H1N1)pdm09易感性的关联分析:rs361525位点的Meta分析结果显示,等位基因模型(A vs. G)异质性检验结果I2>50%(P=0.07,I2=58%),说明研究之间异质性较大,采用随机效应模型,此模型结果具有统计学意义(A vs. G:OR=2.25,95%CI:1.09~4.65,P=0.03),携带A等位基因者患A(H1N1)pdm09是G等位基因的2.25倍。纳入的原始文献中,有3篇为墨西哥人种[9-11],另1篇研究针对高加索人种[8],亚组分析的结果提示A等位基因是高加索人种感染A(H1N1)pdm09的危险因素(P < 0.05)。见表 3,图 2。
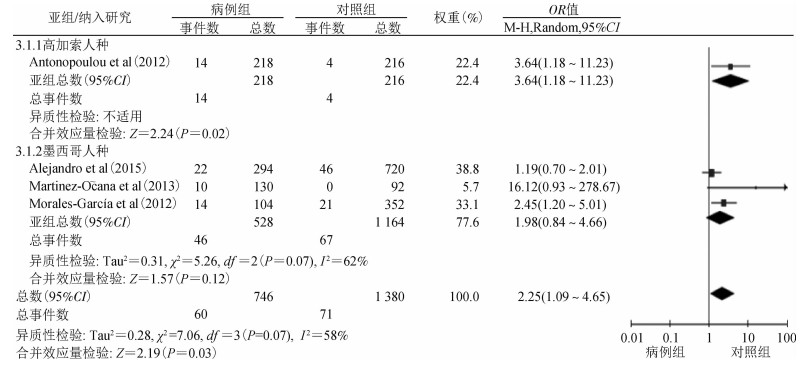
|
| 图 2 rs361525基因多态性与A(H1N1)pdm09易感性的森林图(A vs. G) |
以基因型AA为暴露因素,基因型GG为非暴露因素,针对共显性遗传模型(AA vs. GG)的异质性检验结果显示,研究间异质性较低(P=0.06,I2=43%),采用固定效应模型,模型具有统计学意义(AA vs. GG:OR=4.34,95%CI:1.65~11.41,P=0.003),AA基因型的携带者感染A(H1N1)pdm09的可能性为GG基因型携带者的4.34倍。按照人种进行亚组分析后,结果显示AA基因型是墨西哥人种感染A(H1N1)pdm09的危险因素(P < 0.05)。见表 3。
以基因型AA为暴露因素,基因型AG+GG为非暴露因素的隐性遗传模型(AA vs. AG+GG)的异质性检验结果显示,该模型异质性I2为46%(P=0.16,I2=46%),采用固定效应模型,模型具有统计学意义(AA vs. AG+GG:OR=4.38,95%CI:1.67~11.48,P=0.003)。AA基因型的携带者感染A(H1N1)pdm09的可能性为AG+GG基因型携带者的4.38倍。按照人种进行的亚组分析结果显示,AA基因型是墨西哥人种感染A(H1N1)pdm09的危险因素(P < 0.05)。见表 3。
以基因型AA+AG为暴露因素,基因型GG为非暴露因素,针对显性遗传模型(AA+AG vs. GG)的异质性检验结果显示,该模型异质性I2略高于50%(P=0.09,I2=53%),采用随机效应模型,结果显示差异无统计学意义(AA+AG vs. GG:OR=1.86,95%CI:0.86~4.01,P=0.11)。亚组分析的结果显示,基因型AA+AG是高加索人种感染A(H1N1)pdm09的危险因素(P < 0.05)。见表 3,图 3。

|
| 图 3 rs361525基因多态性与A(H1N1)pdm09易感性的森林图(AA+AG vs. GG) |
(2)rs1800629与A(H1N1)pdm09易感性的关联分析:分别进行rs1800629位点等位基因模型(A vs. G)、共显性遗传模型(AA vs. GG)、隐性遗传模型(AA vs. AG+GG)、显性遗传模型(AA+AG vs. GG)的Meta分析,结果显示rs1800629 A vs. G(OR=1.06,95%CI:0.70~1.60,P=0.77)、AA vs. GG(OR=1.27,95%CI:0.46~3.48,P=0.64)、AA vs. GG(OR=1.25,95%CI:0.46~3.42,P=0.67)、AA+AG vs. GG(OR=1.04,95%CI:0.66~1.65,P=0.86)均与人群感染A(H1N1)pdm09无显著相关性;按照人种分类的亚组分析的结果也未发现此位点与患A(H1N1)pdm09的风险之间的关联(P>0.05)。见表 3。
(3)rs1800750与A(H1N1)pdm09易感性的关联分析:对纳入的rs1800750的相关研究中,有2篇原始研究未检测到AA基因型[8, 11],因此针对共显性遗传模型(AA vs. GG)和隐性遗传模型(AA vs. AG+GG)进行分析时,最后仅1篇文献在此模型中起作用[10],该文献结果显示AA基因型的携带者感染A(H1N1)pdm09的可能性分别为GG基因型、AG+GG携带者的6.68、6.47倍(AA vs. GG:OR=6.68,95%CI:2.00~22.27,P=0.002;AA vs. AG+GG:OR=6.47,95%CI:1.95~21.51,P=0.002)。
针对显性遗传模型(AA+AG vs. GG)的Meta分析结果显示,基因型AA+AG可能是人群感染A(H1N1)pdm09的危险因素(AA+AG vs. GG:OR=2.42,95%CI:1.24~4.71,P=0.01)。见图 4。
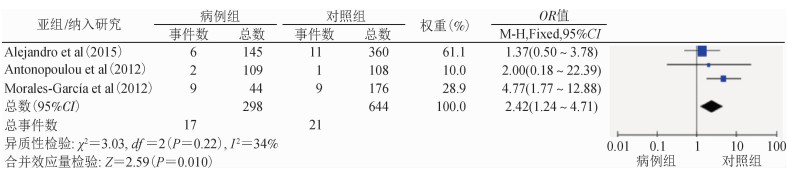
|
| 图 4 rs1800750基因多态性与A(H1N1)pdm09易感性的森林图(AA+AG vs. GG) |
等位基因模型(A vs. G)的结果无统计学意义(OR=2.71,95%CI:0.95~7.77,P=0.06)。此位点按照人种分类后文献过少,因此未进行亚组分析。
3. rs333的Meta分析结果:共4项研究报道了rs333位点,包括病例组(严重病例)177例,对照组(轻症病例)358例。Meta分析结果显示Δ32 vs. WT(OR=0.69,95%CI:0.13~3.55,P=0.66)、Δ32/Δ32 vs. WT/WT(OR=2.18,95%CI:0.07~72.67,P=0.66)、Δ32/Δ32 vs. Δ32/WT+WT/WT(OR=1.99,95%CI:0.08~48.32,P=0.67)、Δ32/Δ32+Δ32/WT vs. WT/WT(OR=0.97,95%CI:0.17~5.40,P=0.97)均为阴性结果,即CCR5Δ32遗传变异与H1N1型流感严重程度无关。见表 3。
4.敏感性分析与发表偏倚:由于各个位点纳入的文章数量不多,因此本研究采取的是排除各位点中,不符合哈温伯格定律(HWE < 0.05)的相关原始文献的方法进行敏感性分析。敏感性分析的结果显示,仅rs1800750位点的结果发生了改变,在排除Morales-García等[9]的文章后,该位点AA+AG vs. GG无统计学意义(OR=2.42,95%CI:1.24~4.71,P=0.43),表明该结果不稳定。本研究的Begger’s和Egger’s检验分析结果显示,各文献之间未发现发表偏倚(P>0.05),见表 3。
讨论本研究发现,针对rs361525来说,具有等位基因A或者AA基因型是感染A(H1N1)pdm09的危险因素(P < 0.05):具有等位基因A的个体感染H1N1的风险是对照组的2.25倍;具有AA基因型的个体感染H1N1的风险甚至是GG基因型个体的4倍以上。此前的一些研究发现,不同人种对于疾病的易感性可能存在差异[16-17],因此本文还进行了分人种的亚组分析。针对不同的人种,结果显示,等位基因A是高加索人种感染A(H1N1)pdm09的危险因素(P < 0.05);AA基因型是墨西哥人种感染A(H1N1)pdm09的危险因素(P < 0.05);尽管显性遗传模型(AA+AG vs. GG)的总分析结果差异无统计学意义(P>0.05),但此模型的亚组分析表明,基因型AA+AG是高加索人种感染A(H1N1)pdm09的危险因素(P < 0.05),因此,人种可能是此模型总分析结果的一个混杂因素,也可能是模型分析时导致异质性的原因之一。
对于rs1800750位点来说,显性遗传模型(AA+AG vs. GG)的Meta分析结果表明,基因型AA+AG可能是人群感染A(H1N1)pdm09的危险因素(P < 0.05),共显性遗传模型(AA vs. GG)以及隐性遗传模型(AA vs. AG+GG)中,部分原始文献的研究对象未检测到AA基因型,因而导致这2个模型最终均只有Morales-García等[9]的文章起作用,2个模型的结果均显示,相对于AA基因型的人群更易感染H1N1型流感。
针对目前国际上已发表的CCR5Δ32与流感相关的文献进行Meta分析后,结果显示尚未发现此位点与A(H1N1)pdm09严重程度之间的联系。4个基因模型的异质性均较大(I2>50%),均采用随机模型进行Meta分析,可能是由于该位点的原始文献的研究人群来自于不同国家,且Maestri等[13]的单篇原始研究针对的也是混合人种,研究对象人种差异较大;此外,原始研究对象的年龄差异(Sironi等[14]的研究未对研究对象年龄进行描述)也可能是导致合并结果异质性较高的原因之一。
在对纳入文献进行敏感性分析时,结果显示,rs1800750位点在排除Morales-García等[9]文章后,显性遗传模型(AA+AG vs. GG)变成了无统计学意义(P=0.43),可能就是纳入群体的该位点基因遗传不平衡导致了该结果的不稳定(哈温伯格定律P < 0.05)。由于几篇原始文献对于PCR的检测方法的描述并没有特别详细,我们不排除不同的文献对于感染病毒的检测方法具有一些细微差异的可能性,这也可能是对此项结果产生影响的原因之一。
从检索到的文献来看,目前TNF-α、CCR5与A(H1N1)pdm09之间的关联存在争议性,这也是本研究目的所在。尽管本研究进行了4个位点的Meta分析,发现了部分相关位点的突变对于A(H1N1)pdm09的感染产生了影响,也尽可能对于能够进行亚组分析的位点进行了不同人群的分析,使结果更有针对性。但本研究仍有局限性,因为在中文数据库中未检索到相关文章,只纳入了英文文献;由于研究参与者的语种所限,也未能纳入其他语种的高质量文献。此外,从总体的文献检索来看,国际上目前对于TNF-α、CCR5与A(H1N1)pdm09之间关联的研究并不多,研究的样本量并不大;由于纳入的原始文献按人种分亚组后过少,rs1800750、rs333纳入的文献未进行亚组分析,合并的结果可能受人种的影响;由于纳入文献未涉及亚洲人种,研究结果的公共卫生意义也具有一定局限性。
综上所述,TNF-α的基因多态性与A(H1N1)pdm09的发病风险可能具有相关性,rs361525位点具有等位基因A或者AA基因型,或rs1800750位点具有基因型AA+AG是感染A(H1N1)pdm09的危险因素。但今后仍需要更多大样本量、设计严谨的科学研究进一步证实相关结论。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Aggarwal BB. Signalling pathways of the TNF superfamily:a double-edged sword[J]. Nat Rev Immunol, 2003, 3(9): 745-756. DOI:10.1038/nri1184 |
| [2] |
Liu N, Liu GJ, Liu J. Genetic association between TNF-α promoter polymorphism and susceptibility to squamous cell carcinoma, basal cell carcinoma, and melanoma:A Meta-analysis[J]. Oncotarget, 2017, 8(32): 53873-53885. DOI:10.18632/oncotarget.17179 |
| [3] |
Rasmussen SK, Urhammer SA, Jensen JN, et al. The -238 and -308 G→A polymorphisms of the tumor necrosis factor α gene promotor are not associated with features of the insulin resistance syndrome or altered birth weight in Danish Caucasians[J]. J Clin Endocrinol Metab, 2000, 85(4): 1731-1734. DOI:10.1210/jcem.85.4.6563 |
| [4] |
Bayley JP, Ottenhoff TH, Verweij CL. Is there a future for TNF promoter polymorphisms?[J]. Genes Immun, 2004, 5(5): 315-329. DOI:10.1038/sj.gene.6364055 |
| [5] |
Baran W, Szepietowski JC, Mazur G, et al. A-308 promoter polymorphism of tumor necrosis factor alpha gene does not associate with the susceptibility to psoriasis vulgaris. No difference either between psoriasis typeⅠand typeⅡpatients[J]. Acta Dermatovenerol Alp Pannonica Adriat, 2006, 15(3): 113-118. |
| [6] |
Sanchooli J, Sanadgol N, Arababadi MK, et al. CCR5 plays important roles in hepatitis B infection[J]. Viral Immunol, 2014, 27(1): 2-6. DOI:10.1089/vim.2013.0067 |
| [7] |
Ghorban K, Dadmanesh M, Hassanshahi G, et al. Is the CCR5Δ32 mutation associated with immune system-related diseases?[J]. Inflammation, 2013, 36(3): 633-642. DOI:10.1007/s10753-012-9585-8 |
| [8] |
Antonopoulou A, Baziaka F, Tsaganos T, et al. Role of tumor necrosis factor gene single nucleotide polymorphisms in the natural course of 2009 influenza A H1N1 virus infection[J]. Int J Infect Dis, 2012, 16(3): e204-208. DOI:10.1016/j.ijid.2011.11.012 |
| [9] |
Morales-García G, Falfán-Valencia R, García-Ramírez RA, et al. Pandemic influenza A/H1N1 virus infection and TNF, LTA, IL1B, IL6, IL8, and CCL polymorphisms in Mexican population:a case-control study[J]. BMC Infect Dis, 2012, 12: 299. DOI:10.1186/1471-2334-12-299 |
| [10] |
Martinez-Ocaña J, Olivo-Diaz A, Salazar-Dominguez T, et al. Plasma cytokine levels and cytokine gene polymorphisms in Mexican patients during the influenza pandemic A(H1N1)pdm09[J]. J Clin Virol, 2013, 58(1): 108-113. DOI:10.1016/j.jcv.2013.05.013 |
| [11] |
García-Ramírez RA, Ramírez-Venegas A, Quintana-Carrillo R, et al. TNF, IL6, and IL1B polymorphisms are associated with severe influenza A (H1N1) virus infection in the Mexican population[J]. PLoS One, 2015, 10(12): e0144832. DOI:10.1371/journal.pone.0144832 |
| [12] |
Falcon A, Cuevas MT, Rodriguez-Frandsen A, et al. CCR5 deficiency predisposes to fatal outcome in influenza virus infection[J]. J Gen Virol, 2015, 96(8): 2074-2078. DOI:10.1099/vir.0.000165 |
| [13] |
Maestri A, dos Santos MC, Ribeiro-Rodrigues EM, et al. The CCR5Δ32(rs333) polymorphism is not a predisposing factor for severe pandemic influenza in the Brazilian admixed population[J]. BMC Res Notes, 2015, 8: 326. DOI:10.1186/s13104-015-1299-1 |
| [14] |
Sironi M, Cagliani R, Pontremoli C, et al. The CCR5Δ32 allele is not a major predisposing factor for severe H1N1pdm09 infection[J]. BMC Res Notes, 2014, 7: 504. DOI:10.1186/1756-0500-7-504 |
| [15] |
Keynan Y, Juno J, Meyers A, et al. Chemokine receptor 5Δ32 allele in patients with severe pandemic (H1N1) 2009[J]. Emerg Infect Dis, 2010, 16(10): 1621-1622. DOI:10.3201/eid1610.100108 |
| [16] |
Ingram VM. A specific chemical difference between the globins of normal human and sickle-cell Anemia Hemoglobin[J]. Nature, 1956, 178(4537): 792-794. DOI:10.1038/178792a0 |
| [17] |
Wu QY, Zhang J, Zhu PR, et al. The susceptibility of FSHB-211G>T and FSHR G-29A, 919A>G, 2039A>G polymorphisms to men infertility:an association study and Meta-analysis[J]. BMC Med Genet, 2017, 18: 81. DOI:10.1186/s12881-017-0441-4 |
 2020, Vol. 41
2020, Vol. 41