文章信息
- 王玉琢, 沈洪兵.
- Wang Yuzhuo, Shen Hongbing
- 孟德尔随机化研究应用于因果推断的影响因素及其结果解读面临的挑战
- Challenges and factors that influencing causal inference and interpretation, based on Mendelian randomization studies
- 中华流行病学杂志, 2020, 41(8): 1231-1236
- Chinese Journal of Epidemiology, 2020, 41(8): 1231-1236
- http://dx.doi.org/10.3760/cma.j.cn112338-20200521-00749
-
文章历史
收稿日期: 2020-05-21
因果与因果推断是流行病学研究的核心问题。观察性流行病学是因果推断中常用的研究方法,但由于潜在混杂因素、反向因果关联等诸多挑战,研究结果往往存在争议[1]。随机对照试验(randomised controlled trial,RCT)是流行病学研究中进行因果推断最为可靠的方法。然而由于RCT试验设计和实施条件要求高、控制严、难度较大,且须考虑医学伦理问题,往往难以直接用来研究疾病的病因[2]。
近年来,孟德尔随机化(Mendelian randomization,MR)方法的提出为基于观察性流行病学研究的因果推断提供了新的途径。MR研究使用与暴露因素具有强相关关系的遗传变异作为工具变量(instrumental variable,Ⅳ),推断暴露因素与结局之间的因果关系。由于配子形成时遵循“亲代等位基因随机分配给子代”的孟德尔遗传规律,遗传变异不会受到环境暴露、社会经济地位、行为因素等传统混杂因素的影响;此外,遗传变异继承自父母,且出生后保持不变,其与结局之间的关联具有时间顺序合理性。因此,MR能够克服传统观察性流行病学研究中存在的混杂和反向因果问题[2]。
随着大样本全基因组关联研究(genome-wide association study,GWAS)数据的累积以及多组学技术的普及,MR研究在因果推断中的应用日益广泛,不仅验证出确证的因果关系(例如LDL-C和肥胖能够增加冠心病发生风险[3-4]),而且针对长期存在的具有争议性的因果关联问题得到了更为可靠的结论(例如低胆固醇水平不会导致癌症[5]、少量饮酒不能预防心血管疾病[6])。此外,MR研究通常能够获得与RCTs相一致的结论,有望为疾病预防和治疗提供理论依据(例如硒元素补充不能预防前列腺癌[7-8],PCSK9抑制剂可降低LDL-C水平以预防冠心病发生[9-10])。然而,MR研究中的工具变量依赖于3个核心假设,当任一假设不成立时,MR研究将难以获得准确的因果关联估计。即使核心假设成立,MR研究在因果推断方面的应用还受到其他局限性的影响。此外,MR研究结果的解读需要综合考虑多方面的因素。本文将围绕MR研究应用于因果推断的影响因素和研究结果的解读做一综述。
一、MR研究的核心假设及其影响因素MR研究选择与暴露因素具有强相关关系的遗传变异作为工具变量,指代待研究的暴露因素,通过分析遗传变异与暴露因素、遗传变异与结局之间的关联,推断暴露因素与结局之间的因果关系。既往文献已详细介绍了MR研究的原理、设计和实施策略,可为研究者开展MR研究提供参考[2]。MR研究中的工具变量须满足3个核心假设(图 1)[11-12]:即①工具变量与暴露因素之间存在稳健的强相关关系(关联性假设);②工具变量与影响“暴露-结局”关系的混杂因素相互独立(独立性假设);③遗传变异只能通过暴露因素影响结局发生,而不能通过其他途径对结局产生作用(排他性假设)。工具变量核心假设的合理性是应用MR研究进行因果推断的前提。然而,弱工具变量(weak instrument)、遗传变异的多效性(horizontal pleiotropy)、连锁不平衡(linkage disequilibrium,LD)、人群分层(population stratification)和Collider偏倚(collider bias)等因素均可能导致工具变量核心假设的违背。
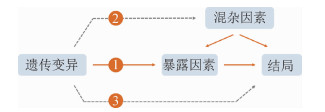
|
| 图 1 孟德尔随机化研究中工具变量的核心假设示意图 |
1.弱工具变量:当遗传变异与暴露因素不具有强相关关系,或者遗传变异仅能解释小部分的表型变异时,研究者称之为“弱工具变量”。弱工具变量的使用在单样本MR(one-sample MR)研究中往往高估暴露因素与结局之间的关联,而在两样本MR(two-sample MR)研究中则会低估关联强度[12]。此外,弱工具变量可导致统计学检验效能的降低,并且违背其他核心假设(例如遗传变异的多效性)所产生的偏倚将可能被放大[12]。为降低弱工具变量导致的偏倚,可采取以下两种策略:一是增加研究样本量。工具变量与暴露因素的关联强度通常使用回归模型中的F统计量加以评估。根据经验,工具变量的F统计量应>10[13]。由于F统计量受样本量的影响,使用公开GWAS或者GWAS汇总数据以增加样本量是减少弱工具变量偏倚的方法之一。二是增加表型解释度。相比于单个遗传变异,多个遗传变异能够解释更大比例的表型变异。通过构建等位基因评分(也称为遗传风险评分)以综合多个遗传变异的效应,将其作为工具变量来预测危险因素的暴露水平,能够增加工具变量所解释的表型变异、降低弱工具变量偏倚[14]。
2.遗传变异的多效性:当遗传变异可通过“遗传变异-暴露因素-结局”以外的其他通路影响结局发生时,该遗传变异即具有多效性,这种多效性可能导致独立性假设和排他性假设的不成立[11]。例如,位于ApoE-CI-CII基因簇的遗传变异rs4420638与血浆C反应蛋白(CRP)水平显著相关,同时rs4420638可影响血浆TC、LDL-C和TG等多个生物标志物的浓度,并且LDL-C是确证的冠心病危险因素[15-16],因此使用rs4420638推断血浆CRP水平与冠心病的因果关联时,该遗传变异对血浆LDL-C浓度的影响可导致MR研究得出错误结论[15]。遗传变异的多效性普遍存在,目前降低遗传变异多效性影响的有效方法之一是使用生物学功能明确的遗传变异作为工具变量[13],例如与血浆CRP浓度直接相关的CRP基因变异[17]、可直接调节血浆LDL-C浓度的前蛋白转化酶枯草溶菌素9(PCSK9)基因无义突变[9]。此外,当使用多个遗传变异作为工具变量时,还可以借助多种统计学方法以检测遗传变异多效性,并校正其对于因果推断的影响。既往文献已对工具变量的多效性偏倚校正方法进行了综述[11]。其中,MR-Egger方法通过对基因-结局与基因-暴露的关联效应拟合回归模型,检验并校正由工具变量多效性所产生的偏倚。如果工具变量不具有定向多效性(directional pleiotropy),模型的截距应为零,因此截距可作为检验统计量以检测工具变量是否存在多效性。MR-Egger回归的斜率则反映了校正多效性所产生的偏倚后,暴露对结局的因果关联效应。MR-Egger回归允许所有遗传变异均具有多效性,但是须满足遗传变异多效性对结局的效应独立于遗传变异对暴露因素的效应(InSIDE)假设。当InSIDE假设成立时,MR-Egger回归可以准确估算因果效应,但是结果的精确度和统计学检验效能将有所降低[18]。此外,加权中位数估计法(weighted median estimator,WME)在50%以下的遗传变异违背MR核心假设的情况下仍然能够准确计算因果关联效应[19]。在MR研究中,可使用多种统计学方法进行敏感性分析,以检测和校正遗传变异的多效性。
3.连锁不平衡:基因组位置相近的遗传变异倾向于共同遗传,这种现象被称为连锁不平衡。假设使用遗传变异A作为工具变量以探索暴露因素M与结局之间的因果关联,遗传变异A与遗传变异B之间具有连锁不平衡关系,并且遗传变异B可通过暴露因素N影响结局发生,这就可能违背MR核心假设从而得出暴露因素M导致结局发生的错误结论。与遗传变异的多效性相似,使用生物学功能明确的遗传变异作为工具变量,或者借助统计学方法能够有效控制连锁不平衡造成的偏倚[13]。近年来,随着高通量测序技术的普及,越来越多的研究开始整合基因组和转录组测序数据以更好的理解复杂性疾病发生的分子机制。在此背景下,Giambartolomei等[20]提出了贝叶斯共定位分析方法,通过检测待研究分子表型与结局是否具有相同的因果变异(causal variant),以推测遗传变异是否通过待研究分子表型导致结局发生。
4.人群分层:是指遗传变异频率在不同遗传背景的人群间存在差异,导致遗传变异与结局之间出现虚假关联。在MR研究中,人群分层可导致独立性假设或排他性假设不成立,继而得出错误的因果推断[21]。例如Haworth等[21]通过UK Biobank数据发现遗传变异和主要健康结局均与出生地有关;如果不能很好地校正人群分层,则会导致遗传变异与主要健康结局之间的虚假关联。纳入具有相同遗传背景的人群开展遗传关联研究是规避人群分层偏倚最直接的方式。然而,由于传统GWAS的统计学检验效能偏低,扩大样本量开展多中心GWAS已经成为趋势。在此情况下,将主成份纳入统计学模型或者使用线性混合模型进行关联分析能够有效校正人群分层对于关联结果的影响[22-23]。
5. Collider偏倚:当暴露和结局能够独立影响某个危险因素时,调整该危险因素可能产生Collider偏倚。例如,慢性非传染性疾病(例如心脑血管疾病和恶性肿瘤)的发病年龄较高,针对此类疾病的遗传关联研究所招募的研究对象大多为高龄人群,这可能会违背独立性假设,致使MR研究的因果关联估计产生偏倚。假设一项MR研究是基于年龄>80岁的人群,该研究使用APOE遗传变异作为血浆胆固醇水平的工具变量,评估胆固醇水平与心肌梗死之间的因果关系。由于胆固醇水平能够影响人体寿命,并且寿命可同时受到其他因素(例如吸烟)的影响,纳入年龄>80岁的人群将会导致APOE遗传变异与吸烟之间产生虚假“关联”,违背独立性假设。换言之,假设危险等位基因G可通过调节胆固醇水平而增加个体死亡风险,那么在存活到>80岁的高龄人群中,携带该危险等位基因的人群具有其他导致死亡风险增高危险因素(例如更低的吸烟率或吸烟剂量)的可能性将低于不携带危险等位基因的人群。如果吸烟亦能够增加心肌梗死的发生风险,那么将会导致遗传变异与结局之间因果关联的估计值产生偏倚[24]。此类由高龄人群为主要研究对象所导致的偏倚亦被称为“生存者偏倚(survivor bias)”,是Collider偏倚中较为常见的一类。近年来,研究者提出了一系列用以校正生存者偏倚的统计学模型,包括逆概率加权法(inverse probability weighting)、半参数相加风险模型等[25-28]。其中逆概率加权法根据受试者纳入研究的概率进行逆向加权(例如当具有某种特征的人群被纳入研究的概率较低,则使用逆概率加权法给予该人群更大的权重),使得研究人群的特征变量分布与源人群相似[26]。
二、MR研究的局限性在工具变量核心假设成立的基础上,一些其他因素如Beavis效应、发育代偿(canalization/developmental compensation)机制、低统计学检验效能(low power)、生物学复杂性(complexity of biology)和表型异质性(trait heterogeneity)等亦能够影响MR研究在因果推断中的应用[13, 29]。
1. Beavis效应:疾病或性状相关的基因区域通常包含数十个乃至上百个满足全基因组显著性标准的遗传变异,GWAS仅报道其中关联性最为显著的遗传变异,可能高估遗传变异与暴露因素之间的关联,该现象亦被称为“胜利者的诅咒(winner’s curse)”,通常由遗传变异与潜在混杂因素之间的关联所导致。如果MR研究通过设定显著性标准从GWAS中筛选工具变量,则Beavis效应可导致基因-暴露之间的效应被高估,进而MR研究的因果关联效应被低估[29-30]。在单样本MR研究中可以使用多个遗传变异的简单相加等位基因评分进行敏感性分析,评估Beavis效应对因果关联估计的影响;在两样本MR研究中,使用验证样本的基因-暴露效应进行因果关联估算能够减少Beavis效应所导致的偏倚[13]。
2.发育代偿机制:个体在发育过程中可能对遗传变异所导致的表型改变启动代偿性反应,使得暴露与结局之间的关联被低估。表达于胚胎发育期的蛋白质编码基因区域的功能性遗传变异较易受到发育代偿机制的影响;而针对出生后环境暴露或生活习惯的MR研究则较少受到发育代偿机制的影响[30]。目前尚没有方法能够评估或校正发育代偿机制对MR研究因果关联估计的影响。深入理解发育过程中的基因表达或调控机制可能是评估发育代偿机制偏倚的必要前提[13]。
3.生物学复杂性:虽然GWAS在复杂性疾病和性状的遗传易感性研究方面取得了巨大成功,但是其所发现的大多数遗传变异的生物学机制尚不清楚,这将为MR研究的因果推断带来一定的挑战性。例如,表达于肝脏细胞的清道夫受体B1(SR-B1)可促进肝脏选择性吸收循环HDL-C,在胆固醇逆向转运中发挥生物学作用[31]。胆固醇逆向转运能够减轻动脉粥样化负担,从而降低冠心病发生风险[32]。SR-B1编码基因的罕见变异SCARB1 P376L能够导致SR-B1功能丢失[33],使得携带该遗传变异的个体具有更高的血浆HDL-C水平,同时动脉粥样化负担增加导致冠心病发生风险增高。因此,该研究结果不能被简单地解释为“循环HDL-C水平增高导致冠心病发生”。综合生物信息学分析和功能实验等方法深入解析遗传变异的生物学机制将有助于合理解读MR研究结果。
4.表型异质性:某些遗传变异能够影响暴露因素的多个方面,使用这些遗传变异作为工具变量不影响暴露因素整体与结局之间的因果推断,但却难以推断暴露因素的某个方面与结局之间的因果关系[13]。例如,15号染色体尼古丁乙酰胆碱受体基因区域(CHRNA5-A3-B4)的遗传变异rs1051730能够影响吸烟行为的多个方面,包括吸烟深度、每日吸烟量、吸烟年数等,这些因素均能影响个体的烟草暴露水平[13, 34]。因此rs1051730可用作工具变量以推断吸烟行为与结局之间的因果关联,但无法推断每日吸烟量、吸烟年数与结局之间的因果关系。与生物学复杂性相似,深入理解遗传变异的生物学机制可能有助于推断暴露因素的某个方面与结局之间的因果关系。
三、MR研究结果解读基于MR研究的因果关联证据的可靠性介于观察性流行病学研究和实验流行病学研究之间(图 2)[35]。相较于传统的观察性流行病学研究,一项设计严谨、核心假设成立的MR研究通常可以提供更为可靠的证据以指导RCT的开展,并且可以在RCT(例如由于伦理学原因)无法实施的情况下提供有力证据[12]。由于任何单一的证据来源都不足以建立确证的因果关系,MR研究结果的解读还应基于“综合证据(triangulation of evidence)”:即针对某个因果关联问题,通过对多项研究(采用不同的研究设计)结果的整合获取更可靠的结论[36]。尽管每项研究均可能存在偏倚,但是单项研究中的偏倚对关联效应大小乃至方向的影响独立于其他研究,因此多项研究结果的一致性程度越高,研究结论就越可靠[36]。
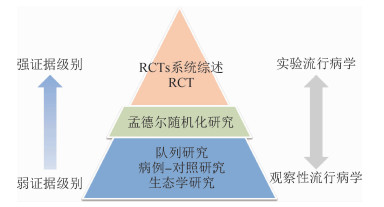
|
| 图 2 研究设计与证据分级 |
MR研究成果能否转化应用于临床和公共卫生实践仍需结合实际问题进行评估。例如,从疾病预防的角度,医务工作者需要知晓重度吸烟(吸烟包年≥30)的男性人群如果从55岁开始戒烟,10年后发生肺癌的绝对风险降低程度。然而,MR研究仅能提供危险因素的终身暴露对结局发生的效应,难以评估特定期限内危险因素暴露的改变所产生的效应。此外,某些暴露因素对结局的效应具有时间依赖性(time-dependent causal effects)。例如,维生素D可降低多发性硬化症发生风险,但这种保护效应局限于儿童期和青春期[37]。虽然MR研究能够发现维生素D降低多发性硬化症发病风险的因果关联[38],但是在关键时期(儿童期和青春期)之后补充维生素D可能无法降低多发性硬化症的发生风险。因此,后续如果使用RCT来验证维生素D补充能否降低多发性硬化症的发生风险,需招募处于儿童期的高风险人群(可通过家族史或遗传风险评分识别)[12]。
最后,MR研究在疾病预后相关因果推断中的应用较为有限。例如CHRNA5基因的遗传变异与吸烟成瘾和肺癌发生风险增加有关,携带该基因遗传变异的肺癌患者确诊后减少吸烟或戒烟的难度更大[39]。然而,CHRNA5基因遗传变异不能作为工具变量用以推断减少吸烟或戒烟对肺癌患者预后的影响,因为该基因遗传变异与肺癌患者预后之间的关联反映了个体终生烟草暴露的效应,而不是确诊后吸烟状态的改变对于肺癌预后的影响[40]。更重要的是,疾病发生和发展(或预后)可能具有不同的影响因素。例如,叶酸在前列腺癌和结肠直肠癌的发生和发展中发挥双重作用:在肿瘤发生前保护DNA免受损伤,但在肿瘤发生后通过增强肿瘤增殖和侵袭促进肿瘤进展[41-42]。这意味着MR的开展需基于疾病进展或预后研究(而不是疾病易感性研究),以提供与疾病治疗直接相关的证据[40]。
四、总结MR研究能够有效克服混杂和反向因果问题所带来的偏倚,为推断暴露因素与结局之间的因果关系提供可靠的证据。然而,MR研究中的工具变量依赖于3个核心假设,当3个核心假设均成立时,MR研究才能获得可靠的因果推断。MR研究结果的解读需要考虑多方面的因素,包括MR研究设计是否合理、工具变量是否正确、统计学检验效能是否充足、以及生物学复杂性是否会使结果的解读复杂化。在以上条件均满足的基础上,MR研究结果的解读仍应基于“综合证据”,多项研究结果的一致性程度越高,研究结论就越可靠。总之,MR研究能够为暴露因素与结局之间的因果推断提供有力证据,研究发现有望指导临床试验和药物开发,为临床和公共卫生决策提供理论依据[43-44]。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Smith GD, Ebrahim S. Data dredging, bias, or confounding[J]. BMJ, 2002, 325(7378): 1437-1438. DOI:10.1136/bmj.325.7378.1437 |
| [2] |
王莉娜, Zhang ZF. 孟德尔随机化法在因果推断中的应用[J]. 中华流行病学杂志, 2017, 38(4): 547-552. Wang LN, Zhang ZF. Mendelian randomization approach, used for causal inferences[J]. Chin J Epidemiol, 2017, 38(4): 547-552. DOI:10.3760/cma.j.issn.0254-6450.2017.04.027 |
| [3] |
Ference BA, Yoo W, Alesh I, et al. Effect of long-term exposure to lower low-density lipoprotein cholesterol beginning early in life on the risk of coronary heart disease:a Mendelian randomization analysis[J]. J Am Coll Cardiol, 2012, 60(25): 2631-2639. DOI:10.1016/j.jacc.2012.09.017 |
| [4] |
Larsson SC, Back M, Rees JMB, et al. Body mass index and body composition in relation to 14 cardiovascular conditions in UK Biobank:a Mendelian randomization study[J]. Eur Heart J, 2020, 41(2): 221-226. DOI:10.1093/eurheartj/ehz388 |
| [5] |
Benn M, Tybjaerg-Hansen A, Stender S, et al. Low-density lipoprotein cholesterol and the risk of cancer:a Mendelian randomization study[J]. J Natl Cancer Inst, 2011, 103(6): 508-519. DOI:10.1093/jnci/djr008 |
| [6] |
Millwood IY, Walters RG, Mei XW, et al. Conventional and genetic evidence on alcohol and vascular disease aetiology:a prospective study of 500000 men and women in China[J]. Lancet, 2019, 393(10183): 1831-1842. DOI:10.1016/S0140-6736(18)31772-0 |
| [7] |
Lippman SM, Klein EA, Goodman PJ, et al. Effect of selenium and vitamin E on risk of prostate cancer and other cancers:the Selenium and Vitamin E Cancer Prevention Trial (SELECT)[J]. JAMA, 2009, 301(1): 39-51. DOI:10.1001/jama.2008.864 |
| [8] |
Yarmolinsky J, Bonilla C, Haycock PC, et al. Circulating selenium and prostate cancer risk:a Mendelian randomization analysis[J]. J Natl Cancer Inst, 2018, 110(9): 1035-1038. DOI:10.1093/jnci/djy081 |
| [9] |
Cohen JC, Boerwinkle E, Mosley TH Jr, et al. Sequence variations in PCSK9, low LDL, and protection against coronary heart disease[J]. N Engl J Med, 2006, 354(12): 1264-1272. DOI:10.1056/NEJMoa054013 |
| [10] |
Sabatine MS, Giugliano RP, Keech AC, et al. Evolocumab and clinical outcomes in patients with cardiovascular disease[J]. N Engl J Med, 2017, 376(18): 1713-1722. DOI:10.1056/NEJMoa1615664 |
| [11] |
高雪, 王慧, 王彤. 孟德尔随机化中多效性偏倚校正方法简介[J]. 中华流行病学杂志, 2019, 40(3): 360-365. Gao X, Wang H, Wang T. Review on correction methods related to the pleiotropic effect in Mendelian randomization[J]. Chin J Epidemiol, 2019, 40(3): 360-365. DOI:10.3760/cma.j.issn.0254-6450.2019.03.020 |
| [12] |
Davies NM, Holmes MV, Smith GD. Reading Mendelian randomisation studies:a guide, glossary, and checklist for clinicians[J]. BMJ, 2018, 362: k601. DOI:10.1136/bmj.k601 |
| [13] |
Zheng J, Baird D, Borges MC, et al. Recent developments in Mendelian randomization studies[J]. Curr Epidemiol Rep, 2017, 4(4): 330-345. DOI:10.1007/s40471-017-0128-6 |
| [14] |
Burgess S, Thompson SG. Use of allele scores as instrumental variables for Mendelian randomization[J]. Int J Epidemiol, 2013, 42(4): 1134-1144. DOI:10.1093/ije/dyt093 |
| [15] |
Elliott P, Chambers JC, Zhang WH, et al. Genetic Loci associated with C-reactive protein levels and risk of coronary heart disease[J]. JAMA, 2009, 302(1): 37-48. DOI:10.1001/jama.2009.954 |
| [16] |
Pagidipati NJ, Navar AM, Mulder H, et al. Comparison of recommended eligibility for primary prevention statin therapy based on the us preventive services task force recommendations vs the ACC/AHA guidelines[J]. JAMA, 2017, 317(15): 1563-1567. DOI:10.1001/jama.2017.3416 |
| [17] |
Wensley F, Gao P, Burgess S, et al. Association between C reactive protein and coronary heart disease:Mendelian randomisation analysis based on individual participant data[J]. BMJ, 2011, 342: d548. DOI:10.1136/bmj.d548 |
| [18] |
Bowden J, Del Greco MF, Minelli C, et al. Assessing the suitability of summary data for two-sample Mendelian randomization analyses using MR-Egger regression:the role of the I2 statistic[J]. Int J Epidemiol, 2016, 45(6): 1961-1974. DOI:10.1093/ije/dyw220 |
| [19] |
Bowden J, Smith GD, Haycock PC, et al. Consistent estimation in Mendelian randomization with some invalid instruments using a weighted median estimator[J]. Genet Epidemiol, 2016, 40(4): 304-314. DOI:10.1002/gepi.21965 |
| [20] |
Giambartolomei C, Vukcevic D, Schadt EE, et al. Bayesian test for colocalisation between pairs of genetic association studies using summary statistics[J]. PLoS Genet, 2014, 10(5): e1004383. DOI:10.1371/journal.pgen.1004383 |
| [21] |
Haworth S, Mitchell R, Corbin L, et al. Apparent latent structure within the UK Biobank sample has implications for epidemiological analysis[J]. Nat Commun, 2019, 10(1): 333. DOI:10.1038/s41467-018-08219-1 |
| [22] |
Price AL, Patterson NJ, Plenge RM, et al. Principal components analysis corrects for stratification in genome-wide association studies[J]. Nat Genet, 2006, 38(8): 904-909. DOI:10.1038/ng1847 |
| [23] |
Loh PR, Tucker G, Bulik-Sullivan BK, et al. Efficient Bayesian mixed-model analysis increases association power in large cohorts[J]. Nat Genet, 2015, 47(3): 284-290. DOI:10.1038/ng.3190 |
| [24] |
Boef AG, Le Cessie S, Dekkers OM. Mendelian randomization studies in the elderly[J]. Epidemiology, 2015, 26(2): e15-16. DOI:10.1097/EDE.0000000000000243 |
| [25] |
Smit RAJ, Trompet S, Dekkers OM, et al. Survival bias in mendelian randomization studies:a threat to causal inference[J]. Epidemiology, 2019, 30(6): 813-816. DOI:10.1097/EDE.0000000000001072 |
| [26] |
Gkatzionis A, Burgess S. Contextualizing selection bias in Mendelian randomization:how bad is it likely to be?[J]. Int J Epidemiol, 2019, 48(3): 691-701. DOI:10.1093/ije/dyy202 |
| [27] |
Vansteelandt S, Dukes O, Martinussen T. Survivor bias in Mendelian randomization analysis[J]. Biostatistics, 2018, 19(4): 426-443. DOI:10.1093/biostatistics/kxx050 |
| [28] |
Carlin CS, Solid CA. An approach to addressing selection bias in survival analysis[J]. Stat Med, 2014, 33(23): 4073-4086. DOI:10.1002/sim.6211 |
| [29] |
Lawlor DA. Commentary:Two-sample Mendelian randomization:opportunities and challenges[J]. Int J Epidemiol, 2016, 45(3): 908-915. DOI:10.1093/ije/dyw127 |
| [30] |
Haycock PC, Burgess S, Wade KH, et al. Best (but oft-forgotten) practices:the design, analysis, and interpretation of Mendelian randomization studies[J]. Am J Clin Nutr, 2016, 103(4): 965-978. DOI:10.3945/ajcn.115.118216 |
| [31] |
Zhang Y, Da Silva JR, Reilly M, et al. Hepatic expression of scavenger receptor class B typeⅠ(SR-BⅠ) is a positive regulator of macrophage reverse cholesterol transport in vivo[J]. J Clin Invest, 2005, 115(10): 2870-2874. DOI:10.1172/JCI25327 |
| [32] |
Rosenson RS, Brewer HB Jr, Davidson WS, et al. Cholesterol efflux and atheroprotection:advancing the concept of reverse cholesterol transport[J]. Circulation, 2012, 125(15): 1905-1919. DOI:10.1161/CIRCULATIONAHA.111.066589 |
| [33] |
Zanoni P, Khetarpal SA, Larach DB, et al. Rare variant in scavenger receptor BI raises HDL cholesterol and increases risk of coronary heart disease[J]. Science, 2016, 351(6278): 1166-1171. DOI:10.1126/science.aad3517 |
| [34] |
Taylor AE, Davies NM, Ware JJ, et al. Mendelian randomization in health research:using appropriate genetic variants and avoiding biased estimates[J]. Econ Hum Biol, 2014, 13: 99-106. DOI:10.1016/j.ehb.2013.12.002 |
| [35] |
Zuccolo L, Holmes MV. Commentary:Mendelian randomization-inspired causal inference in the absence of genetic data[J]. Int J Epidemiol, 2017, 46(3): 962-965. DOI:10.1093/ije/dyw327 |
| [36] |
Lawlor DA, Tilling K, Smith GD. Triangulation in aetiological epidemiology[J]. Int J Epidemiol, 2016, 45(6): 1866-1886. DOI:10.1093/ije/dyw314 |
| [37] |
Dean G, Elian M. Age at immigration to England of Asian and Caribbean immigrants and the risk of developing multiple sclerosis[J]. J Neurol Neurosurg Psychiatry, 1997, 63(5): 565-568. DOI:10.1136/jnnp.63.5.565 |
| [38] |
Mokry LE, Ross S, Ahmad OS, et al. Vitamin D and risk of multiple sclerosis:a Mendelian randomization study[J]. PLoS Med, 2015, 12(8): e1001866. DOI:10.1371/journal.pmed.1001866 |
| [39] |
Thorgeirsson TE, Geller F, Sulem P, et al. A variant associated with nicotine dependence, lung cancer and peripheral arterial disease[J]. Nature, 2008, 452(7187): 638-642. DOI:10.1038/nature06846 |
| [40] |
Paternoster L, Tilling K, Smith GD. Genetic epidemiology and Mendelian randomization for informing disease therapeutics:Conceptual and methodological challenges[J]. PLoS Genet, 2017, 13(10): e1006944. DOI:10.1371/journal.pgen.1006944 |
| [41] |
Rycyna KJ, Bacich DJ, O'Keefe DS. Opposing roles of folate in prostate cancer[J]. Urology, 2013, 82(6): 1197-1203. DOI:10.1016/j.urology.2013.07.012 |
| [42] |
Kim YI. Role of folate in colon cancer development and progression[J]. J Nutr, 2003, 133(11): 3731S-3739. DOI:10.1093/jn/133.11.3731S |
| [43] |
Davey Smith G, Paternoster L, Relton C. When will mendelian randomization become relevant for clinical practice and public health?[J]. JAMA, 2017, 317(6): 589-591. DOI:10.1001/jama.2016.21189 |
| [44] |
Walker VM, Smith GD, Davies NM, et al. Mendelian randomization:a novel approach for the prediction of adverse drug events and drug repurposing opportunities[J]. Int J Epidemiol, 2017, 46(6): 2078-2089. DOI:10.1093/ije/dyx207 |
 2020, Vol. 41
2020, Vol. 41


