文章信息
- 杜志成, 顾菁, 李菁华, 林晓, 王莹, 陈龙, 郝元涛.
- Du Zhicheng, Gu Jing, Li Jinghua, Lin Xiao, Wang Ying, Chen Long, Hao Yuantao
- 基于区间删失数据估计方法的COVID-19潜伏期分布估计
- Estimating the distribution of COVID-19 incubation period by interval-censored data estimation method
- 中华流行病学杂志, 2020, 41(7): 1000-1003
- Chinese Journal of Epidemiology, 2020, 41(7): 1000-1003
- http://dx.doi.org/10.3760/cma.j.cn112338-20200313-00331
-
文章历史
收稿日期: 2020-03-13
2. 中山大学全球卫生研究中心, 广州 510275;
3. 广东省卫生健康委员会政务服务中心, 广州 510060
2. Sun Yat-sen Global Health Institute, Sun Yat-sen University, Guangzhou 510275, China;
3. Government Affairs Service Center, Health Commission of Guangdong Province, Guangzhou 510060, China
自2019年12月武汉市发现不明原因肺炎病例以来,截至1月31日国内累计报告确诊病例数已超1万例[1],并且被WHO定为国际突发公共卫生紧急事件(Public health emergency of international concern,PHEIC)[2]。截至2020年3月11日,国内累计确诊病例数已超过8万例[1],新型冠状病毒肺炎(COVID-19)疫情已影响了海外100多个国家[3],WHO也将其定性为全球大流行[4]。
疫情发展至今,人们对COVID-19的流行病学特征的了解仍然有限。WHO在COVID-19全球研究路线报告中提到尚待研究的流行病学问题包括[5]:有无症状感染与传播率的关系?不同年龄人群传播率的差异?精确的基本再生数?流行病学时间延迟(如潜伏期和发病到确诊时长等)?等。其中,潜伏期是传染病的一个重要流行病学特征[6],而且常常是其他重要参数估计的前提。基本再生数的估计需要可靠的潜伏期信息支撑,如利用指数增长法和极大似然法等计算基本再生数时必须提供潜伏期的分布[7]。传染病动力学模型的构建也需要可靠的潜伏期信息支撑[8]。但对COVID-19潜伏期的认识仍存在争议。我国新型冠状病毒肺炎诊疗指南(试行第七版)中提出,基于目前的流行病学调查,潜伏期为1.0~14.0 d,多为3.0~7.0 d[9],说明当前对于潜伏期的点估计(与区间估计对应)仍然存在不确定性。部分研究报道的COVID-19潜伏期M=4.0~5.1 d[10-11],但因很多研究是按照暴露窗口期的最后1 d估计潜伏期,可能存在所估计潜伏期偏短的风险[10]。本研究基于官方公开的病例信息,利用区间删失数据的统计模型,克服人为确定窗口期的风险,对COVID-19的潜伏期进行点估计及分布估计。
资料与方法1.数据收集:根据各省市卫生健康委员会官方发布媒体(含网站、微信公众号、微博等信息平台)报告的数据,收集确诊病例的暴露与发病信息。纳入标准:①具有明确发病日期(含时点和区间)信息;②具有明确暴露窗口期(含时点和区间)信息。排除标准:湖北省常住人口。本研究收集与记录的信息包括报告省份、报告城市、年龄、性别、暴露窗口期、发病日期和来源链接等,例如北京市卫生健康委员会2020年1月21日报道的“37岁男性患者,于1月10日赴武汉出差,1月11日返回北京,1月14日出现发热症状”[12]。
2.统计学方法:采用Excel 2016软件进行数据库整理,采用R 3.6.3软件进行数据处理。根据区间删失数据估计法对COVID-19的潜伏期进行点估计及分布估计。
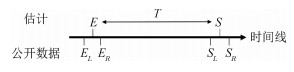
假设某病例的潜伏期已知,其暴露时点为E(infecting exposure),症状出现时点为S(symptom onset),那么潜伏期T(true incubation period)为S-E。本研究收集的公开数据中,E和S都落在一个有限的区间中,即区间删失(interval-censored)[13]。区间的左右端分别以L(left)和R(right)表示,那么T、E、S、EL、ER、SL、SR的关系可表示为:
T为非负的连续随机变量,常见的分布为Log-normal、Gamma和Weibull分布,设其概率密度函数为fθ(t),θ为函数的参数;同理,设E和S的概率密度函数分别为hλ(e)和g(s)。由T、E、S的关系可得,g(s∣e)=fθ(s-e∣e)=fθ(s-e)。那么,T的似然函数(likelihood)可表示为:
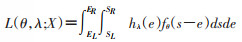
式中,X为观测数据(EL、ER、SL、SR)。由此,n个病例T的似然函数可表示为:
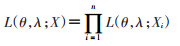
对上式利用极大似然函数法即可求出潜伏期T所服从分布的参数θ。
本研究基于区间删失数据可利用加速时间失效模型(accelerated failure time model,AFT)来刻画潜伏期T[14],不考虑协变量的AFT模型如下:

式中,Y为T的对数线性形式,μ为截距,σ为残差W的系数。潜伏期T常见的分布有对数正态分布(Log-normal)、伽马分布(Gamma)和韦伯分布(Weibull)[15-17],通过T的概率密度函数fθ(t),以及T~Log-normal(μ,σ2)、T~Gamma(α,β)和T~Weibull(k,λ),可得:
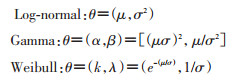
1.基本特征:本研究共收集确诊病例109例,平均年龄为39.825岁,男性占59.63%,记录的暴露窗口期范围为2020年1月3-29日,记录的发病日期范围为2020年1月8日至2月5日。纳入省份中,例数较多为广东省(54例)、北京市(10例)和河南省(7例)。见表 1。
2.基于不同分布模型概率密度函数参数(θ):Log-normal分布的均数μ=1.616和标准差σ=0.541,Gamma分布的形状参数α=3.831和尺度参数β=1.517,Weibull分布的形状参数k=2.071和比例参数λ=6.622。Gamma分布的对数似然函数值最大=-150.867。上述各个参数的95%CI范围均较小,且对数似然函数值相差也较小。见表 2。
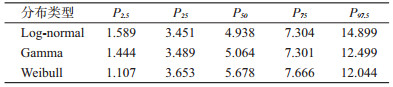
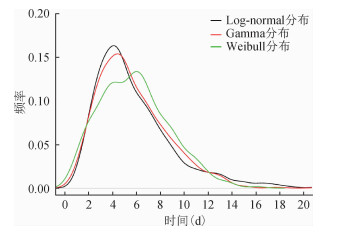
3.基于不同分布模型参数的潜伏期:基于Log- normal和Gamma分布的潜伏期分布基本一致,且与基于Weibull分布的潜伏期分布差别相对较大,但总体趋势一致;最长潜伏期可超过20 d,但概率基本接近于0(图 2)。基于Log-normal分布的潜伏期M=4.938(P25~P75:3.451~7.304)d,基于Gamma分布的潜伏期M=5.064(P25~P75:3.489~7.301)d,基于Weibull分布的潜伏期M=5.678(P25~P75:3.653~7.666)d(表 3)。
|
| 图 1 基于区间数据的潜伏期示意图 |
|
| 图 2 基于不同分布类型估计的潜伏期频率分布 |
本研究基于确诊病例的暴露与发病信息,利用区间删失数据估计方法,对COVID-19的潜伏期分布进行估计,得到COVID-19的潜伏期符合Gamma分布,其潜伏期M=5.064(P25~P75:3.489~7.301)d,且Gamma分布的形状参数α=3.831和尺度参数β=1.517。采用不同分布类型计算的各参数95%CI范围均较小,对数似然函数值相差也较小,说明结果估计较为稳健。
不同研究报道的COVID-19中位潜伏期存在一定差别。本研究估计的潜伏期M=5.064 d,介于过往研究报道的4.0~6.4 d区间内[10-11, 18]。部分研究采用平均值指标得出COVID-19的平均潜伏期为5.0~5.2 d[19-20],但COVID-19的潜伏期服从偏态分布,平均值指标的可靠性有待商榷。
不同研究估计COVID-19潜伏期的有效样本量有限,差别也较大。过往研究的有效量本量为10~291例[10, 18-20],本研究估计潜伏期所利用的有效样本量为109例,处于中等水平。潜伏期分布估计时应该排除处于高发期的疫源地常住居民,以排除不明确的暴露史,但既往部分研究仍纳入了武汉市常住居民作为研究对象[18, 20],存在着暴露窗口期难以确定的风险。另外,与Guan等[10]对来自全国30个省份552家医院2019年12月11日至2020年1月29日的1 099例确诊病例的年龄M=47(P25~P75:35~58)岁相比,本研究所收集病例的平均年龄为39.825岁,处于前者四分位数间距的中等偏低水平,提示疫情早期有明确接触史的确诊病例人群较全人群年轻。
本研究通过比较Log-normal、Weibull和Gamma分布得到,Gamma分布具有较大的对数似然函数值,为最优分布。而Backer等[18]比较3个分布的留一法信息准则(leave-one-out information criterion)得到Weibull分布的拟合效果最好,而Linton等[20]比较3个分布的加权赤池信息准则(weighted Akaike information criterion)得到Log-normal分布最优。可能原因为3个分布类型近似,综合考虑有效样本量和样本代表性,以及上述研究并未提供分布的具体参数,还需后续研究对潜伏期分布进一步深入探讨。
本研究存在局限性。第一,本研究收集的数据均来自官方公布的流行病学调查数据,具有权威性,但信息来源于对确诊病例的问询,存在回忆偏倚,而本研究利用区间删失数据估计方法在一定程度上克服了回忆具体时点带来的偏差。第二,本研究收集的样本量有限,后续研究可在增加有效可靠样本的情况下进行估计。第三,潜伏期在不同的阶段(如疫情周期)以及环境(如人群、社会经济、生态环境等)中可能存在差异,本研究因样本量有限并未进行亚组估计。
综上所述,本研究发现Gamma分布可能是COVID-19潜伏期的最优分布,基于区间删失数据的估计方法可用于传染病潜伏期分布的估计,尽早明确COVID-19的潜伏期有助于以便制定更有效的疫情防控策略。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
国家卫生健康委员会.新型冠状病毒感染的肺炎疫情最新情况[EB/OL]. (2020-03-12)[2020-03-13]. http://www.nhc.gov.cn/wjw/rdts/list.shtml. National Health Commission. The latest situation of COVID-19[EB/OL]. (2020-03-12)[2020-03-03]. http://www.nhc.gov.cn/wjw/rdts/list.shtml. |
| [2] |
Magnusson R. Advancing the right to health:the vital role of law[M]. Geneva: WHO, 2017.
|
| [3] |
World Health Organization. Coronavirus disease (COVID-19) outbreak[EB/OL]. (2020-03-12)[2020-03-12]. https://www.who.int/emergencies/diseases/novel-coronavirus-2019.
|
| [4] |
World Health Organization. WHO Director-General's opening remarks at the media briefing on COVID-19-11 March 2020[EB/OL]. (2020-03-11)[2020-03-12]. https://www.who.int/dg/speeches/detail/who-director-general-s-opening-remarks-at-the-media-briefing-on-covid-19-11-march-2020.
|
| [5] |
World Health Organization. Coronavirus disease (COVID-2019) R&D[EB/OL]. (2020-02-12)[2020-03-12]. https://www.who.int/blueprint/priority-diseases/key-action/novel-coronavirus/en/.
|
| [6] |
Sartwell PE. The distribution of incubation periods of infectious disease[J]. Am J Epidemiol, 1995, 141(5): 386-394. DOI:10.1093/oxfordjournals.aje.a119397 |
| [7] |
王莹, 尤心怡, 王奕婧, 等. 中国新型冠状病毒肺炎疫情再生系数评估[J]. 中华流行病学杂志, 2020, 41(4): 476-479. Wang Y, You XY, Wang YJ, et al. Estimating the basic reproduction number of COVID-19 in Wuhan, China[J]. Chin J Epidemiol, 2020, 41(4): 476-479. DOI:10.3760/cma.j.cn112338-20200210-00086 |
| [8] |
魏永越, 卢珍珍, 杜志成, 等. 基于改进的SEIR+CAQ传染病动力学模型进行新型冠状病毒肺炎疫情趋势分析[J]. 中华流行病学杂志, 2020, 41(4): 470-475. Wei YY, Lu ZZ, Du ZC, et al. Fitting and forecasting the trend of COVID-19 by SEIR+CAQ dynamic model[J]. Chin J Epidemiol, 2020, 41(4): 470-475. DOI:10.3760/cma.j.cn112338-20200216-00106 |
| [9] |
国家卫生健康委办公厅.关于印发新型冠状病毒肺炎诊疗方案(试行第七版)的通知[EB/OL]. (2020-03-03)[2020-03-12]. http://www.nhc.gov.cn/yzygj/s7653p/202003/46c9294a7dfe4cef80dc7f5912eb1989.shtml. General Office of National Health Commission. Notification on the issuance of the diagnosis and treatment protocol COVID-19(pilot 7th edition)[EB/OL]. (2020-03-03)[2020-03-12]. http://www.nhc.gov.cn/yzygj/s7653p/202003/46c9294a7dfe4cef80dc7f5912eb1989.shtml. |
| [10] |
Guan W, Ni Z, Hu Y, et al. Clinical Characteristics of Coronavirus Disease 2019 in China[J]. NEJM, 2020. DOI:10.1056/NEJMoa2002032 |
| [11] |
Lauer SA, Grantz KH, Bi Q, et al. The Incubation Period of Coronavirus Disease 2019(COVID-19) From Publicly Reported Confirmed Cases:Estimation and Application[J]. Ann Intern Med, 2020. DOI:10.7326/M20-0504 |
| [12] |
北京市卫生健康委员会.我市新增5例新型冠状病毒感染的肺炎病例[EB/OL]. (2020-01-21)[2020-03-12]. http://wjw.beijing.gov.cn/xwzx_20031/wnxw/202001/t20200121_1620353.html. Health Commission of Beijing. Five new cases of COVID-19 in Beijing[EB/OL]. (2020-01-21)[2020-03-12]. http://wjw.beijing.gov.cn/xwzx_20031/wnxw/202001/t20200121_1620353.html. |
| [13] |
Zhang Z, Sun J. Interval censoring[J]. Stat Methods Med Res, 2010, 19(1): 53-70. DOI:10.1177/0962280209105023 |
| [14] |
Swindell WR. Accelerated failure time models provide a useful statistical framework for aging research[J]. EXP GERONTOL, 2009, 44(3): 190-200. DOI:10.1016/j.exger.2008.10.005 |
| [15] |
Blackwood LG. The lognormal distribution, environmental data, and radiological monitoring[J]. Environ Monit Assess, 1992, 21(3): 193-210. DOI:10.1007/BF00399687 |
| [16] |
Einberg I, Thode HJ, Chugani HT, et al. Gamma distribution model describes maturational curves for delta wave amplitude, cortical metabolic rate and synaptic density[J]. J Theor Biol, 1990, 142(2): 149-161. DOI:10.1016/s0022-5193(05)80218-8 |
| [17] |
Verma A, Narula A, Katyal A, et al. Failure rate prediction of equipment:can Weibull distribution be applied to automated hematology analyzers?[J]. Clin Chem Lab Med, 2018, 56(12): 2067-2071. DOI:10.1515/cclm-2018-0569 |
| [18] |
Backer JA, Klinkenberg D, Wallinga J. Incubation period of 2019 novel coronavirus (2019-nCoV) infections among travellers from Wuhan, China, 20-28 January 2020[J]. Euro Survel, 2020, 25(5): 2000062. DOI:10.2807/1560-7917.ES.2020.25.5.2000062 |
| [19] |
Li Q, Guan X, Wu P, et al. Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus-Infected Pneumonia[J]. NEJM, 2020. DOI:10.1056/NEJMoa2001316 |
| [20] |
Linton NM, Kobayashi T, Yang Y, et al. Incubation Period and Other Epidemiological Characteristics of 2019 Novel Coronavirus Infections with Right Truncation:A Statistical Analysis of Publicly Available Case Data[J]. J Clin Med, 2020, 9(2): 538. DOI:10.3390/jcm9020538 |
 2020, Vol. 41
2020, Vol. 41