文章信息
- 巫晶晶, 黄鹏, 岳明, 汪春晖, 吴超, 邵建国, 薛红, 符祖强, 卓凌云, 喻荣彬, 张云.
- Wu Jingjing, Huang Peng, Yue Ming, Wang Chunhui, Wu Chao, Shao Jianguo, Xue Hong, Fu Zuqiang, Zhuo Lingyun, Yu Rongbin, Zhang Yun.
- TNFRSF11A和TNFRSF11B基因多态性与HCV感染转归的关系
- Association between TNFRSF11A and TNFRSF11B gene polymorphisms and the outcome of hepatitis C virus infection
- 中华流行病学杂志, 2019, 40(10): 1291-1295
- Chinese Journal of Epidemiology, 2019, 40(10): 1291-1295
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.10.022
-
文章历史
收稿日期: 2018-11-09
2. 南京医科大学第一附属医院感染科 210029;
3. 东部战区疾病预防控制中心, 南京 210002;
4. 南京大学医学院附属鼓楼医院感染科 210008;
5. 南通大学附属南通市第三人民医院消化科 226001;
6. 南通大学附属南通市第三人民医院四病区 226001
2. Department of Infectious Diseases, The First Affiliated Hospital of Nanjing Medical University, Nanjing 210029, China;
3. Eastern Theater Command Center for Disease Prevention and Control, Nanjing 210002, China;
4. Department of Infectious Diseases, Nanjing Drum Tower Hospital, The Affiliated Hospital of Nanjing University Medical School, Nanjing 210008, China;
5. Department of Gastroenterology, The Third People's Hospital of Nantong Affiliated to Nantong University, Nantong 226001, China;
6. Fourth Ward, The Third People's Hospital of Nantong Affiliated to Nantong University, Nantong 226001, China
丙型肝炎(丙肝)病毒(HCV)感染是重要的全球公共卫生问题之一,据估计全球约有7 100万例HCV慢性感染者[1]。但是HCV感染及进展中的具体机制尚不清楚,目前研究发现这一过程除涉及病毒因素、环境因素外,不同个体之间遗传差异导致的不同细胞免疫应答反应在HCV感染过程中有着重要作用[2]。在参与病毒感染细胞免疫调节的分子中,肿瘤坏死因子超家族(tumor necrosis factor superfamily,TNFSF)/肿瘤坏死因子受体超家族(tumor necrosis factor receptor superfamily,TNFRSF)逐渐引起关注,两者相互作用参与的生理和病理过程在炎症免疫性疾病发生、发展过程中起着重要作用[3]。研究发现,HCV感染慢性化的机制可能同TNFSF/TNFRSF介导的异常信号传导有关,通过HCV核心蛋白与两种受体的结合,引起复杂的细胞凋亡及抗凋亡信号,导致感染慢性化[4-5]。TNFRSF的两个成员:TNFRSF11A位于18号染色体,编码核因子κB受体活化因子(receptor activator of NF-κB,RANK);TNFRSF11B位于8号染色体,编码诱导受体骨保护素(osteoprotegerin,OPG)。RANK和OPG都是RANK配体(RANKL)的受体,三者形成的RANKL/RANK/OPG通路在细胞死亡和增殖、炎症和免疫中起重要作用[6]。本研究选取了TNFRSF11A基因的rs1805034位点和TNFRSF11B基因的rs2073617位点,探讨其基因多态性与HCV感染易感性及感染慢性化的关系。
对象与方法1.研究对象:2008-2016年从血液透析人群、吸毒人群和既往有偿献血人群中纳入2 729例研究对象,均为汉族,其中包括723例血液透析者(江苏省9家医院血液透析中心肾透析人群)、455例吸毒者(南京市公安局强制戒毒所吸毒人群)以及1 551例有偿献血者(江苏省镇江某地区6个自然行政村的既往有偿献血人群)。根据血清学检查结果对研究对象进行分组,其中抗-HCV阳性者作为HCV感染组(1 243例),抗-HCV阴性者作为对照组(1 486例)。HCV感染组根据HCV RNA浓度可进一步分组,RNA阴性作为自限清除组(494例),RNA阳性者作为持续感染组(749例)。所有分组结果均是基于6个月的随访所确定的。纳入研究的HCV感染病例均未经任何抗病毒药物治疗,且未联合感染其他类型肝炎病毒。
2.研究方法:
(1)流行病学调查:由经过专门培训的调查人员对调查对象进行问卷调查。针对有偿献血者,采用《江苏省某地区既往有偿献血人群丙肝状况调查表》;针对透析人群,采用《丙肝肾透析病例健康状况相关因素调查表》;针对吸毒人群,采用《药物滥用者丙肝感染相关因素调查表》。调查内容包括一般人口学资料和相应的环境暴露资料。本研究通过解放军南京军区军事医学研究所伦理委员会审查(2017012)。所有研究对象均签署知情同意书。
(2)血清学检测和基因DNA的提取:调查后由专业医护人员抽取各研究对象静脉血5 ml,EDTA抗凝,在24 h内分离血浆、白细胞和红细胞,分装1.5 ml冻存管,置-20 ℃冷冻贮存待检。取适量血浆用于检测HCV抗体、乙肝五项及肝功能生化指标。HCV抗体检测采用北京金豪制药股份有限公司ELISA国产第三代HCV抗体检测EIA-1试剂盒进行初筛,采用雅培公司Abbott HCV EIA3.0试剂盒进行复检。基因DNA的提取采用传统的酚-氯仿抽提法。所有操作步骤严格按照说明书进行。
(3)基因多态性位点筛选和基因分型:从NCBI、HapMap等数据库中寻找和确认TNFRSF家族基因单核苷酸多态性(SNP)位点,选取在汉族人群中的最小等位基因频率(MAF)>5%的SNP,并结合国内外文献报道,最终选取了rs1805034和rs2073617两个位点。应用TaqMan探针实时荧光定量PCR技术对位点进行基因分型。引物和探针由南京骥骜生物技术有限公司设计,引物和探针序列见表 1。
3.统计学分析:采用EpiData 3.10软件建立数据库,采用Stata 15.0软件进行相关统计学分析。对照组、自限清除组和持续感染组之间一般人口学特征比较采用χ2检验;拟合优度χ2检验用于计算对照组的基因型分布是否符合Hardy-Weinberg平衡。根据4种遗传模型运用logistic回归分析两个位点与HCV感染转归的关系。分层分析用于控制混杂因素对分析结果的影响;采用Q检验确定亚组之间的异质性。以P<0.05为差异有统计学意义。
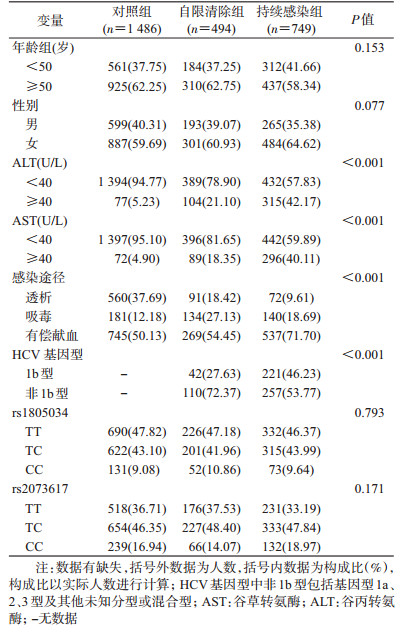
结果1.一般特征:对照组、自限清除组和持续感染组年龄和性别构成差异无统计学意义,3组AST、ALT水平、不同感染途径和基因型差异有统计学意义。位点rs1805034和rs2073617的基因型频率在对照组中符合Hardy- Weinberg平衡定律(P值分别为0.587和0.185),提示对照组人群具有代表性。见表 2。
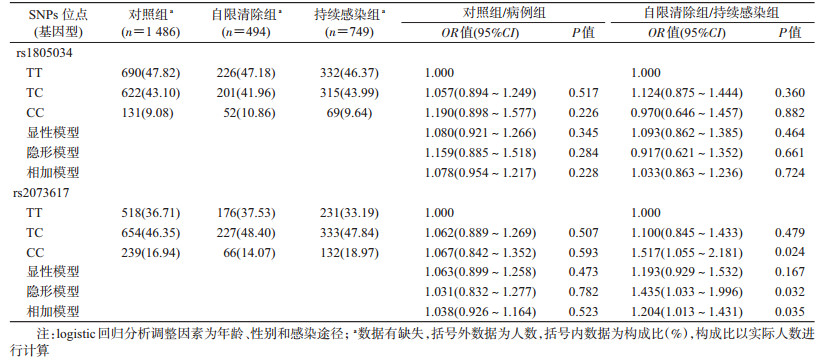
2.候选基因位点与HCV感染转归结局的关系:在对照组和HCV感染组(包括自限清除组和持续感染组)之间比较候选等位基因频率,进行HCV遗传易感性分析。结果显示rs1805034和rs2073617与HCV易感性的关联均无统计学意义。在自限清除组和持续感染组之间进行比较,分析宿主感染HCV后对病毒的清除能力。共显性模型结果显示,与携带TNFRSF11B rs2073617 TT基因型的个体相比,携带CC基因的个体易发生HCV感染慢性化(OR=1.517,95%CI:1.055~2.181,P=0.024)。隐性模型结果显示,与携带rs2073617 TT或TC基因型的个体相比,携带CC基因的个体易发生HCV感染慢性化(OR=1.435,95%CI:1.033~1.996,P=0.032);相加模型显示,随着携带C等位基因个数的增加,个体发生HCV感染慢性化的风险亦可增加(OR=1.204,95%CI:1.013~1.431,P=0.035)。未发现TNFRSF11A rs1805034与HCV感染慢性化存在关联。见表 3。
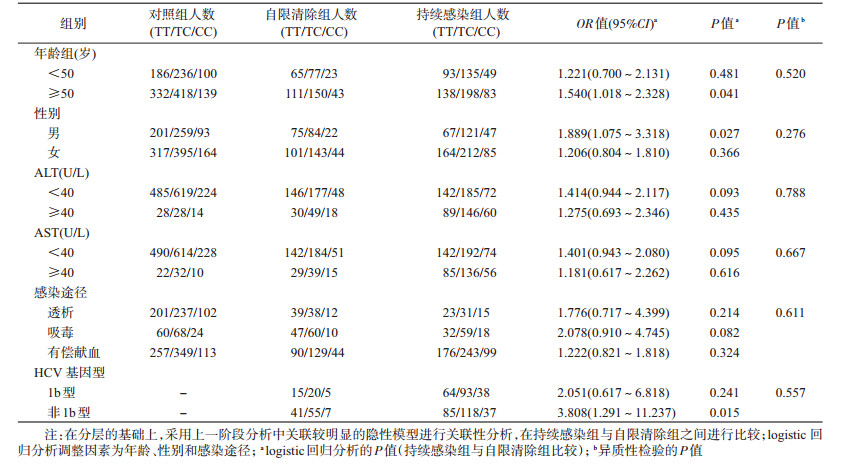
3. TNFRSF11B基因rs2073617位点多态性与HCV感染慢性化关联分层分析:分别以年龄、性别、ALT、AST、感染途径和HCV基因型作为分层因素,对rs2073617位点的基因型与HCV感染慢性化的关联进行分层分析,采用上一阶段分析中关联较明显的隐性模型进行关联性分析,结果见表 4。携带rs2073617位点CC基因型的年龄≥50岁者发生HCV感染慢性化的风险增加(OR=1.540,95%CI:1.018~2.328,P=0.041);携带rs2073617位点CC基因型的男性发生HCV感染慢性化的风险增加(OR=1.889,95%CI:1.075~3.318,P=0.027);在HCV非1b基因型人群中,携带rs2073617位点CC基因型的个体发生HCV感染慢性化的风险亦增加(OR=3.808,95%CI:1.291~11.237,P=0.015)。异质性检验结果显示各组之间均不存在显著异质性(P>0.05)。
丙肝作为感染慢性化程度较高的感染性疾病,研究TNFRSF11A、TNFRSF11B基因多态性与HCV感染及转归的关系有助于更好地阐明HCV感染遗传学的生物学机制。本研究发现携带TNFRSF11B rs2073617 C等位基因的病例更易发生感染慢性化。
研究发现,TNFRSF11A/TNFSF11/TNFRSF11B通路的表达和分泌可以调控免疫细胞的生长、分化,在免疫过程中发挥着重要作用,例如TNFRSF11A/TNFSF11/TNFRSF11B系统可以调控TNFRSF11A的表达水平,进而影响体内淋巴结的形成、T、B细胞的生长成熟过程和激活以及树突状细胞的生存、凋亡[7]。因此,我们推测,当HCV侵入机体后,TNFRSF11A/TNFSF11/TNFRSF11B系统参与重要的免疫调节,进而影响HCV感染转归的结局。
位于8号染色体上的TNFRSF11B基因,全长29 kb,是包含5个外显子的单拷贝基因[8]。目前研究已经发现TNFRSF11B基因SNPs与炎症性疾病的发展密切相关[9-11]。rs2073617位点位于TNFRSF11B基因启动子区域,既往的研究表明,rs2073617位点的SNP与炎症性肠病和类风湿性关节炎等炎症相关疾病有关[6, 12]。本研究结果显示,TNFRSF11B基因rs2073617位点突变基因型C是HCV慢性感染的危险基因型,其基因多态性可能在HCV感染的进展中起重要作用。在分层分析中,我们发现携带rs2073617位点突变基因型的≥50岁、男性和HCV非1b型人群更易形成HCV的慢性感染。进一步通过生物信息学预测发现,rs2073617的RegulomeDB评分为1d,表明该位点可以影响表达数量性状基因座(expression Quantitative Trait Loci,eQTL)、转录因子结合、结合模体和脱氧核糖核酸酶(deoxyribonuclease,DNase)活性峰值(http://www.regulomedb.org/results)。这些结果为进一步阐明rs2073617位点潜在的功能机制提供了一定的帮助。rs2073617可能通过影响TNFRSF11B基因的转录和表达功能,从而影响宿主免疫功能,最终影响HCV感染慢性化结局。
TNFRSF11A基因表达产物是内在造血细胞表面受体,可以刺激NF-κB受体激活,对T细胞和树突细胞有重要的调控作用,同时也可促进淋巴结发展[13]。本研究中调整年龄、性别和感染途径后,未发现rs1805034与HCV感染转归结局有统计学关联。
本研究存在局限性。因为选取了既往有偿献血、吸毒和肾透析3种高危人群作为研究对象,使研究结果缺乏代表性。其次,本研究仅研究了两个SNP位点,需要开展更多的关联性和功能性研究来证明这一结论。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
World Health Organization. Global hepatitis report 2017[R]. Geneva: WHO, 2017.
|
| [2] |
樊圆圆, 邵建国, 黄鹏, 等. 核转录因子κB基因多态性与人群感染丙型肝炎病毒的关联性[J]. 中华流行病学杂志, 2018, 39(9): 1261-1264. Fan YY, Shao JG, Huang P, et al. Association between nuclear factor kappa-light-chain-enhancer of activated B cells genetic polymorphisms and HCV susceptibility among the Chinese population under high-risk[J]. Chin J Epidemiol, 2018, 39(9): 1261-1264. DOI:10.3760/cma.j.issn.0254-6450.2018.09.022 |
| [3] |
Whalen DM, Hymowitz SG. Shining LIGHT on functional promiscuity in the TNF and TNFR superfamilies[J]. Structure, 2014, 22(9): 1221-1222. DOI:10.1016/j.str.2014.08.003 |
| [4] |
Saito K, Meyer K, Warner R, et al. Hepatitis C virus core protein inhibits tumor necrosis factor alpha-mediated apoptosis by a protective effect involving cellular FLICE inhibitory protein[J]. J Virol, 2006, 80(9): 4372-4379. DOI:10.1128/JVI.80.9.4372-4379.2006 |
| [5] |
Pompili M, Biolato M, Miele L, et al. Tumor necrosis factor-α inhibitors and chronic hepatitis C:a comprehensive literature review[J]. World J Gastroenterol, 2013, 19(44): 7867-7873. DOI:10.3748/wjg.v19.i44.7867 |
| [6] |
Zavala-Cerna MG, Moran-Moguel MC, Cornejo-Toledo JA, et al. Osteoprotegerin polymorphisms in a mexican population with rheumatoid arthritis and generalized osteoporosis:a preliminary report[J]. J Immunol Res, 2015, 2015: 376197. DOI:10.1155/2015/376197 |
| [7] |
Kong YY, Yoshida H, Sarosi I, et al. OPGL is a key regulator of osteoclastogenesis, lymphocyte development and lymph-node organogenesis[J]. Nature, 1999, 397(6717): 315-323. DOI:10.1038/16852 |
| [8] |
Xue JB, Zhan XL, Wang WJ, et al. OPG rs2073617 polymorphism is associated with upregulated OPG protein expression and an increased risk of intervertebral disc degeneration[J]. Exp Ther Med, 2016, 12(2): 702-710. DOI:10.3892/etm.2016.3342 |
| [9] |
Cai YM, Wang J, Wang QW, et al. Association of OPG gene polymorphism with susceptibility to rheumatoid arthrits in Chinese Han[J]. Immunol Lett, 2015, 165(2): 102-106. DOI:10.1016/j.imlet.2014.07.011 |
| [10] |
Zhou J, Zhao Y. Osteoprotegerin gene (OPG) polymorphisms associated with peri-implantitis susceptibility in a Chinese Han population[J]. Med Sci Monit, 2016, 22: 4271-4276. DOI:10.12659/MSM.897592 |
| [11] |
Kadkhodazadeh M, Tabari ZA, Ardakani MR, et al. Analysis of osteoprotegerin (OPG) gene polymorphism in Iranian patients with chronic periodontitis and peri-implantitis. A cross-sectional study[J]. Eur J Oral Implantol, 2012, 5(4): 381-388. DOI:10.1016/j.jds.2012.10.003 |
| [12] |
Krela-Kazmierczak I, Kaczmarek-Rys M, Szymczak A, et al. Bone metabolism and the c.-223C>T polymorphism in the 5' UTR region of the osteoprotegerin gene in patients with inflammatory bowel disease[J]. Calcif Tissue Int, 2016, 99(6): 616-624. DOI:10.1007/s00223-016-0192-9 |
| [13] |
Anderson DM, Maraskovsky E, Billingsley WL, et al. A homologue of the TNF receptor and its ligand enhance T-cell growth and dendritic-cell function[J]. Nature, 1997, 390(6656): 175-179. DOI:10.1038/36593 |
 2019, Vol. 40
2019, Vol. 40