文章信息
- 姚苹苹, 陈钢, 徐芳, 杨章女, 陈晨, 孙一晟, 卢杭景, 庞卫龙, 张云, 朱函坪, 项海青.
- Yao Pingping, Chen Gang, Xu Fang, Yang Zhangnyu, Chen Chen, Sun Yisheng, Lu Hangjing, Pang Weilong, Zhang Yun, Zhu Hanping, Xiang Haiqing.
- 浙江省天台县2011-2018年汉坦病毒基因型别和进化变异研究
- Genotype and evolution of hantavirus in Tiantai of Zhejiang province, 2011-2018
- 中华流行病学杂志, 2019, 40(10): 1285-1290
- Chinese Journal of Epidemiology, 2019, 40(10): 1285-1290
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.10.021
-
文章历史
收稿日期: 2019-02-05
2. 天台县疾病预防控制中心 317200;
3. 南京军区军事医学研究所 210002;
4. 杭州市卫生事业发展中心 310001
2. Tiantai Municipal Center for Disease Control and Prevention, Tiantai 317200, China;
3. Institute of Military Medicine, Nanjing Command, Nanjing 210002, China;
4. Hangzhou Municipal Center for Health Development, Hangzhou 310001, China
汉坦病毒属于布尼亚病毒科汉坦病毒属(hantavirus,HV),是有胞膜、单股、负链、分节段的RNA病毒,其基因结构分为大(L)、中(M)、小(S)3个片段,分别编码病毒的RNA聚合酶、糖蛋白(GP)G1和G2以及核衣壳蛋白(NP)[1]。HV的宿主动物是啮齿类动物并由啮齿动物传播给人类,人类感染HV主要导致肾综合征出血热(hemorrhagic fever with renal syndrome,HFRS)和HV肺综合征(Hantavirus pulmonary syndrome,HPS)。HV包括汉滩病毒(hantaan virus,HTNV)、汉城病毒(seoul virus,SEOV)、多布拉法-贝尔格莱德病毒(dobrava-beigrade,DOBV)、普马拉病毒(puumala virus,PUUV)等型别的病毒[2],我国以HTNV和SEOV为主要流行株[3]。
中国是HFRS流行的主要地区,发病率占全世界90%以上[4],浙江省是我国HFRS的主要疫区之一[5],天台县又是浙江省HFRS的高发区。庞卫龙等[6]报道天台县自1965年发现首例HFRS以来,疫情开始逐渐上升,20世纪80年代达到高峰,1983年发病822例,发病率为170.4/10万,居浙江省首位[6]。1984年开展监测并采取针对性防治措施,发病率逐年下降,但仍是全国疫情较重的发病县之一,为HFRS国家监测点。
为了研究天台县HV的基因型别分布及病毒进化变异规律和流行趋势,我们持续对该地区的HV宿主动物进行调查[7]。本研究在前期的工作基础上利用HTNV和SEOV特异性引物,应用巢式PCR方法对2011-2018年HV抗原阳性的鼠肺标本进行病毒核酸扩增分型和序列测定,将本次测序的天台HV部分M片段序列与HV国内外其他病毒株进行比较分析。
资料与方法1.研究样本:来自2011-2018年浙江省HFRS国家监测点天台县捕获的野外和居民区鼠形动物中HV抗原阳性的鼠肺标本67份。
2.病毒RNA提取:天台县HV抗原阳性的鼠肺标本经超声粉碎后采用EX2400核酸自动提取仪(上海之江生物科技股份有限公司)进行RNA提取,最后用40 μl的无RNA酶的去离子水溶解,-70 ℃保存备用。
3.巢式PCR分型和测序:使用M-MLV反转录酶和反转录引物(5′-TAGTAGTAGACTCC-3′)合成cDNA;应用M片段的通用外引物(上游引物:5′-AAAGTAGGTGITAYATCYTIACAATGTGG-3′、下游引物:5′-GTACAICCTGIRCCIACCCC -3′);型特异性引物:HTNV上游引物(5′-GAATCGATACT GTGGGCTGCAAGTGC -3′)、下游引物(5′-GGA TTAGAACCCCAGCTCGTCTC -3′);SEOV上游引物(5′- GTGGACTCTTCTTCTCATTATT-3′)、下游引物(5′-TGGGCAATCTGGGGGGTTGCATG -3′);PCR扩增的反应条件为95 ℃ 10 min,94 ℃ 60 s、55 ℃ 60 s、72 ℃ 45 s,扩增30个循环,72 ℃延伸12 min;扩增产物用1%~2%琼脂糖凝胶电泳鉴定,若条带的分子量与预期片段大小相同(HTNV扩增片段长度为385 bp,SEOV扩增片段长度为418 bp),则表明为特异性扩增产物,确定病毒的型别。天台县鼠肺标本经巢式PCR扩增后的阳性产物委托杭州擎科梓熙生物技术有限公司测序。
4.分析用毒株:选取DNA序列数据库(GenBank)有代表性的27株HV与天台县2011-2018年31份HV的序列进行比较分析,HTNV 17株和SEOV 10株,包括HTNV国际标准株76-118、SEOV国际标准株80-39、浙江省以往分离株及国内外其他分离株。27株病毒的型别、名称、宿主动物、来源以及序列号等信息,见表 1。参照孙彦峰等[8]对HV基因分型的研究,本研究的30株天台黑线姬鼠携带的病毒为HTNV的同一亚型;1株天台黑线姬鼠携带的病毒为SEOV,与浙江株Z37、ZT71和ZT10同为一个亚型。
5.核苷酸序列分析:2011-2018年天台县31份HV与GenBank中选择的国内外有代表性的27株HV,利用M基因G2区2003~2303 nt的核苷酸序列,应用Mega 5.0和DNA Star 7.1软件包进行系统发生分析,以邻位相连法(NJ)、T92+G模型构建系统进化树[8],分析病毒基因的型别和进化变异。
结果1. PCR检测及分型:采用巢式PCR方法,从天台县67份HV抗原阳性的鼠肺标本提取RNA,利用HTNV和SEOV特异性引物,经巢式PCR扩增,30份标本用HTNV特异性引物扩增出目的条带385 bp(2012年4份:T2012-52、T2012-58、T2012-61、T2012-80;2013年9份:T2013-2、T2013-9、T2013- 10、T2013-15、T2013-16、T2013-35、T2013-36、T2013- 66、T2013-67;2017年5份:T2017-2、T2017-3、T2017-12、T2017-63、T2017-71;2018年12份:T2018-9、T2018-108、T2018-123、T2018-130、T2018- 131、T2018-133、T2018-148、T2018-153、T2018- 164、T2018-167、T2018-180、T2018-187),而用SEOV特异性引物未扩增出基因片段;1份标本用SEOV特异性引物扩增出目的条带418 bp(2016年1份:T2016-31),而用HTNV特异性引物未扩增出基因片段。31份PCR产物阳性标本均来自黑线姬鼠,这表明PCR产物阳性的30只黑线姬鼠携带HTNV,1只黑线姬鼠携带SEOV。HTNV和SEOV PCR产物条带示意图见图 1。
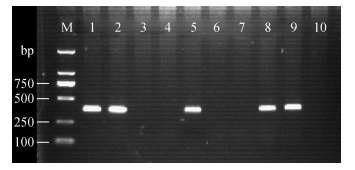
|
| 注:M:Mark;1和2:HTNV阳性鼠肺标本HTNV引物;3和4:HTNV阳性鼠肺标本SEOV引物;5:HTNV阳性对照Z10株HTNV引物;6:阴性对照(H2O)HTNV引物;7:SEOV阳性鼠肺标本HTNV引物;8:SEOV阳性鼠肺标本SEOV引物;9:SEOV阳性对照Z37株SEOV引物;10:阴性对照(H2O)SEOV引物 图 1 HV抗原阳性鼠肺标本经型特异性M基因引物巢式PCR扩增分型结果 |
2.同源性分析:天台监测点HV M基因G2区2003~2303 nt处300 bp的核苷酸序列经测序,30株HTNV中的T2018-130株与天台县其余29株及HTNV国际标准株76-118和国内外其他株相比较同源性差异均较大,T2018-130株与天台县其余29株同源性为84.8%~87.9%,与HTNV国际标准株76-118同源性为74.2%,与邻县嵊州市Z10株(本实验室HTNV疫苗株)同源性为76.0%,与国内外其他HTNV同源性为76.8%~86.6%;而天台县其余29株HTNV之间同源性为95.3%~100.0%。1株来自天台县黑线姬鼠的SEOV T2016-31株与SEOV国际标准株80-39同源性为93.4%,与同为天台县2003年分离自褐家鼠的SEOV ZT71株和2004年分离自东方田鼠的ZT10株同源性分别为98.7%和99.3%,与浙江省新昌县和温州市分离株同源性为92.0%~99.0%和84.1%~85.4%。
3.病毒基因系统发生树分析:利用M基因G2区2003~2303 nt的核苷酸序列进行系统发生分析,2011-2018年天台县监测点的31株病毒中30株分布在HTNV单元群,从系统发生树上看同为天台县的29株亲缘关系较近,分布较集中,而天台县T2018-130株与其余天台株及国内外其他株相比亲缘关系较远,但还是与天台株同分布在HTNV单元中的一个分支。来自天台县黑线姬鼠的SEOV T2016-31株分布在SEOV发生群,与天台株ZT10、ZT71和温州株Z37(本实验室SEOV疫苗株)构成一进化支,亲缘关系较近。构建的进化树见图 2。

|
| 注:-:信息未检索到;NR:序列未注册到GenBank;SNV:辛诺柏病毒;KBRV:哈巴罗夫斯基病毒 图 2 天台县2011-2018年HV与国内外代表性HV部分M片段核苷酸序列(2003~2303 nt)以NJ法构建的系统进化树 |
HV引发疾病的类型及其严重程度取决于病毒的型别。因此,对HV的分型以及阐明病毒之间的关系对HV病原学理论研究及其防治具有重要的意义。HV的基因结构分为大(L)、中(M)、小(S)3个片段,3个基因片段的核酸变异以M基因片段的变异最显著,被认为是决定HV毒力和致病性的主要因素[9]。不同型别HV的M片段核苷酸序列有明显差异,是研究HV基因变异和分型的主要区域[10]。张永振等[11]研究发现以NJ法用G2区的2003~2303 nt的核苷酸序列可以代替G1、M或M+S基因片段的全序列构建系统发生树对HV进行分型,而且NJ法可能优于最大简约法。基于病毒基因序列数据分析不但能对病毒进行基因分型,而且也能较好地反映病毒间的系统发生关系。本研究利用巢式PCR分型技术及核苷酸序列测定技术,采用NJ法对2011-2018年天台县HV M基因G2区2003~2303 nt的核苷酸序列构建系统发生树进行病毒基因分型及进化分析。巢式PCR经HTNV和SEOV型特异性引物扩增,67份HV抗原阳性鼠肺标本HTNV 30株、SEOV 1株。HTNV的主要自然宿主为黑线姬鼠,姚苹苹等[7]对天台县2011-2018年HV宿主动物进行了调查监测,发现当地鼠种78.0%为黑线姬鼠,本研究67份HV抗原阳性鼠肺1份来自褐家鼠,其余66份均来自黑线姬鼠。天台县属以黑线姬鼠为主要传染源、HTNV为主SEOV为辅的HFRS混合型疫区。
30株HTNV中29株病毒核苷酸同源性高达95.3%~100.0%,而T2018-130株与其余29株同源性为84.8%~87.9%,但与HTNV国际标准株76-118和国内外其他株相比较同源性更低,与76-118株为74.2%,与国内外其他HTNV为76.8%~86.6%。从系统发生树看,T2018-130株还是与天台县其余29株病毒分布在HTNV单元群中的同一分支,属同一亚型。
1株SEOV T2016-31来自黑线姬鼠,与SEOV国际标准株80-39同源性为93.4%,与同来源于天台县本实验室2003年分离自褐家鼠的SEOV ZT71株[12]和2004年分离自东方田鼠的ZT10株[13]同源性分别为98.7%和99.3%,与20世纪80年代分离自新昌县和温州株的同源性为92.0%~99.0%和84.1%~85.4%。从系统发生树看,本研究T2016-31株与天台县以往分离ZT10、ZT71株分布在同一进化支,同属一个亚型,3株同地区不同宿主来源的SEOV在系统发生树上亲缘关系较近。这个现象再次证明浙江省HV的基因差异和亲缘远近关系主要表现在地区性,而与病毒的宿主关系并不大[14]。这种地区性现象在浙江省的SEOV尤为明显,徐芳等[15]曾发现在浙江省建德市存在SEOV发生群中独立分支的特殊亚型病毒Gou3和ZJ5株,这2株病毒系本实验室分别在1981和2001年分离[16],时间跨度20年,但基因序列表现出高度的地区同源性[14]。
多年的HV进化理论认为HV与其原始宿主间存在共进化关系,即一个型别的HV往往只有一种原始宿主动物,即每型病毒具有相对稳定的宿主与啮齿类动物的种类形成对应关系[17],一般认为同一个型别的HV在一定区域只有一种宿主动物。浙江省的HTNV和SEOV的宿主动物大部分分别为黑线姬鼠和褐家鼠,但HV在与宿主的共进化过程中,也会出现病毒与宿主动物不一致的现象。2003年本实验室从天台县的褐家鼠中分离到SEOV ZT71株,2004年率先从天台县的东方田鼠中分离到SEOV ZT10株,本研究又从天台县的黑线姬鼠中发现SEOV T2016-31株。天台县的这种原始宿主与其携带病毒不一致的现象称为宿主“溢出”(spillover),国内近年研究也证实了不同型HV基因宿主转换现象:Liu等[18]研究发现湖北省出现SEOV溢出感染HV的自然宿主黑线姬鼠;范胜涛等[19]也报道了吉林省的褐家鼠、黑线姬鼠和小家鼠中均检测到SEOV的存在。宿主溢出可能是暂时的,很快在动物体内得以清除,对病毒的进化和维持没有太大的意义;但也可能是永久性的,发生宿主转换并加以进化[20]。
天台县属浙中丘陵山区,其中山丘占天台县总面积的82.3%、溪流山塘占4.0%,气候温暖湿润,生态环境非常适合啮齿类动物的生存和活动,是浙江省乃至全国重要的HFRS自然疫源地。本实验室研究发现天台县SEOV进化过程中由褐家鼠溢出到东方田鼠和黑线姬鼠,而且这一模式在鼠类中可能不断循环。天台县HV主要以HTNV为主的地区聚集性及SEOV可能不断进化突破了宿主之间的间隔现象,使此地HV防控形势变得更加严峻。因此,有必要持续深入开展HV的宿主动物调查和病毒基因进化变异研究,进一步了解HV进化规律,为寻找HV的进化动力提供资料。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
张俊丽, 肖寒. 肾综合征出血热病原学、实验室检测、防治措施的研究进展[J]. 医学综述, 2017, 23(21): 4303-4307. Zhang JL, Xiao H. Research progress of etiology, laboratory detection, prevention of hemorrhagic fever with renal syndrome[J]. Med Recapit, 2017, 23(21): 4303-4307. DOI:10.3969/j.issn.1006-2084.2017.21.028 |
| [2] |
杨其松, 张天琛, 刘晓青, 等. 汉坦病毒及其与宿主基因相互作用研究进展[J]. 中华疾病控制杂志, 2018, 22(7): 751-754. Yang QS, Zhang TC, Liu XQ, et al. Research progress of Hantavirus and its interaction with host genes[J]. Chin J Dis Control Prev, 2018, 22(7): 751-754. DOI:10.16462/j.cnki.zhjbkz.2018.07.022 |
| [3] |
黄鹏, 杨章女, 刘源, 等. 东南沿海地区1980-2015年汉坦病毒分子特征及流行病学分析[J]. 中国媒介生物学及控制杂志, 2017, 28(4): 354-358. Huang P, Yang ZN, Liu Y, et al. The molecular characteristics and epidemiological analysis of Hantavirus in southeast coastal area of China from 1980 to 2015[J]. Chin J Vector Biol Control, 2017, 28(4): 354-358. DOI:10.11853/j.issn.1003.8280.2017.04.012 |
| [4] |
王芹, 李建东, 张全福, 等. 2014年全国肾综合征出血热监测总结和疫情分析[J]. 疾病监测, 2016, 31(3): 192-199. Wang Q, Li JD, Zhang QF, et al. Surveillance for hemorrhagic fever with renal syndrome in China, 2014[J]. Dis Surveill, 2016, 31(3): 192-199. DOI:10.3784/j.issn.1003-9961.2016.03.005 |
| [5] |
Wu HC, Wang XY, Xue M, et al. Spatial-temporal characteristics and the epidemiology of haemorrhagic fever with renal syndrome from 2007 to 2016 in Zhejiang province, China[J]. Sci Rep, 2018, 8(1): 10244. DOI:10.1038/s41598-018-28610-8 |
| [6] |
庞卫龙, 葛君华, 周雪冰. 2011-2016年天台县肾综合征出血热监测分析[J]. 中国农村卫生事业管理, 2018, 38(7): 914-916. Pang WL, Ge JH, Zhou XB. A surveillance analysis of hemorrhagic fever with renal syndrome in Tiantai county, Zhejiang province from 2011 to 2016[J]. Chin Rural Health Serv Adm, 2018, 38(7): 914-916. DOI:10.3969/j.issn.1005-5916.2018.07.021 |
| [7] |
姚苹苹, 徐芳, 朱函坪, 等. 天台县汉坦病毒宿主动物调查及病毒分离结果[J]. 预防医学, 2019, 31(5): 433-436, 440. Yao PP, Xu F, Zhu HP, et al. The host investigation and virus isolation of Hantavirus in Tiantai county[J]. Prev Med, 2019, 31(5): 433-436, 440. DOI:10.19485/j.cnki.issn2096-5087.2019.05.001 |
| [8] |
孙彦峰, 胡侃, 武永平, 等. 宝鸡地区汉坦病毒基因分型及序列特征研究[J]. 职业与健康, 2016, 32(8): 1049-1052. Sun YF, Hu K, Wu YP, et al. Study on genotype and gene sequence characters of Hantavirus in Baoji[J]. Occup Health, 2016, 32(8): 1049-1052. DOI:10.13329/j.cnki.zyyjk.2016.0290 |
| [9] |
车雷, 张国斌, 高丹, 等. 沈阳市鼠携带汉坦病毒G2片段基因分型及序列特征分析[J]. 中国媒介生物学及控制杂志, 2019, 30(1): 91-94. Che L, Zhang GB, Gao D, et al. Genotyping and sequence analysis of G2 segment of Hantavirus carried by rats in Shenyang, China[J]. Chin J Vector Biol Control, 2019, 30(1): 91-94. DOI:10.11853/j.issn.1003.8280.2019.01.022 |
| [10] |
Hu TS, Fan QS, Hu XB, et al. Molecular and serological evidence for Seoul virus in rats (Rattus norvegicus) in Zhangmu, Tibet, China[J]. Arch Virol, 2015, 160(5): 1353-1357. DOI:10.1007/s00705-015-2391-5 |
| [11] |
张永振, 屈勇刚, 杨东明, 等. 汉坦病毒系统发生分析及其基因分型的研究[J]. 中国人兽共患病学报, 2006, 22(3): 193-197, 201. Zhang YZ, Qu YG, Yang DM, et al. The genotypes and phylogenetic analysis of Hantavirus[J]. Chin J Zoonoses, 2006, 22(3): 193-197, 201. DOI:10.3969/j.issn.1002-2694.2006.03.001 |
| [12] |
谢荣辉, 翁景清, 姚苹苹, 等. 浙江新分离汉坦病毒ZT71株S基因片段的克隆及序列分析[J]. 中国人兽共患病杂志, 2005, 21(7): 570-573. Xie RH, Weng JQ, Yao PP, et al. Meolecular cloning and nucletide sequence analysis of the S segment of Hantavirus ZT71 strain isolated in Zhejiang province[J]. Chin J Zoonoses, 2005, 21(7): 570-573. DOI:10.3969/j.issn.1002-2694.2005.07.007 |
| [13] |
翁景清, 谢荣辉, 姚苹苹, 等. 汉坦病毒东方田鼠分离株ZT10的分子特性研究[J]. 中华实验和临床病毒学杂志, 2008, 22(1): 2-5. Weng JQ, Xie RH, Yao PP, et al. Molecular characterization of Hanta virus Zhejiang isolate ZT10 strain from M. fartis[J]. Chin J Exp Clin Virol, 2008, 22(1): 2-5. DOI:10.3760/cma.j.issn.1003-9279.2008.01.002 |
| [14] |
姚苹苹, 朱函坪, 徐芳, 等. 浙江省汉坦病毒基因分子进化分析[J]. 病毒学报, 2010, 26(6): 465-470. Yao PP, Zhu HP, Xu F, et al. Molecular evolution analysis of Hantaviruses in Zhejiang province[J]. Chin J Virol, 2010, 26(6): 465-470. DOI:10.13242/j.cnki.bingduxuebao.002124 |
| [15] |
徐芳, 姚苹苹, 朱函坪, 等. 汉坦病毒浙江分离株的基因分型[J]. 中华实验和临床病毒学杂志, 2012, 26(5): 344-347. Xu F, Yao PP, Zhu HP, et al. Genotype of Hantavirus in Zhejiang province[J]. Chin J Exp Clin Virol, 2012, 26(5): 344-347. DOI:10.3760/cma.j.issn.1003-9279.2012.05.008 |
| [16] |
姚苹苹, 徐芳, 朱函坪, 等. 汉坦病毒Vero细胞灭活疫苗候选毒种筛选[J]. 中国公共卫生, 2007, 23(2): 208-209. Yao PP, Xu F, Zhu HP, et al. Selection of virus strain for production of inactivated Hantavirus vaccine of Vero cells[J]. Chin J Public Health, 2007, 23(2): 208-209. DOI:10.11847/zgggws2007-23-02-45 |
| [17] |
马超锋, 余鹏博, 吴瑞, 等. 携带汉坦病毒宿主动物分布特征研究[J]. 中国人兽共患病学报, 2015, 31(1): 26-29. Ma CF, Yu PB, Wu R, et al. Spatial and temporal distribution of hosts carrying Hantaviruses[J]. Chin J Zoonoses, 2015, 31(1): 26-29. DOI:10.3969/cjz.j.issn.1002-2694.2015.01.006 |
| [18] |
Liu J, Liu DY, Chen W, et al. Genetic analysis of Hantaviruses and their rodent hosts in central-south China[J]. Virus Res, 2012, 163(2): 439-447. DOI:10.1016/j.virusres.2011.11.006 |
| [19] |
范胜涛, 高晓龙, 李元果, 等. 吉林省汉坦病毒宿主动物携带病毒的遗传进化分析[J]. 中国生物制品学杂志, 2014, 27(4): 467-471, 475. Fan ST, Gao XL, Li YG, et al. Genetics and evolution of viruses carried by animal hosts of Hantaviruses in Jilin province, China[J]. Chin J Biol, 2014, 27(4): 467-471, 475. DOI:10.13200/j.cnki.cjb.000245 |
| [20] |
Hu XQ, Li SG, Liu H, et al. Diversity and distribution of host animal species of Hantavirus and risk to human health in Jiuhua Mountain Area, China[J]. Biomed Environ Sci, 2014, 27(11): 849-857. DOI:10.3967/bes2014.122 |
 2019, Vol. 40
2019, Vol. 40