文章信息
- 郭颂, 凌锋, 王金娜, 吴瑜燕, 侯娟, 龚震宇.
- Guo Song, Ling Feng, Wang Jinna, Wu Yuyan, Hou Juan, Gong Zhenyu.
- 基于微卫星标记的中国南方沿海地区白纹伊蚊种群遗传多样性研究
- Genetic diversity of Aedes albopictus populations in the coastal areas of southern China, using the microsatellite markers
- 中华流行病学杂志, 2019, 40(8): 992-996
- Chinese Journal of Epidemiology, 2019, 40(8): 992-996
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.08.022
-
文章历史
收稿日期: 2019-03-04
近年来,登革热在中国南方各省份暴发风险不断增加[1-2],其中广东、广西、海南、福建、浙江等省份常有登革热暴发疫情,为登革热监测Ⅰ类地区。白纹伊蚊是中国登革热主要的传播媒介。研究表明不同地理种群白纹伊蚊对于登革热病毒易感性存在差异[3],这种差异在某种程度上源于不同地理种群伊蚊存在生理结构上的不同[4]。分子生物学为探索物种的遗传差异提供了有效的途径,常用的微卫星标记在亲权鉴定、种类鉴定、种群遗传学、亲缘地理学和基因图谱构建等方面有着很重要的应用价值[5]。微卫星位点具有相当高多态性,相比较其他中性分子,高多态性的微卫星标记能检测出更多物种种群内部的变异,并能有效的区分种群亚结构[6-7]。本研究通过微卫星分子标记研究我国南方沿海不同地理区域白纹伊蚊种群的遗传差异及种群间的系统发育关系,建立中国南方沿海地区白纹伊蚊种群的本底资料,为病媒生物控制和登革热风险预测提供理论依据。
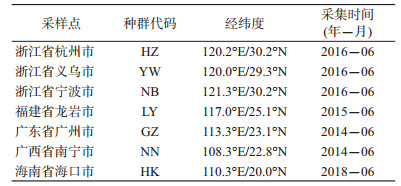
材料和方法1.白纹伊蚊样本的采集和基因组DNA的提取:综合考虑地理区域代表性原则,选择7个白纹伊蚊采样点:浙江省杭州市、义乌市和宁波市、福建省龙岩市、广东省广州市、广西壮族自治区南宁市、海南省海口市,均为登革热流行高风险区域。每个采样点在6-10月按照东南西北方位通过人诱法捕捉白纹伊蚊雌性成蚊,每个采样点的采集数量≥40只,采集的标本浸入无水乙醇置于-80 ℃冰箱(日本SANYO公司)保存。采用DNA提取试剂盒(德国QIAGEN公司)提取单个白纹伊蚊基因组DNA,-20 ℃保存备用。
2.微卫星标记扩增引物的筛选:通过文献查找可用于白纹伊蚊或者近似种的微卫星标记引物[8-9],对于能扩增出条带,且能够通过优化反应条件扩增出单一条带(或两条带)的引物进行进一步的筛选,获得高多态性位点的引物。扩增产物进行3%琼脂糖凝胶电泳,选取能扩增出≥4个等位基因,且扩增稳定的引物,对选取的微卫星引物上游进行荧光标记(FAM蓝色、HEX绿色或ROX红色,上海生工生物工程股份有限公司),通过毛细管电泳检测样本扩增的条带情况,选择多态信息量较高的引物作为最后入选的引物。
3.微卫星标记的扩增和电泳:使用已经筛选好的微卫星引物对所有的白纹伊蚊标本进行PCR扩增。每对微卫星引物的上游进行荧光标记(FAM蓝色、HEX绿色或ROX红色,上海生工生物工程股份有限公司),采用25 μl反应体系:LA Taq(5 U/μl,日本TaKaRa公司)0.15 μl;dNTP Mix (10 mmol/L each,日本TaKaRa公司) 0.5 μl;10×Buffer (日本TaKaRa公司)2.5 μl;Primers(10 μmol/L,上海生工生物工程股份有限公司)0.5 μl;DNA 1 μl;ddH2O(上海生工生物工程股份有限公司)20.35 μl。用3%琼脂糖凝胶电泳对扩增结果进行检测,对未扩增出条带或者扩增条带不清晰的样品,重新进行扩增。将所有扩增出清晰条带的样品送上海生工生物工程股份有限公司进行毛细管电泳。
4.数据读取:毛细管电泳结果采用Genemapper 4.0软件分析,查看电泳的质量,并将峰图信号转化为等位基因长度,从而获得的个体微卫星基因型用于种群遗传多样性分析。
5.统计学分析:综合所有微卫星位点的数据,利用GeneAlex 6.41软件和FSTAT 2.9.4软件计算群体的等位基因数(Number of different alleles,NA)、有效等位基因数(Number of effective alleles,NE)、等位基因丰度(Allele richness,RS)、信息指数(Shannon’s information index,I)、观察杂合度(Observed heterozygosity,HO)、期望杂合度(Expected heterozygosity,HE)、近交系数(Inbreeding coefficient,FIS),并综合各个指标分析种群间的遗传多样性差异;采用FSTAT 2.9.4软件分别检验每个群体每个位点是否符合Hardy-Weinberg equilibrium平衡(HWE),以及成对位点的连锁不平衡水平(Linkage disequilibrium,LD);白纹伊蚊种群的遗传分化程度通过不同地理区域种群间的分化系数(Fixation index,FST)值来衡量,对比分析区域内种群的差异程度,基因流(Number of migrants per generation,Nm)计算公式为Nm=(1-FST)/4 FST;在Mega 5.0软件中利用Nei遗传距离获得种群间的Neighbor-Joining法系统发育树,分析种群间的聚类关系;应用贝叶斯法在Structure 2.3.4软件中推论分支数(K),应用的模型是假设等位基因频率与群体相关联(设λ为1,其他参数为默认),运行时选择混合选项(admixture model),对于每个K值(K=1-7)进行10次独立的运算,参数设置主要为在Run Length中,Length of Burn in Period为1 000 000,Number of MCMC Reps after Burn in为100 000,应用Evanno等[10]的方法确定分支数的最大可能性。双侧检验,以P<0.05为差异有统计学意义。
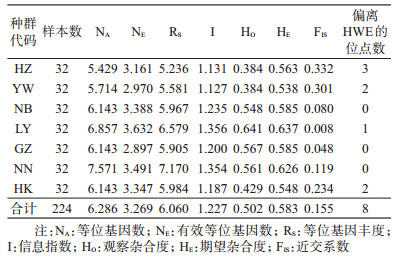
结果1.本研究采样点及采集信息:每个采样点用于检测的白纹伊蚊数量是32只,7个采样点共检测白纹伊蚊标本224只。白纹伊蚊的鉴定通过形态学和分子鉴定同时进行,避免采集的标本中混入其他近似种的蚊虫。筛选的微卫星标记位点信息见表 1,通过两轮筛选,共获得7对引物,每个引物的上游分别进行荧光标记,并成功对白纹伊蚊DNA进行PCR扩增和毛细管电泳,所筛选的微卫星位点和引物被证明适用于白纹伊蚊的分子生物学分析。见表 2。
2.各采样点白纹伊蚊微卫星标记遗传特征:整体上看7个采样点的微卫星位点特征较接近,微卫星位点在各群体均存在一定程度的多态性。南宁种群的NA(7.571)和RS(7.170)均高于其他地区,而龙岩种群的NE(3.632)和I(1.356)最高,这两个种群表现出相对较丰富的多样性,来自杭州和义乌的白纹伊蚊种群表现出较低水平多样性,各个参数(NA、NE、RS、I)值均相对较低。见表 3。
在所有的白纹伊蚊种群中,龙岩种群的HO(0.641)和HE(0.637)最高,该指标同样表现出较高水平的多样性,杭州市、义乌市和海口市的白纹伊蚊种群均HO<HE,表现为杂合子不足。FIS也表现出类似的结论,本研究白纹伊蚊7个群体的FIS范围为0.008(LY)~0.332(HZ),平均值为0.155,结果提示群体内的杂合子不足,发生近交的机会较多,这与白纹伊蚊活动范围有限,较难发生远交有关。但是龙岩种群的FIS值(0.008)接近0,且群体HO(0.641)与HE(0.637)接近,则表示群体受近交和外来选择等因素的影响较小,群体处于遗传平衡的状态。
对所有群体的所有位点进行49次HWE检验,结果显示共7个位点在群体中偏离HWE(表 1)。杭州白纹伊蚊种群存在3个偏离HWE的位点,龙岩种群仅有1个,而在宁波、广州和南宁种群中无位点偏离HWE。对所有群体的所有位点进行连锁不平衡检测,结果显示在各个种群中未发现存在相互连锁关系的位点(P<0.05)。
3.各采样点白纹伊蚊种群的配对FST值:一般认为FST<0.05显示种群出现轻度分化,0.05<FST<0.15显示种群出现中度分化,0.15<FST<0.25显示种群出现高度分化。7个采样点的白纹伊蚊种群总体FST为0.058,表明7个白纹伊蚊种群间的遗传变异为5.8%,表现出中低水平的遗传分化,最大值0.140出现在杭州和海口种群之间,表示二者的种群遗传分化程度较大,最小值出现在杭州和义乌种群之间,表示二者的种群遗传分化程度较小。均Nm>1,可以抵御遗传漂变造成的群体间遗传分化,最大值71.18出现在杭州和义乌种群之间,表现出较高的基因交流程度。见表 4。
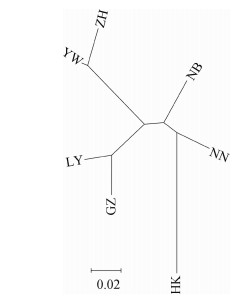
4.将所有白纹伊蚊种群的微卫星遗传距离数据用于构建Neighbor-Joining聚类关系,结果表明中国南方沿海的白纹伊蚊种群差异随着地理距离的增加而增大,其中来自浙江省杭州市和义乌市的白纹伊蚊种群构建成为1个分支,福建省和广东省的白纹伊蚊种群构成1个分支,广西壮族自治区和海南省的白纹伊蚊种群构成1个分支,且海南种群与其他种群的遗传差异较大。见图 1。
|
| 图 1 依据白纹伊蚊地理株Nei遗传距离构建的聚类关系(Neighbor-Joining法) |
5.应用Structure 2.3.4软件对所有微卫星数据进行贝叶斯分支分析,通过连续K值间的似然函数变异的二阶比率分析表明所有白纹伊蚊种群最可能的分支数K为3,第1分支用蓝色表示,主要包含杭州和义乌种群,第2分支用绿色表示,主要包含福建省龙岩市、广东省广州市和广西壮族自治区南宁市种群,第3分支用红色表示,主要包含海南种群,宁波种群3个分支存在较均衡,各个种群整体显示出地理区域差异。见图 2。

|
| 图 2 Structure 2.3.4软件贝叶斯法分支分析(K=3) |
本研究通过微卫星标记研究中国南方沿海地区白纹伊蚊种群的遗传多样性,每个种群选择的样本量>30个,符合文献报道中对样本量的要求[11],且所有种群7个微卫星位点的NA为6.286,RS为6.060,I为1.227,上述数据提示本研究应用的微卫星位点的多态性适当,是较为理想的检测白纹伊蚊群体遗传差异的分子标志,为分析遗传多样性提供充分的信息。
蚊虫群体多样性跟蚊虫所分布地区的地理特征、气候和蚊虫的生态习性等密切相关[12-14]。微卫星分析结果显示所采集的白纹伊蚊种群呈现一定程度的遗传多样性,与其他地区的结果类似[15-16],其多样性明显高于白纹伊蚊新入侵的区域[17],本研究中7个微卫星位点的HE为0.583,HO为0.502,均>0.5,表明其遗传多样性比较丰富,但是存在地区差异:来自浙江省杭州和义乌市的白纹伊蚊种群HO明显低于总体平均值,且这两个种群中均有微卫星位点偏离HWE,可能是由于群体内近交,或种群迁徙、基因突变、遗传漂变有关[18],也可能是种群历史上的瓶颈效应等因素造成的[19]。白纹伊蚊是中国南方的蚊虫优势种之一,适应当地的气候和地理环境,为南方登革热的主要传播媒介,且中国南方与东南亚和非洲经济文化交流频繁,每年登革热输入性病例较多,近年来南方多地相继暴发了登革热,在流行区实施以化学灭蚊为主的防控措施[20],使得白纹伊蚊种群密度迅速下降,孳生地不连续,局部群体内个体出现近交,同时高温、降雨量减少和各地开展的大规模灭蚊也会对白纹伊蚊种群多样性造成影响。这些可能是部分地区白纹伊蚊种群多样性较低的原因。
本研究白纹伊蚊种群的7个微卫星位点整体表现出中低水平遗传分化,群体间随机交配程度较高,FIS平均值为0.155,表明群体内存在较普遍的近交现象,造成杂合子缺失[21];与白纹伊蚊的飞行距离有限相关,同时其卵适应恶劣环境气候的能力较强,交通运输等因素间接地增加了白纹伊蚊的扩散能力[22-23],对白纹伊蚊的基因交流提供了条件,降低了各个地域的遗传分化。通过聚类分析和贝叶斯分析表明中国南方沿海地区的白纹伊蚊遗传分化程度随着地理距离的增加而增大,海南种群的分布地岛屿属于较为特殊的地理类型,存在明显的地理隔离,导致种群呈现明显的遗传分化,且该种群表现出较低的多样性,推测可能归因于“奠基者效应”[24],即由其他区域迁移而建立的种群,经长时间繁衍,虽个体数增加,但整个种群遗传多样性却未有提高。
中国南方沿海地区的白纹伊蚊种群存在中低水平的分化,频繁的基因交流是保持种群间不出现分化的重要因素,同时基因交流也使得不同地理区域白纹伊蚊种群具有相似的带毒能力[25],在登革热等蚊媒传染病输入性病例压力不断增加的形势下,基于综合防制和可持续控制策略的媒介伊蚊控制仍然需要得到重视。
综上所述,本研究通过微卫星位点分析中国南方沿海地区不同地理区域白纹伊蚊种群的遗传差异,7个白纹伊蚊种群整体呈现较丰富的遗传多样性和中低水平的遗传分化,种群间遗传差异随着地理距离的增加而增大,特别是海南种群与其他地理区域种群间出现了明显分化,但是单一遗传标记可能存在代表性不足的问题,还需要结合线粒体和核基因等做进一步研究,为探索白纹伊蚊的媒介效能和登革热风险评估提供本底数据和理论依据。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Luo L, Jiang LY, Xiao XC, et al. The dengue preface to endemic in mainland China:the historical largest outbreak by Aedes albopictus in Guangzhou, 2014[J]. Infect Dis Poverty, 2017, 6: 148. DOI:10.1186/s40249-017-0352-9 |
| [2] |
Sun JM, Lu L, Wu HX, et al. Epidemiological trends of dengue in mainland China, 2005-2015[J]. Int J Infect Dis, 2017, 57: 86-91. DOI:10.1016/j.ijid.2017.02.007 |
| [3] |
Phanpoowong T, Lek-Uthai U, Thongrungkiat S, et al. Dengue-2 virus carrying capacity of Thai Aedes aegypti strains with different susceptibility to deltamethrin[J]. Southeast Asian J Trop Med Public Health, 2012, 43(3): 634-640. DOI:10.1055/s-0031-1285856 |
| [4] |
Taracena ML, Bottino-Rojas V, Talyuli OAC, et al. Regulation of midgut cell proliferation impacts Aedes aegypti susceptibility to dengue virus[J]. PLoS Negl Trop Dis, 2018, 12(5): e0006498. DOI:10.1371/journal.pntd.0006498 |
| [5] |
Li YC, Korol AB, Fahima T, et al. Microsatellites:genomic distribution, putative functions and mutational mechanisms:a review[J]. Mol Ecol, 2002, 11(12): 2453-2465. DOI:10.1046/j.1365-294X.2002.01643.x |
| [6] |
Balloux F, Lugon-Moulin N. The estimation of population differentiation with microsatellite markers[J]. Mol Ecol, 2002, 11(2): 155-165. DOI:10.1046/j.0962-1083.2001.01436.x |
| [7] |
Lukindu M, Bergey CM, Wiltshire RM, et al. Spatio-temporal genetic structure of Anopheles gambiae in the Northwestern Lake Victoria Basin, Uganda:implications for genetic control trials in malaria endemic regions[J]. Parasit Vectors, 2018, 11: 246. DOI:10.1186/s13071-018-2826-4 |
| [8] |
Porretta D, Gargani M, Bellini R, et al. Isolation of microsatellite markers in the tiger mosquito Aedes albopictus (Skuse)[J]. Mol Ecol Notes, 2006, 6(3): 880-881. DOI:10.1111/j.1471-8286.2006.01384.x |
| [9] |
Behbahani A, Dutton TJ, Raju AK, et al. Polymorphic microsatellite loci in the mosquito Aedes polynesiensis[J]. Mol Ecol Notes, 2004, 4(1): 59-61. DOI:10.1046/j.1471-8286.2003.00567.x |
| [10] |
Evanno G, Regnaut S, Goudet J. Detecting the number of clusters of individuals using the software STRUCTURE:a simulation study[J]. Mol Ecol, 2005, 14(8): 2611-2620. DOI:10.1111/j.1365-294X.2005.02553.x |
| [11] |
Hernandez PA, Graham CH, Master LL, et al. The effect of sample size and species characteristics on performance of different species distribution modeling methods[J]. Ecography, 2006, 29(5): 773-785. DOI:10.1111/j.0906-7590.2006.04700.x |
| [12] |
Lutomiah J, Bast J, Clark J, et al. Abundance, diversity, and distribution of mosquito vectors in selected ecological regions of Kenya:public health implications[J]. J Vector Ecol, 2013, 38(1): 134-142. DOI:10.1111/j.1948-7134.2013.12019.x |
| [13] |
Abella-Medrano CA, Ibáñez-Bernal S, MacGregor-Fors I, et al. Spatiotemporal variation of mosquito diversity (Diptera:Culicidae) at places with different land-use types within a neotropical montane cloud forest matrix[J]. Parasit Vectors, 2015, 8: 487. DOI:10.1186/s13071-015-1086-9 |
| [14] |
Wagner S, Guidi V, Torgerson PR, et al. Diversity and seasonal abundances of mosquitoes at potential arboviral transmission sites in two different climate zones in Switzerland[J]. Med Vet Entomol, 2018, 32(2): 175-185. DOI:10.1111/mve.12292 |
| [15] |
Maynard AJ, Ambrose L, Cooper RD, et al. Tiger on the prowl:invasion history and spatio-temporal genetic structure of the Asian tiger mosquito Aedes albopictus (Skuse 1894) in the Indo- Pacific[J]. PLoS Negl Trop Dis, 2017, 11(4): e0005546. DOI:10.1371/journal.pntd.0005546 |
| [16] |
Manni M, Gomulski LM, Aketarawong N, et al. Molecular markers for analyses of intraspecific genetic diversity in the Asian Tiger mosquito, Aedes albopictus[J]. Parasit Vectors, 2015, 8: 188. DOI:10.1186/s13071-015-0794-5 |
| [17] |
Delatte H, Toty C, Boyer S, et al. Evidence of habitat structuring Aedes albopictus populations in réunion island[J]. PLoS Negl Trop Dis, 2013, 7(3): e2111. DOI:10.1371/journal.pntd.0002111 |
| [18] |
Smith BL, Lu CP, García-Cortés B, et al. Multilocus Bayesian estimates of intra-oceanic genetic differentiation, connectivity, and admixture in Atlantic Swordfish (Xiphias gladius L.)[J]. PLoS One, 2015, 10(6): e0127979. DOI:10.1371/journal.pone.0127979 |
| [19] |
Ferreira E, Souto L, Soares AMVM, et al. Genetic structure of the wild boar population in Portugal:evidence of a recent bottleneck[J]. Mamm Biol, 2009, 74(4): 274-285. DOI:10.1016/j.mambio.2008.05.009 |
| [20] |
Li YJ, Xu JB, Zhong DB, et al. Evidence for multiple-insecticide resistance in urban Aedes albopictus populations in southern China[J]. Parasit Vectors, 2018, 11: 4. DOI:10.1186/s13071-017-2581-y |
| [21] |
Politov DV, Pirko YV, Pirko NN, et al. Analysis of mating system in two Pinus cembra L. populations of the Ukrainian Carpathians[J]. Ann For Res, 2008, 51(1): 11-18. |
| [22] |
Alto BW, Juliano SA. Precipitation and temperature effects on populations of Aedes albopictus (Diptera:Culicidae):implications for range expansion[J]. J Med Entomol, 2001, 38(5): 646-656. DOI:10.1603/0022-2585-38.5.646 |
| [23] |
Kampen H, Kronefeld M, Zielke D, et al. Further specimens of the Asian tiger mosquito Aedes albopictus (Diptera, Culicidae) trapped in southwest Germany[J]. Parasitol Res, 2013, 112(2): 905-907. DOI:10.1007/s00436-012-3128-y |
| [24] |
Alessandri JL, Gordon CT, Jacquemont ML, et al. Recessive loss of function PIGN alleles, including an intragenic deletion with founder effect in La Réunion Island, in patients with Fryns syndrome[J]. Eur J Hum Genet, 2018, 26(3): 340-349. DOI:10.1038/s41431-017-0087-x |
| [25] |
张瑞玲, 姚广琴, 潘晓倩, 等. 不同地理种群白纹伊蚊线粒体基因COI的遗传多样性分析[J]. 中国人兽共患病学报, 2017, 33(4): 316-320. Zhang RL, Yao GQ, Pan XQ, et al. Genetic diversities of different geographical populations of Aedes albopictus based on mitochondrial gene COI[J]. Chin J Zoon, 2017, 33(4): 316-320. DOI:10.3969/j.issn.1002-2694.2017.04.005 |
 2019, Vol. 40
2019, Vol. 40