文章信息
- 肖金荣, 王可, 刘颖, 李泽武, 周玉静, 王唤卓, 卢静雅, 程珊珊, 魏晟.
- Xiao Jinrong, Wang Ke, Liu Ying, Li Zewu, Zhou Yujing, Wang Huanzhuo, Lu Jingya, Cheng Shanshan, Wei Sheng.
- 基于公共数据库挖掘肝细胞癌预后相关的长链非编码RNA分子标签
- Exploring of a prognostic long non-coding RNA signature of hepatocellular carcinoma by using public database
- 中华流行病学杂志, 2019, 40(7): 805-809
- Chinese journal of Epidemiology, 2019, 40(7): 805-809
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.07.014
-
文章历史
收稿日期: 2018-11-08
我国是肝癌高发地区之一,2014年中国肝癌新发病例36.5万例,发病率为26.67/10万,位居恶性肿瘤发病的第四位;因肝癌死亡数为31.9万,死亡率为23.31/10万,位居恶性肿瘤死亡的第二位[1-2]。肝癌发病较为隐匿,病情进展迅速,最新研究表明肝癌的5年存活率仅为10%~30%[3-4]。肝癌中最常见的类型为肝细胞癌。因此,寻找影响肝细胞癌预后相关指标,用于评估和预测肝细胞癌的预后,进而提高病例的生存率具有积极的作用。作为新一代的肿瘤分子标志物,长链非编码RNA(long non-protein coding RNA,lncRNA)是一种非蛋白编码的RNA,其长度大于200 nt。目前研究表明,lncRNA与肝细胞癌的发生、发展密切相关,在肝细胞癌的发病、转移和复发中起着重要的作用[5-7]。癌症和肿瘤基因图谱(The Cancer Genome Atlas,TCGA)数据库是当前全球最大的肿瘤组学数据数据库之一[8]。人类肿瘤相关基因表达汇编(Gene Expression Omnibus,GEO)数据库是一个高通量组学数据库[9-10]。本研究提取TCGA数据库中肝细胞癌RNA测序数据中lncRNA表达数据及临床数据,通过差异表达分析和LASSO Cox回归,挖掘与肝细胞癌预后相关的lncRNA,并根据LASSO回归分析结果构建预测肝细胞癌预后相关的lncRNA分子标签,用于评估肝细胞癌的预后情况。
资料与方法1.资料来源:lncRNA测序数据从https://portal.gdc.cancer.gov/下载得到。截至2018年2月,TCGA数据库中共有377例肝细胞癌癌组织样本及50例癌旁组织样本的RNA测序数据。TCGA数据库中RNA测序数据采用GENCODE v22数据库进行注释。根据GENCODE v22数据库中lncRNA基因注释文件提供的信息,提取出lncRNA表达数据。为了避免缺失值的影响,本研究去除lncRNA表达水平缺失值>总数10%的lncRNA位点,最终纳入15 901个lncRNA进入分析。去除临床信息缺失的个体,纳入297例病例进入分析。
2.分析方法:
(1)lncRNA在肝细胞癌和癌旁组织中差异表达分析:将50例癌和癌旁组织的样本通过t检验进行差异性分析,对癌和癌旁差异倍数对数的绝对值>1、错误发现率(FDR)<0.05的lncRNA进行后续研究。
(2)筛选与肝细胞癌预后相关的lncRNA:将上一步研究得出的差异lncRNA表达水平与肝细胞癌病例的临床预后资料进行拼接,然后采用LASSO Cox回归从中筛选与肝细胞癌预后相关的lncRNA。
(3)构建预测预后的lncRNA分子标签:根据LASSO Cox的回归系数建立一个lncRNA分子标签,lncRNA分子标签=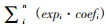
(4)评估lncRNA分子标签与预后关系:采用Cox回归分析单个lncRNA分子标签及多个因素对预后的影响。
(5)评估构建分子标签的lncRNA在中国肝细胞癌病例癌和癌旁表达水平的差异:从GEO数据库中下载中国肝细胞癌病例癌和癌旁组织有关lncRNA表达数据进行比较,评估有关lncRNA用于中国肝细胞癌人群的可能性。
3.统计学分析:采用R语言及SAS 9.4软件进行统计学分析。组间比较采用t、χ2、非参数检验等方法。LASSO Cox采用R语言中的glmnet及survival软件包进行。生存分析采用Kaplan-Meier生存曲线、log-rank检验及多元Cox回归分析方法,所有统计检验采用双侧检验,以P<0.05为差异有统计学意义。
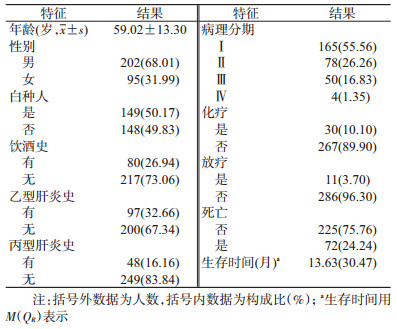
结果1.基本特征:通过TCGA数据库提取关于肝细胞癌病例肿瘤组织中lncRNA表达数据以及年龄、性别、病理分期、术后放化疗情况、生存状态等,经过数据拼接,同时具有lncRNA表达数据和完整临床数据的肝细胞癌病例有297例。其中男性202例(68.01%),女性95例(31.99%);年龄为(59.02±13.30)岁,中位生存时间为13.63个月;性别、种族、饮酒史、乙型肝炎史、丙型肝炎史、病理分期、化疗、放疗情况见表 1。
2.癌和癌旁组织的差异表达分析:通过对50例肝细胞癌病例癌和癌旁组织的lncRNA表达水平进行差异分析,癌和癌旁组织中差异倍数对数的绝对值>1、FDR<0.05的有951个lncRNA。见图 1。
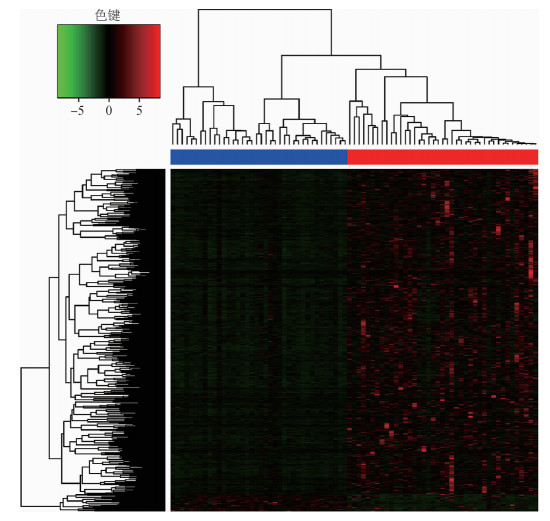
|
| 图 1 癌和癌旁组织中表达水平存在统计学差异的951个lncRNA热图 |
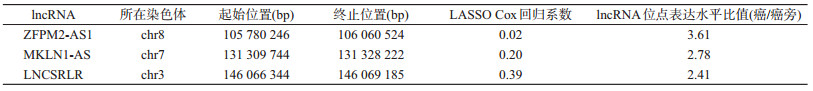
3.筛选与肝细胞癌预后相关的lncRNA:在951个癌和癌旁组织中表达水平有统计学意义差异的lncRNA中,通过LASSO Cox回归分析筛选影响肝细胞癌预后的lncRNA。最终3个lncRNA位点进入模型,分别为LNCSRLR、MKLN1-AS及ZFPM2-AS1。3个lncRNA在染色体上的位置、LASSO Cox回归分析的系数以及癌和癌旁组织中表达水平比值见表 2。
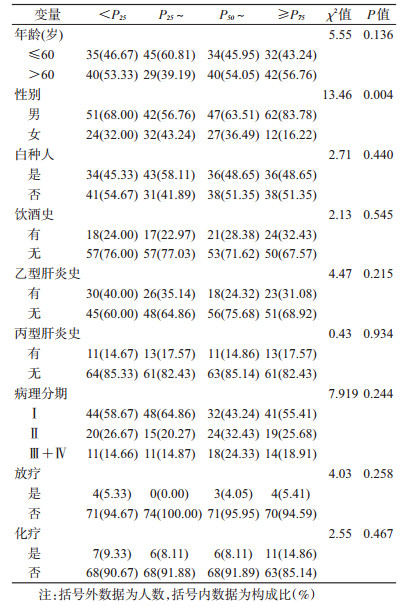
4. lncRNA分子标签的构建:用LASSO Cox回归分析筛选出肝细胞癌预后相关的3个lncRNA构建lncRNA分子标签,其计算公式:表达水平=0.02×ZFPM2-AS1+0.20×MKLN1-AS+0.39×LNCSRLR。根据癌组织中lncRNA分子标签表达水平将肝细胞癌病例划分为4组,各组人群临床特征的分布见表 3。除各组人群的性别分布差异有统计学意义外(χ2=13.46,P<0.01),其他临床特征在各组中分布差异无统计学意义。
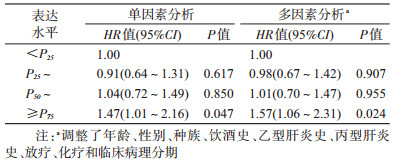
5. lncRNA分子标签与预后关系:log-rank检验显示,lncRNA分子标签不同表达水平组的生存率差异有统计学意义(P<0.01),见图 2。进一步采用LASSO Cox回归分析lncRNA分子标签表达水平对肝细胞癌病例生存的影响,结果显示,在调整年龄、性别、种族、饮酒史、乙型肝炎史、丙型肝炎史、放疗、化疗和临床病理分期协变量之后,lncRNA分子标签表达水平最高(≥P75)人群的死亡风险是分子标签表达水平最低(<P25)人群的1.57倍(95%CI:1.06~2.31)。见表 4。
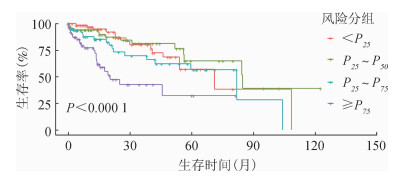
|
| 图 2 lncRNA分子标签不同表达水平组肝细胞癌病例的Kaplan-Meier生存曲线 |
6. 3个lncRNA在中国肝细胞癌病例癌和癌旁组织中表达水平的比较:通过检索GEO数据库,检索到GSE63863数据集中提供了RNA测序的原始数据[11],通过对该数据集中12对中国肝细胞癌病例癌和癌旁组织RNA测序原始数据的重新分析,提取构成分子标签的3个lncRNA在癌和癌旁组织的表达数据。比较分析显示,3个lncRNA在癌组织的表达水平均高于癌旁组织的表达水平。癌组织中ZFPM2-AS1、MKLN1-AS和LNCSRLR表达水平是癌旁组织的4.45倍(P=0.027)、1.31倍(P=0.021)和1.11倍(P=0.569)。
讨论本研究通过对TCGA肝细胞癌数据库的挖掘,筛选出3个lncRNA MKLN1-AS、ZFPM2-AS1和LNCSRLR,由这3个lncRNA构建的分子标签表达水平与肝细胞癌病例的不良预后相关。通过对GEO数据库中国肝细胞癌病例小样本RNA测序数据的分析,结果显示,ZFPM2-AS1、MKLN1-AS在中国肝细胞癌病例癌和癌旁中表达水平差异有统计学意义。
虽然本研究发现的3个lncRNA尚未有研究报道过其对肝细胞癌发生发展的影响,但既往的相关研究发现,这些lncRNA可以影响细胞内的信号通路,进而影响肿瘤的发生发展。LNCSRLR是肿瘤分子靶向药索拉菲尼耐药相关的lncRNA,其表达上调可以明显增加肾细胞癌对该靶向药的耐药性,表明其参与了肿瘤的发生、发展过程[12]。MKLN1-AS相关的研究较少,但早期的一项全基因组的研究表明,其可能是通过对肿瘤发生发展中的关键基因的启动子功能的影响来影响肿瘤的发生发展[13]。ZFPM2-AS1在胃癌组织中表达增加,并且与胃癌的不良预后相关。进一步的研究发现ZFPM2-AS1的表达可以调节胃癌细胞的P53通路上的关键蛋白的表达,促进胃癌细胞的增殖[14]。最近的相关研究也发现该lncRNA的表达对多种肿瘤的预后有影响[15]。
与有关研究中的肝细胞癌预后相关microRNA筛选方法相比[16],本研究使用了LASSO Cox降维回归的方法,而非传统经典的Cox回归,可以更好地解决组学数据高维、低样本量的特点所带来的多重共线性问题,减少过度拟合的现象[17]。本研究在筛选肝细胞癌预后相关的lncRNA基础上,还构建了lncRNA分子标签。由于该分子标签综合了3个lncRNA的信息,显著地提高了其作为肝细胞癌预后评估的价值。相关的研究也表明,构建lncRNA分子标签比单个lncRNA可以更好地评估肿瘤的预后[18]。
本研究存在局限性。第一,未对筛选出的lncRNA进行相关的机制的研究,这些lncRNA是如何影响肝细胞癌细胞的生长和凋亡,进而影响肝细胞癌预后的机制仍有待研究;第二,虽然ZFPM2-AS1、MKLN1-AS在GEO中国肝细胞癌病例癌组织表达量高于癌旁组织,但是由于临床随访信息的缺少,未能验证构建的lncRNA标签能否预测中国肝细胞癌病例的预后,这些问题均需要后续研究进一步探讨。
综上所述,本研究利用公共数据库的挖掘,筛选并构建了新的肝细胞癌预后分子标签,为肝细胞癌的预后研究提供新的研究方向,同时本研究的策略也可用于其他肿瘤预后相关标志物的筛选参考。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
魏矿荣, 彭侠彪, 梁智恒, 等. 全球肝癌流行概况[J]. 中国肿瘤, 2015, 24(8): 621-630. Wei KR, Peng XB, Liang ZH, et al. Liver cancer epidemiology worldwide[J]. Chin Cancer, 2015, 24(8): 621-630. DOI:10.11735/j.issn.1004-0242.2015.08.A001 |
| [2] |
陈万青, 李贺, 孙可欣, 等. 2014年中国恶性肿瘤发病和死亡分析[J]. 中华肿瘤杂志, 2018, 40(1): 5-13. Chen WQ, Li H, Sun KX, et al. Report of cancer incidence and mortality in China, 2014[J]. Chin J Oncol, 2018, 40(1): 5-13. DOI:10.3760/cma.j.issn.0253-3766.2018.01.002 |
| [3] |
Ha J, Yan M, Aguilar M, et al. Race/ethnicity-specific disparities in cancer incidence, burden of disease, and overall survival among patients with hepatocellular carcinoma in the United States[J]. Cancer, 2016, 122(16): 2512-2523. DOI:10.1002/cncr.30103 |
| [4] |
Zeng HM, Chen WQ, Zheng RS, et al. Changing cancer survival in China during 2003-15:a pooled analysis of 17 population-based cancer registries[J]. Lancet Glob Health, 2018, 6(5): e555-567. DOI:10.1016/S2214-109X(18)30127-X |
| [5] |
Gao YZ, Chen G, Zeng YY, et al. Invasion and metastasis-related long noncoding RNA expression profiles in hepatocellular carcinoma[J]. Tumour Biol, 2015, 36(10): 7409-7422. DOI:10.1007/s13277-015-3408-0 |
| [6] |
Liu YR, Tang RX, Huang WT, et al. Long noncoding RNAs in hepatocellular carcinoma:Novel insights into their mechanism[J]. World J Hepatol, 2015, 7(28): 2781-2791. DOI:10.4254/wjh.v7.i28.2781 |
| [7] |
李睿, 罗云波. lncRNA及其生物学功能[J]. 农业生物技术学报, 2016, 24(4): 600-612. Li R, Luo YB. lncRNA and its biological function[J]. J Agric Biotechnol, 2016, 24(4): 600-612. DOI:10.3969/j.issn.1674-7968.2016.04.015 |
| [8] |
Tomczak K, Czerwińska P, Wiznerowicz M. The cancer genome Atlas (TCGA):an immeasurable source of knowledge[J]. Contemp Oncol (Pozn), 2015, 19(1A): A68-77. DOI:10.5114/wo.2014.47136 |
| [9] |
Edgar R, Domrachev M, Lash AE. Gene expression omnibus:NCBI gene expression and hybridization array data repository[J]. Nucleic Acids Res, 2002, 30(1): 207-210. DOI:10.1093/nar/30.1.207 |
| [10] |
Barrett T, Troup DB, Wilhite SE, et al. NCBI GEO:archive for functional genomics data sets-10 years on[J]. Nucleic Acids Res, 2011, 39(Suppl): D1005-1010. DOI:10.1093/nar/gkq1184 |
| [11] |
Zhang H, Weng XL, Ye JY, et al. Promoter hypermethylation of TERT is associated with hepatocellular carcinoma in the Han Chinese population[J]. Clin Res Hepatol Gastroenterol, 2015, 39(5): 600-609. DOI:10.1016/j.clinre.2015.01.002 |
| [12] |
Xu Z, Yang F, Wei D, et al. Long noncoding RNA-SRLR elicits intrinsic sorafenib resistance via evoking IL-6/STAT3 axis in renal cell carcinoma[J]. Oncogene, 2017, 36(14): 1965-1977. DOI:10.1038/onc.2016.356 |
| [13] |
Kimura K, Wakamatsu A, Suzuki Y, et al. Diversification of transcriptional modulation:large-scale identification and characterization of putative alternative promoters of human genes[J]. Genome Res, 2006, 16(1): 55-65. DOI:10.1101/gr.4039406 |
| [14] |
Kong FY, Deng X, Kong XY, et al. ZFPM2-AS1, a novel lncRNA, attenuates the p53 pathway and promotes gastric carcinogenesis by stabilizing MIF[J]. Oncogene, 2018, 37(45): 5982-5996. DOI:10.1038/s41388-018-0387-9 |
| [15] |
Ali MM, Akhade VS, Kosalai ST, et al. PAN-cancer analysis of S-phase enriched lncRNAs identifies oncogenic drivers and biomarkers[J]. Nat Commun, 2018, 9(1): 883. DOI:10.1038/s41467-018-03265-1 |
| [16] |
陈谦, 仇小强. 基于大数据的肝癌血管侵袭标志物分析[J]. 中华流行病学杂志, 2017, 38(4): 522-527. Chen Q, Qiu XQ. Bioinformatics on vascular invasion markers in hepatocellular carcinoma via Big-data analysis[J]. Chin J Epidemiol, 2017, 38(4): 522-527. DOI:10.3760/cma.j.issn.0254-6450.2017.04.022 |
| [17] |
闫丽娜, 覃婷, 王彤. LASSO方法在Cox回归模型中的应用[J]. 中国卫生统计, 2012, 29(1): 58-60. Yan LN, Qin T, Wang T. The application of LASSO in the Cox model[J]. Chin J Health Stat, 2012, 29(1): 58-60. DOI:10.3969/j.issn.1002-3674.2012.01.017 |
| [18] |
Zhu XQ, Tian XL, Yu CY, et al. A long non-coding RNA signature to improve prognosis prediction of gastric cancer[J]. Mol Cancer, 2016, 15: 60. DOI:10.1186/s12943-016-0544-0 |
 2019, Vol. 40
2019, Vol. 40