文章信息
- 黄育北, 宋丰举, 陈可欣.
- Huang Yubei, Song Fengju, Chen Kexin.
- 全基因组关联研究在乳腺癌筛查中的应用价值初探
- Application values of genome-wide association studies in screening for breast cancer
- 中华流行病学杂志, 2019, 40(6): 713-718
- Chinese Journal of Epidemiology, 2019, 40(6): 713-718
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.06.021
-
文章历史
收稿日期: 2018-11-21
近年来,国内外有关乳腺癌全基因组关联研究(GWAS)较多[1-10],其中5篇来自亚洲地区人群的GWAS[11-15]共发现了超过90个单核苷酸多态性(SNP)位点与乳腺癌发病风险相关。这些SNP当中有23个SNP位点与我国女性乳腺癌的风险潜在相关[16]。随着GWAS的不断开展,未来会发现更多与乳腺癌相关的SNP。与此同时,探索这些遗传易感位点在高危人群中的筛查应用价值显得尤为重要[17]。目前有不少研究开始初步探索这些SNP在乳腺癌筛查中的价值[18-21]。在开展大规模的人群筛查验证研究之前,为避免资源浪费,并初步探索这些SNP在乳腺癌筛查中的预期效果,有必要开展合理的数据模拟研究。
对象与方法1.研究对象:依据2017年中国肿瘤登记报告的2013年乳腺癌年龄别发病率[22]、2013年的中国女性年龄别的人口分布[23],以及目前已经明确的乳腺癌传统危险因素(初潮年龄早、初次分娩年龄晚、腰臀比大、乳腺癌家族史、良性乳腺疾病史)分布情况及其与乳腺癌的关联强度[24],模拟200万35~69岁中国女性人群的乳腺癌患病及危险因素分布情况。
具体模拟过程:①依据《中国卫生统计年鉴2014》报告的2013年35~69岁中国女性不同年龄段(每5岁1个年龄段)人口构成[23],计算200万女性不同年龄段的人口数,并进行模拟。②依据2013年乳腺癌年龄别发病率及不同年龄段的人口数[22],计算200万女性不同年龄段的乳腺癌例数和健康人群例数,并进行模拟。③依据Zheng等[24]报告的已经明确的乳腺癌传统危险因素(初潮年龄早、初次分娩年龄晚、腰臀比大、乳腺癌家族史、良性乳腺疾病史)分别在病例组和对照组的分布概率,采用多项分布或二项分布的方法,分别在乳腺癌患者和健康人群中模拟不同危险因素的分布。④由于人群中乳腺癌患者例数明显少于健康人群例数,因此,乳腺癌患者中危险因素的模拟分布与真实分布之间的随机误差,较健康人群中危险因素的模拟分布与真实分布之间的随机误差要大。因此,分别采用不同的标准对乳腺癌患者和健康人群中危险因素分布的随机误差进行校正。即当某一危险因素在乳腺癌患者中的模拟分布概率与既往患者的真实分布概率正负相差超过2%时,以0.05%的精度对相应危险因素在乳腺癌患者的分布概率进行重新模拟。当某一危险因素在健康人群中的模拟分布概率与既往对照人群中的真实分布概率正负相差超过0.03%时,以0.01%的精度对相应危险因素在非乳腺癌患者的分布概率进行重新模拟。⑤对重新模拟的所有人群,分别计算传统危险因素联合预测乳腺癌的受试者作业特征(ROC)曲线下面积(AUC),并计算所有AUC的均值,以最接近AUC均值的模拟人群作为最终的模拟人群。
模拟人群中各传统危险因素的分布及其与乳腺癌的关联强度见表 1。同时基于原始亚洲人群GWAS或亚洲乳腺癌联盟(Asia Breast Cancer Consortium)中所报道的SNP位点在对照组中微小等位基因的分布频率及与乳腺癌关联强度的OR值[11-15, 25],以上述传统危险因素的模拟方法进一步模拟人群中23个与我国乳腺癌风险潜在相关的SNP分布。
2. SNP初筛:采用原始GWAS中所报道的SNP位点在对照组中微小等位基因的分布频率及与乳腺癌关联强度的OR值,采用Pharoah等[20]提出的方法分别计算每个SNP对乳腺癌遗传风险的解释程度(Vi)和解释比例(FVi)。在模拟人群中,以FVi最大的SNP构建遗传风险预测的基础模型,然后按照FVi的大小依次纳入其他的SNP,分别构建23个模型。计算在原有SNP基础上加入新的SNP后,对乳腺癌风险再分类准确性的改善程度,最终选取能够同时带来显著的整合区分改善指数(integrated discrimination improvement,IDI)及净再分类改善指数(net reclassification improvement,NRI)的SNP,作为可以预测乳腺癌高危人群的目标SNP[16]。即目标SNP的初筛依据为在基础模型的基础上,加入目标SNP后,对于乳腺癌风险再分类的IDI值和NRI值所相应的P值均<0.05。
3. SNP筛查应用价值评价:依据传统乳腺癌危险因素、目标SNP,及联合传统危险因素和目标SNP,分别构建乳腺癌传统风险预测模型、遗传风险预测模型及综合风险预测模型。进一步系统评价在传统乳腺癌危险因素的基础上,加入目标SNP后,对高危人群中乳腺癌检出率、对乳腺癌总体风险预测准确性及高危人群中乳腺癌患病风险的影响。其中乳腺癌总体风险预测准确性以风险预测AUC来表示,高危人群中乳腺癌的患病风险以OR值及其95%CI表示。
4.统计学分析:采用SAS 9.3软件进行数据模拟和统计学分析,包括采用χ2分析比较两个检出率差异是否有统计学意义,采用IDI值和NRI值评价SNP对乳腺癌风险再分类准确性改善能力的影响,采用多因素logistic回归计算相应的OR值及其95%CI。采用R 3.5.1软件的ggplot2程序包(2.2.1版本)进行统计绘图;同时采用pROC1.10.0程序包计算AUC及其95%CI,并比较AUC大小。所有检验均采用双侧检验,以P<0.05为差异有统计学意义。
结果1. GWAS发现SNP对女性乳腺癌遗传风险的解释能力:23个SNP共能解释大约11.9%的中国女性乳腺癌遗传风险。23个SNP中,rs2046210所能解释的遗传风险比例最大,占总体可解释乳腺癌遗传风险的21.6%。因此,以rs2046210构建遗传风险预测的基础模型,然后按照FVi的大小依次纳入其他SNP。在200万35~69岁中国女性模拟人群中,发现其他11个SNPs(rs4784227、rs1219648、rs10771399、rs9485372、rs4973768、rs10474352、rs17817449、rs9693444、rs616488、rs16857609、rs17356907)可以显著提高乳腺癌风险再分类的准确性,IDI值从0.000 3%~0.003%不等,NRI值从5.8%~19.4%不等(均P<0.05)。因此,共有12个SNPs可以用于预测乳腺癌高危人群。见表 2。
2. GWAS发现SNP对模拟人群中女性乳腺癌检出率的影响:如果将预测风险位于P95及更高风险的人群定义为高危人群,在以6个传统乳腺癌危险因素预测的高危人群中(共计99 741人),共有177例乳腺癌患者(占所有人群中乳腺癌患者的比例为12.2%),患病率为177.46/10万。以12个目标SNP预测的高危人群中(共计100 005人),共有147例乳腺癌患者(占所有人群中乳腺癌患者的比例为10.1%),患病率为146.99/10万。在6个传统危险因素和12个目标SNP联合预测的高危人群中(共计100 000人),共有229例乳腺癌患者(占所有人群中乳腺癌患者的比例为15.8%),患病率为229.00/10万。也即采用12个目标SNP预测的乳腺癌高危人群中的乳腺癌检出率(146.99/10万)低于采用6个传统危险因素预测的乳腺癌高危人群中的检出率(177.46/10万),差异有统计学意义(P<0.001)。但如果在6个传统危险因素的基础上,加上12个目标SNP预测的乳腺癌高危人群中进行筛查,乳腺癌的检出率(229.00/10万)将提高29.0%,差异有统计学意义(P<0.001)。见图 1。
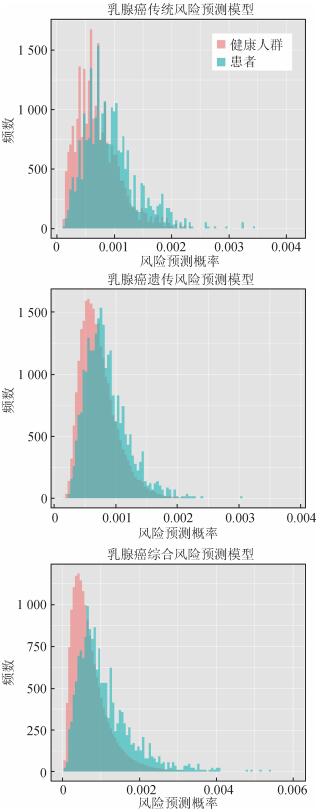
|
| 图 1 200万模拟人群中病例及对照人群乳腺癌风险预测概率的分布 |
3. GWAS发现SNP对乳腺癌总体风险预测准确性及高危人群中乳腺癌相对风险的影响:基于上述乳腺癌传统风险预测模型、遗传风险预测模型及综合风险预测模型得到的乳腺癌总体风险预测准确性的AUC分别为64.4%(95%CI:63.0%~65.8%)、61.1%(95%CI:59.6%~62.5%)及67.8%(95%CI:66.5%~69.2%)。如果以人群预测风险P45~P50的人群作为参考人群,根据12个目标SNP预测的高危人群发生乳腺癌的风险(OR=2.45,95%CI:1.82~3.31)低于6个传统危险因素预测的高危人群发生乳腺癌的风险(OR=3.32,95%CI:2.45~4.49)。联合6个传统危险因素和12个目标SNP综合预测的高危人群发生乳腺癌的风险(OR=4.33,95%CI:3.21~5.84),明显高于单纯基于传统危险因素预测的高危人群的乳腺癌相对风险。也即在6个传统危险因素的基础上,加上12个目标SNP预测高危人群,将显著提高乳腺癌总体风险预测准确性,并能明显提高高危人群中乳腺癌的相对风险(图 2)。

|
| 注:参考人群为预测风险P45~P50的人群(OR=1.00) 图 2 不同乳腺癌风险预测百分位的人群发生乳腺癌的相对风险 |
本研究采用数据模拟的办法,初步探索了GWAS发现SNP在我国女性乳腺癌筛查中的潜在应用价值。研究结果提示:在传统危险因素基础上,加入GWAS发现SNP用于预测乳腺癌高危人群,可显著提高乳腺癌检出率、乳腺癌总体风险预测准确性,并能更好地发现真正的乳腺癌患者。之前有类似研究探讨GWAS发现的SNP在乳腺癌风险预测中的潜在应用价值[17-20, 26],但大部分为理论方法研究。我国人口基数庞大、社会资源非常有限,开展合理的数据模拟研究,对未来更大规模的乳腺癌筛查实践,有很好的指导意义。
本研究在初筛目标SNP时,主要采用SNP对乳腺癌风险再分类准确性(以IDI值或NRI值评价)有显著改善的SNP,而没有直接采用SNP能对乳腺癌总体风险预测准确性的AUC有显著改善的SNP作为目标SNP。因为之前的研究提示:单一SNP对乳腺癌总体风险预测准确性的贡献很有限,因此很可能会遗漏很多潜在有价值的SNP[16, 21]。如本研究所示,在已知的传统乳腺癌危险因素的基础上,加入12个目标SNP,仅能让乳腺癌总体风险预测准确性的AUC提高3.4%。该结果与既往研究结果非常类似,包括Wacholder等[18]采用的10个SNP可在乳腺癌传统风险预测模型基础上将AUC提高3.8%,Mealiffe等[21]报告的7个SNP可将乳腺癌传统Gail风险预测模型的AUC提高3.7%。同时这些研究采用的SNP均为当时发现的所有乳腺癌风险相关SNP,并没有对相应的SNP进行初步筛查,因此对当前如何从GWAS发现的大量SNP中筛选出更有潜在人群应用价值的目标SNP的指导意义相对欠缺。
风险再分类改善指标,可以认为是风险预测准确性的相对指标。也即在原有预测变量的基础上,加入新的预测变量,只要方向上能让患者的风险重新再分类为更高风险,非患者的风险重新再分类为更低风险,即认为风险再分类发生了改善。相对于预测准确与否的二分类预测方法,这种三分类的风险再分类结果(风险上升、风险不变、风险下降)理论上能发现风险预测上更加细微的改善[27]。而且本研究提示目标SNP对乳腺癌风险再分类的改善强于对乳腺癌总体风险预测准确性的改善。在使用风险再分类改善指标初筛目标SNP,进而采用目标SNP初筛乳腺癌高危人群时,需注意以下问题:当得到初步验证的目标SNP相对较多时,须同时兼顾效果和成本两个方面来选择最终需检测的SNP,通常可进一步严格SNP的初筛标准(如将IDI值和NRI值所对应的P值下调至<0.001)。反之,当得到初步验证的目标SNP相对较少时,可放松SNP的初筛标准。
本研究存在局限性,首先,本研究是数据模拟研究,必然与真实世界研究存在一定的差异。其次,本研究选取的目标SNP只是有统计学意义的目标SNP,这些目标SNP之间是否存在生物学上的关联,目前仍在进一步的探讨中。第三,目前尚没有研究报道这些目标SNP与早期乳腺癌之间的关联,因此,本研究也无法探讨目标SNP对早期乳腺癌检出率等筛查效果指标的影响。
综上所述,本研究是一项数据模拟研究,但对我国未来开展更大规模的乳腺癌高危人群筛查,尤其是针对是否应该将GWAS发现SNP用于初筛乳腺癌高危人群,具有一定的参考价值。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Antoniou AC, Wang XS, Fredericksen ZS, et al. A locus on 19p13 modifies risk of breast cancer in BRCA1 mutation carriers and is associated with hormone receptor-negative breast cancer in the general population[J]. Nat Genet, 2010, 42(10): 885-892. DOI:10.1038/ng.669 |
| [2] |
Gaudet MM, Kirchhoff T, Green T, et al. Common genetic variants and modification of penetrance of BRCA2-associated breast cancer[J]. PLoS Genet, 2010, 6(10): e1001183. DOI:10.1371/journal.pgen.1001183 |
| [3] |
Turnbull C, Ahmed S, Morrison J, et al. Genome-wide association study identifies five new breast cancer susceptibility loci[J]. Nat Genet, 2010, 42(6): 504-507. DOI:10.1038/ng.586 |
| [4] |
Thomas G, Jacobs KB, Kraft P, et al. A multistage genome-wide association study in breast cancer identifies two new risk alleles at 1p11.2 and 14q24.1(RAD51L1)[J]. Nat Genet, 2009, 41(5): 579-584. DOI:10.1038/ng.353 |
| [5] |
Ahmed S, Thomas G, Ghoussaini M, et al. Newly discovered breast cancer susceptibility loci on 3p24 and 17q23.2[J]. Nat Genet, 2009, 41(5): 585-590. DOI:10.1038/ng.354 |
| [6] |
Stacey SN, Manolescu A, Sulem P, et al. Common variants on chromosome 5p12 confer susceptibility to estrogen receptor-positive breast cancer[J]. Nat Genet, 2008, 40(6): 703-706. DOI:10.1038/ng.131 |
| [7] |
Gold B, Kirchhoff T, Stefanov S, et al. Genome-wide association study provides evidence for a breast cancer risk locus at 6q22.33[J]. Proc Natl Acad Sci USA, 2008, 105(11): 4340-4345. DOI:10.1073/pnas.0800441105 |
| [8] |
Hunter DJ, Kraft P, Jacobs KB, et al. A genome-wide association study identifies alleles in FGFR2 associated with risk of sporadic postmenopausal breast cancer[J]. Nat Genet, 2007, 39(7): 870-874. DOI:10.1038/ng.2075 |
| [9] |
Stacey SN, Manolescu A, Sulem P, et al. Common variants on chromosomes 2q35 and 16q12 confer susceptibility to estrogen receptor-positive breast cancer[J]. Nat Genet, 2007, 39(7): 865-869. DOI:10.1038/ng.2064 |
| [10] |
Easton DF, Pooley KA, Dunning AM, et al. Genome-wide association study identifies novel breast cancer susceptibility loci[J]. Nature, 2007, 447(7148): 1087-1093. DOI:10.1038/nature05887 |
| [11] |
Cai QY, Long JR, Lu W, et al. Genome-wide association study identifies breast cancer risk variant at 10q21.2:results from the Asia breast cancer consortium[J]. Hum Mol Genet, 2011, 20(24): 4991-4999. DOI:10.1093/hmg/ddr405 |
| [12] |
Zheng W, Long JR, Gao YT, et al. Genome-wide association study identifies a new breast cancer susceptibility locus at 6q25.1[J]. Nat Genet, 2009, 41(3): 324-328. DOI:10.1038/ng.318 |
| [13] |
Cai QY, Zhang B, Sung H, et al. Genome-wide association analysis in East Asians identifies breast cancer susceptibility loci at 1q32.1, 5q14.3 and 15q26.1[J]. Nat Genet, 2014, 46(8): 886-890. DOI:10.1038/ng.3041 |
| [14] |
Long JR, Cai QY, Shu XO, et al. Identification of a functional genetic variant at 16q12.1 for breast cancer risk:results from the Asia breast cancer consortium[J]. PLoS Genet, 2010, 6(6): e1001002. DOI:10.1371/journal.pgen.1001002 |
| [15] |
Long JR, Cai QY, Sung H, et al. Genome-wide association study in east Asians identifies novel susceptibility loci for breast cancer[J]. PLoS Genet, 2012, 8(2): e1002532. DOI:10.1371/journal.pgen.1002532 |
| [16] |
黄育北, 宋丰举, 陈可欣. 乳腺癌全基因组关联研究的现状及其应用价值的分析方法[J]. 中华流行病学杂志, 2015, 36(10): 1058-1061. Huang YB, Song FJ, Chen KX. Current status of genome-wide association studies (GWAS) on breast cancer and application values of single nucleotide polymorphisms identified from GWAS[J]. Chin J Epidemiol, 2015, 36(10): 1058-1061. DOI:10.3760/cma.j.issn.0254-6450.2015.10.004 |
| [17] |
Garcia-Closas M, Gunsoy NB, Chatterjee N. Combined associations of genetic and environmental risk factors:implications for prevention of breast cancer[J]. J Natl Cancer Inst, 2014, 106(11): dju305. DOI:10.1093/jnci/dju305 |
| [18] |
Wacholder S, Hartge P, Prentice R, et al. Performance of common genetic variants in breast-cancer risk models[J]. N Engl J Med, 2010, 362(11): 986-993. DOI:10.1056/NEJMoa0907727 |
| [19] |
Gail MH. Value of adding single-nucleotide polymorphism genotypes to a breast cancer risk model[J]. J Natl Cancer Inst, 2009, 101(13): 959-963. DOI:10.1093/jnci/djp130 |
| [20] |
Pharoah PD, Antoniou AC, Easton DF, et al. Polygenes, risk prediction, and targeted prevention of breast cancer[J]. N Engl J Med, 2008, 358(26): 2796-2803. DOI:10.1056/NEJMsa0708739 |
| [21] |
Mealiffe ME, Stokowski RP, Rhees BK, et al. Assessment of clinical validity of a breast cancer risk model combining genetic and clinical information[J]. J Natl Cancer Inst, 2010, 102(21): 1618-1627. DOI:10.1093/jnci/djq388 |
| [22] |
Zuo TT, Zheng RS, Zeng HM, et al. Female breast cancer incidence and mortality in China, 2013[J]. Thoracic Cancer, 2017, 8(3): 214-218. DOI:10.1111/1759-7714.12426 |
| [23] |
国家卫生和计划生育委员会. 中国卫生和计划生育统计年鉴2014[M]. 北京: 中国协和医科大学出版社, 2014. National Health Commission of the People's Republic of China. China Health Statistics 2014[M]. Beijing: China Union Medical University Press, 2014. |
| [24] |
Zheng W, Wen WQ, Gao YT, et al. Genetic and clinical predictors for breast cancer risk assessment and stratification among Chinese women[J]. J Natl Cancer Inst, 2010, 102(13): 972-981. DOI:10.1093/jnci/djq170 |
| [25] |
Zheng W, Zhang B, Cai QY, et al. Common genetic determinants of breast-cancer risk in East Asian women:a collaborative study of 23637 breast cancer cases and 25579 controls[J]. Human Mol Genet, 2013, 22(12): 2539-2550. DOI:10.1093/hmg/ddt089 |
| [26] |
Gail MH. Discriminatory accuracy from single-nucleotide polymorphisms in models to predict breast cancer risk[J]. J Natl Cancer Inst, 2008, 100(14): 1037-1041. DOI:10.1093/jnci/djn180 |
| [27] |
Pencina MJ, D'Agostino Sr RB, D'Agostino Jr RB, et al. Evaluating the added predictive ability of a new marker:from area under the ROC curve to reclassification and beyond[J]. Stat Med, 2008, 27(2): 157-172. DOI:10.1002/sim.2929 |
 2019, Vol. 40
2019, Vol. 40




