文章信息
- 田国忠, 路殿英, 朴东日, 赵鸿雁, 杨晓雯, 姜海.
- Tian Guozhong, Lu Dianying, Piao Dongri, Zhao Hongyan, Yang Xiaowen, Jiang Hai.
- MLVA基因分型方法研究世界多地区布鲁氏菌的流行病学特征
- Epidemiological characteristics of Brucella species isolated from different regions of the world using the MLVA genotyping
- 中华流行病学杂志, 2019, 40(6): 676-681
- Chinese Journal of Epidemiology, 2019, 40(6): 676-681
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.06.014
-
文章历史
收稿日期: 2018-11-30
布鲁氏菌病(布病)是世界上导致家畜流产和人体致残性感染的最广泛的人畜共患病之一。由于发病率增加,目前被看作一种再发性传染病[1-10]。布病近年来在中国出现流行趋势[11],2013年报告了43 486例确诊病例,年发病率为3.3/10万,目前中国23个省份有布病病例报告[7, 11-14]。布鲁氏菌种型和生物型的分类主要根据其宿主特异性、生化特征进行鉴定[15]。现已鉴定出10个布鲁氏菌菌种和23种生物型,包括牛种布鲁氏菌Brucella abortus(生物1~7、9型,牛)、羊种布鲁氏菌Brucella melitensis(生物1~3型,羊)、绵羊附睾种布鲁氏菌Brucella ovis(绵羊)、犬种布鲁氏菌Brucella canis(犬)、猪种布鲁氏菌Brucella suis(生物1~5型,猪)、沙林鼠种布鲁氏菌Brucella neotomae(沙林鼠)、田鼠种布鲁氏菌Brucella microti(田鼠)、鲸种布鲁氏菌Brucella ceti(鲸目动物类)、鳍种布鲁氏菌Brucella pinnipedialis(海豹)和Brucella inopinata(人类)[15-18]。为了了解全球多地布病流行病学规律,本研究通过已公布的布鲁氏菌菌株可变数目串联重复序列(variable-number tandem-repeat,VNTR)资料进行基因分型,分析世界多地来源布鲁氏菌菌株的遗传变异规律和分子流行病学特征。
资料与方法1.菌株及资料来源:收集1953-2013年的2 747株布鲁氏菌,菌株的VNTR数据来自http://mlva.upsud.fr/数据库,包括菌株的分离国家和地区、分离的来源(宿主)、分离时间、基于生物学特征分类的布鲁氏菌菌株的种型和生物型等资料。
2.多位点串联重复序列分析(MLVA):根据文献[19-20],选择11个串联重复基因位点,包括bruce06、bruce08、bruce11、bruce12、bruce18、bruce19、bruce21、bruce42、bruce43、bruce45和bruce55,使用BioNumerics version 6.6(Applied Maths,Belgium)软件,采用非加权配对算术平均法(UPGMA)对所有数据进行聚类分析,绘制系统发育树和最小生成树,了解菌株的分子流行病学分布特征。
结果1.基本资料分析:
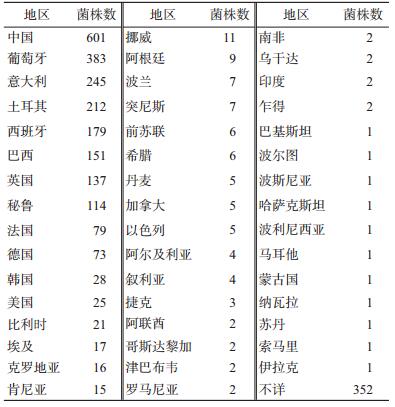
(1)地区分布:共收集全球48个国家和地区分离的2 747株布鲁氏菌,见图 1,其中主要的国家包括秘鲁、英国、巴西、西班牙、土耳其、意大利、葡萄牙和中国,分离的菌株占总数的73.57%(2 021/2 747),另有352株分离地区不详,见表 1。

|
| 图 1 布鲁氏菌系统发育树示意图 |
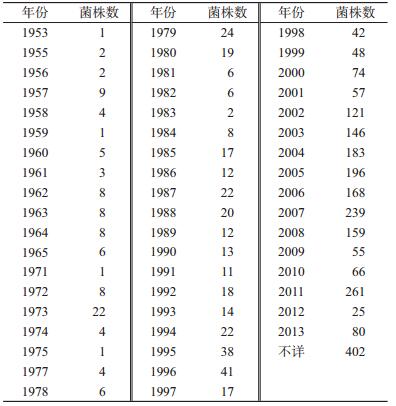
(2)时间分布:根据菌株分离时间和菌株数之间的关系,可归纳为7次高峰期,分别为:1957-1958、1962-1964、1973、1979、1987-1988、1995- 1996、2002-2008年,见表 2。
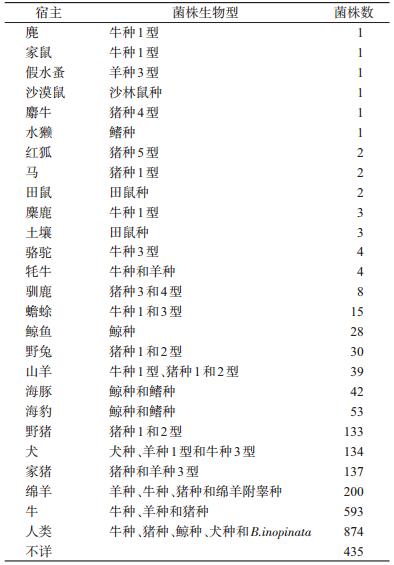
(3)来源分布:菌株的分离来源于人类和27种动物,其中人类和野猪、犬、家猪、绵羊、牛等来源菌株占75.39%(2 071/2 747),其余435株来源不详。见表 3。
(4)布鲁氏菌种和生物型分布:2 747株布鲁氏菌包括812株(29.56%)牛种布鲁氏菌(生物1~6和9型),主要是生物1和3型;1 234株(44.92%)羊种布鲁氏菌(生物1~3型),主要是1和3型;378株(13.76%)猪种布鲁氏菌(生物1~5型),主要是1、2和3型;135株(4.9%)犬种布鲁氏菌(1个生物型);94株(3.42%)鲸种布鲁氏菌(1个生物型);46株(1.67%)鳍种布鲁氏菌(1个生物型);34株(1.24%)绵羊附睾种布鲁氏菌(1个生物型);12株(0.44%)田鼠种布鲁氏菌(1个生物型);1株(0.04%)沙林鼠种布鲁氏菌(1个生物型);1株(0.04%)B. inopinata(1个生物型)。
2. MLVA:11个串联重复基因位点包括bruce06、bruce08、bruce11、bruce12、bruce18、bruce19、bruce21、bruce42、bruce43、bruce45和bruce55,使用BioNumerics version 6.6(Applied Maths,Belgium)软件,基于分类系数法和UPGMA对所有数据进行聚类分析,2 747株布鲁氏菌可分为365个MLVA11 Orsay基因型。812株牛种布鲁氏菌分为83个基因型,1 234株羊种布鲁氏菌分为159个基因型,378株猪种布鲁氏菌分为81种基因型,135株犬种布鲁氏菌分为10个基因型,94个鲸种布鲁氏菌分为6个基因型,34株绵羊附睾布鲁氏菌分为6个基因型,46株鳍种布鲁氏菌分为11个基因型,12株田鼠种布鲁氏菌分为7个基因型,1株沙林鼠种布鲁氏菌和1株B. inopinata株分别代表 1个基因型。
3.布鲁氏菌系统发育树:为了简化布鲁氏菌系统发育树的描述,将布鲁氏菌种和生物型、MLVA11 Orsay基因型以及同一基因型内菌株数重组为1个新的变量,建立了包括基因型在同一种内连续编号,如:“A1(A19,11)”:A1代表牛种布鲁氏菌生物1型,A19代表在牛种布鲁氏菌中基因型为19,11代表该基因型的菌株为11株,其他如M为羊种,M2or3为羊种生物2或3型,S为猪种,O为绵羊附睾种,Ca为犬种,Ce为鲸种,Mi为田鼠种,P为鳍种,In为Brucella inopinata,Ne为沙林鼠种。2 747株布鲁氏菌系统发育树,见图 1。10个布鲁氏菌种和23个生物型具有独立的支系。
4.分子流行病学分布规律:为了简化布鲁氏菌分子流行病学分布的描述,在流行病学分布(Brucella Epidemiological Distribution)图中,对变量进行了重组简化,如“China [A3](18),1990- 1995”:China为地名,[A3]为牛种生物3型,(18)为菌株数,1990-1995为分离时间。其他如A为牛种,M为羊种,M2or3为羊种生物2或3型,S为猪种,Ovis为绵羊附睾种,Canis为犬种,Ceti为鲸种,Micro为田鼠种,Pin为鳍种,Ino为B. inopinata,Neo为沙林鼠种。2 747株布鲁氏菌可归纳为2个大分支和8个群。第一分支包括5大群:群1:绵羊附睾种布鲁氏菌,群2:猪种布鲁氏菌(生物1、2、3和4型)和犬种布鲁氏菌,群3:鲸种布鲁氏菌,群4:鲸种、沙林鼠种、鳍种、田鼠种和B. inopinata种布鲁氏菌,群5:猪种布鲁氏菌生物5型;第二分支包括3大群:群6:牛种布鲁氏菌,群7:羊种布鲁氏菌(生物2和3型),群8:羊种布鲁氏菌(生物1型)。
(1)绵羊附睾种布鲁氏菌:群1内包括34株绵羊附睾种布鲁氏菌,分离自1974-1978年的巴西、法国和阿根廷,菌株之间的遗传关系较近。
(2)猪种布鲁氏菌:群2和群5包括了378株猪种布鲁氏菌,其中群2可分为4个亚群:亚群2.1是猪种生物2型179株,分离自2000-2005年的葡萄牙和西班牙,以及2000-2003年的克罗地亚、德国和法国;亚群2.2是猪种生物1型29株菌株,分离自1987-1988年的法国、比利时、德国和丹麦;亚群2.3是猪种生物1型菌株,分离自1973-1979年的中国(28株),以及1980-1987和1992-1998年的波兰、美国和克罗地亚31株菌株;亚群2.4内173株菌株,包括加拿大和美国的猪种生物4型9株菌株,和1964-1973年的中国和印度的猪种生物3型29株菌株;亚群2.4内还包括来自中国和韩国的135株犬种布鲁氏菌。群5是生物5型3株菌株,分离自前苏联。
(3)犬种布鲁氏菌:群4包括135株犬种布鲁氏菌,分离自1986-1987、2003-2007和2011年的中国和韩国。
(4)鲸种、鳍种、田鼠种、沙林鼠种和B.inopinata种布鲁氏菌:鲸种、鳍种、田鼠种、沙林鼠种和B. inopinata种布鲁氏菌具有同源性。94株鲸种布鲁氏菌分布在群3和群4内,群3内27株分离自1999-2002和2005-2007年的苏格兰;群4内67株分离自2002-2005年的苏格兰。12株田鼠种布鲁氏菌分离自奥地利和乍得共和国,46株鳍种布鲁氏菌分离自1993-1994和2002年的德国和苏格兰。沙林鼠种和B. inopinata各1株菌株,为群4内的一组。
(5)牛种布鲁氏菌:所有的牛种布鲁氏菌均在群6内,群6又可细分为4个亚群。亚群6.1主要是牛种生物3、6和9型58株菌株,来自巴西的50株菌株与比利时、英国和中国的8株有差别;亚群6.2是牛种生物1型583株,是牛种主要流行株,占所有牛种菌株的71.80%,其中,中国和巴西有79株菌具有同源性,韩国、英国、意大利和葡萄牙有322株菌株具有同源性,巴西和葡萄牙除了有本地特征菌株外,还有和其他欧洲地区的国家具有同源性的菌株,2006-2008年分离自巴西的13株牛种生物1型菌株具有同源性;亚群6.3包括牛种生物3型211株,中国76株生物3型菌株与其他国家有差异,其中1980-1985、1990-1995和1953-1973、1979-1985年的菌株有差异;意大利、葡萄牙、法国和德国分离的135株菌株具有同源性;亚群6.4分离自肯尼亚和乌干达的牛种生物3或6型16株菌,独成一亚群。
(6)羊种布鲁氏菌:羊种布鲁氏菌所有菌株分布在群7和群8内。羊种生物1型菌株与羊种生物2和3型菌株有明显差别。群7可以细分为5个亚群。亚群7.1和亚群7.2内27株羊种生物3型菌株分离2013年的中国;亚群7.2和亚群7.3内68株菌株,包括2013年中国菌株的6株和2003-2008年分离自西班牙的62株菌株,具有同源性;亚群7.4内共有768株菌株,占所有菌株的62.24%,包括了本研究菌株来源的所有国家和地区,是羊种布鲁氏菌的流行株;亚群7.5内168株菌株,主要来自法国、德国、意大利和瑞士等,2005-2011年来自意大利和瑞士的56株菌株具有同源性,2005-2011年来自意大利、法国、瑞士、突尼斯和德国的106株菌株具有同源性。群8又可细分为2个亚群,包括了羊种生物1型所有菌株,亚群8.1内140株来自2000-2009年的秘鲁;亚群8.2内的59株分别来自葡萄牙(22株)和西班牙(37株)具有同源性,但是国家间有差异。
5.中国分离的布鲁氏菌:中国布鲁氏菌流行株主要是羊种,其次为牛种,主要来自于北部畜牧业为主的地区。猪种布鲁氏菌主要是猪种1和3型,分布于中国南部地区,而欧洲地区主要是猪种2型。犬主要分离自犬类,无人类感染,我国的犬种布鲁氏菌与邻国韩国具有相同的基因型。
讨论以噬菌体裂解试验、生长时CO2的需求、硫堇和复红抑菌试验为基础的布鲁氏菌种型和生物型的分类是布鲁氏菌分型的经典方法,但是该方法不能显示菌株的布鲁氏菌遗传变异和流行病学分布规律[21-22]。基因分型常用于分子流行病学调查,并可用于观察种群之间的关系。VNTR分析对近缘菌株的种类鉴定和疫情暴发同源性追踪方面具有较高的鉴别能力[4-8, 19-20]。目前用于布鲁氏菌基因分型的主要有16个位点,包括bruce04、bruce07、bruce09、bruce16、bruce30、bruce06、bruce08、bruce11、bruce12、bruce18、bruce19、bruce21、bruce42、bruce43、bruce45和bruce55。其中bruce04、bruce07、bruce09、bruce16和bruce30等位点,由于其变异系数大,适合疫情暴发的菌株溯源,而bruce06、bruce08、bruce11、bruce12、bruce18、bruce19、bruce21、bruce42、bruce43、bruce45和bruce55等位点(MLVA11 Orsay)的变异系数小,适合研究布鲁氏菌的进化关系和流行病学特征[5, 21, 23]。MLVA11 Orsay基因分型与MLVA71 Orsay基因分型[19]、布鲁氏菌特异性IS711分子分析[25],以及基于全基因组的系统发育研究具有相似性[24]。因此,本研究采用MLVA11 Orsay方法,通过布鲁氏菌菌株大数据资料,分析布鲁氏菌的进化特征和全球多地区的布鲁氏菌分子流行病学特征研究。
聚类分析表明,以11个位点bruce06、bruce08、bruce11、bruce12、bruce18、bruce19、bruce21、bruce42、bruce43、bruce45和bruce55的MLVA11 Orsay聚类分析,与布鲁氏菌菌株分离的时间、地域和宿主具有较好的一致性。2002-2008年分离了1 212株菌株,占所有株菌的44.12%(1 212/2 747),而1953-1999年47年间只分离到18.75%(515/2 747)菌株,提示2002-2008年是布病流行期。所有菌株中以牛种和羊种布鲁氏菌为主,牛种中以生物1和3型为主,羊种中以生物2和3型为主,动物中以犬种、鲸种、鳍种和田鼠种布鲁氏菌为主。分离来源主要是人、牛、绵羊、山羊、犬、野猪、海豹、海豚、野兔和鲸鱼等。菌株分离地区主要有中国、葡萄牙、意大利、土耳其、西班牙、巴西、英国和秘鲁等。中国羊种布鲁氏菌与土耳其、黎巴嫩、以色列和德国同源性较近,牛种生物1型与巴西和葡萄牙同源性较近,牛种生物3型具有中国地理特异性,中国猪种布鲁氏菌以生物1和3型为主,并且具有地域特异性,尽管波兰、美国和克罗地亚也有生物1型菌株,但与中国菌株有基因型上的差异,中国分离的生物3型与印度分离的菌株具有同源性。欧洲地区猪种菌主要是生物2型为主,且葡萄牙和西班牙具有极高的同源性;犬种菌主要是来自中国和韩国,而且具有同源性。从海洋哺乳动物中分离的菌株具有密切的亲缘关系,如鲸种和鳍种布鲁氏菌菌株。从聚类分析图可以看出,分离自前苏联的猪种生物5型3株菌株,与猪种其他生物型遗传关系较远,可以从猪种中分离出来,独成一个新种。犬种布鲁氏菌与猪种生物4型菌株相似,可以归在一个种内。沙林鼠种与田鼠种布鲁氏菌关系较近,而分离自人体内的1株B. inopinata和鳍种布鲁氏菌关系较近,它们可以合并成一个种型。同样来自苏格兰的鲸种菌株,1999-2002和2005-2007年分离的27株菌株,与2002-2005年分离的67株菌株遗传关系较远,而且与其他海洋动物分离的鳍种关系较远,这27株鲸种菌株似乎可以独成一个布鲁氏菌种型。羊种生物2和3型菌株应用该分型方法不能区分,实际上应用其他分型方法也有相似的结果,他们之间具有极高的相似性,可以归在同一生物型。牛种生物3和6型,生物5和9型基因型相似度较高。来自肯尼亚和乌干达分离的16株牛种生物3或6型菌株,与其他牛种菌株基因关系较远,可以独立成一个牛种新型。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Pappas G, Papadimitriou P, Akritidis N, et al. The new global map of human brucellosis[J]. Lancet Infect Dis, 2006, 6(2): 91-99. DOI:10.1016/S1473-3099(06)70382-6 |
| [2] |
Seleem MN, Boyle SM, Sriranganathan N. Brucellosis:a re-emerging zoonosis[J]. Vet Microbiol, 2010, 140(3/4): 392-398. DOI:10.1016/j.vetmic.2009.06.021 |
| [3] |
Mailles A, Rautureau S, Le Horgne JM, et al. Re-emergence of brucellosis in cattle in France and risk for human health[J]. Euro Surveill, 2012, 17(30): 20227. |
| [4] |
Ferreira AC, Chambel L, Tenreiro T, et al. MLVA16 typing of Portuguese human and animal Brucella melitensis and Brucella abortus isolates[J]. PLoS One, 2012, 7(8): e42514. DOI:10.1371/journal.pone.0042514 |
| [5] |
Garofolo G, di Giannatale E, de Massis F, et al. Investigating genetic diversity of Brucella abortus and Brucella melitensis in Italy with MLVA-16[J]. Infect Genet Evol, 2013, 19: 59-70. DOI:10.1016/j.meegid.2013.06.021 |
| [6] |
Minharro S, Silva Mol JP, Dorneles EM, et al. Biotyping and genotyping (MLVA16) of Brucella abortus isolated from cattle in Brazil, 1977 to 2008[J]. PLoS One, 2013, 8(12): e81152. DOI:10.1371/journal.pone.0081152 |
| [7] |
Jiang H, Wang H, Xu LQ, et al. MLVA genotyping of Brucella melitensis and Brucella abortus isolates from different animal species and humans and identification of Brucella suis vaccine strain S2 from cattle in China[J]. PLoS One, 2013, 8(10): e76332. DOI:10.1371/journal.pone.0076332 |
| [8] |
Her M, Kang SI, Cho DH, et al. Application and evaluation of the MLVA typing assay for the Brucella abortus strains isolated in Korea[J]. BMC Microbiol, 2009, 9: 230. DOI:10.1186/1471-2180-9-230 |
| [9] |
Sanogo M, Abatih E, Thys E, et al. Importance of identification and typing of Brucellae from West African cattle:a review[J]. Vet Microbiol, 2013, 164(3/4): 202-211. DOI:10.1016/j.vetmic.2013.02.009 |
| [10] |
Muendo EN, Mbatha PM, Macharia J, et al. Infection of cattle in Kenya with Brucella abortus biovar 3 and Brucella melitensis biovar 1 genotypes[J]. Trop Anim Health Prod, 2012, 44(1): 17-20. DOI:10.1007/s11250-011-9899-9 |
| [11] |
Shang DQ, Xiao DL, Yin JM. Epidemiology and control of brucellosis in China[J]. Vet Microbiol, 2002, 90(1/4): 165-182. DOI:10.1016/S0378-1135(02)00252-3 |
| [12] |
Zhang WY, Guo WD, Sun SH, et al. Human brucellosis, inner Mongolia, China[J]. Emerg Infect Dis, 2010, 16(12): 2001-2003. DOI:10.3201/eid1612.091081 |
| [13] |
Li ZJ, Cui BY, Chen H, et al. Molecular typing of Brucella suis collected from 1960s to 2010s in China by MLVA and PFGE[J]. Biomed Environ Sci, 2013, 26(2): 504-508. DOI:10.3967/0895-3988 |
| [14] |
Di DD, Cui BY, Wang H, et al. Genetic polymorphism characteristics of Brucella canis isolated in China[J]. PLoS One, 2014, 9(1): e84862. DOI:10.1371/journal.pone.0084862 |
| [15] |
Ulu-Kilic A, Metan G, Alp E. Clinical presentations and diagnosis of brucellosis[J]. Recent Pat Antiinfect Drug Discov, 2013, 8(1): 34-41. DOI:10.2174/1574891X11308010007 |
| [16] |
Foster G, Osterman BS, Godfroid J, et al. Brucella ceti sp. nov. and Brucella pinnipedialis sp. nov. for Brucella strains with cetaceans and seals as their preferred hosts[J]. Int J Syst Evol Microbiol, 2007, 57(11): 2688-2693. DOI:10.1099/ijs.0.65269-0 |
| [17] |
Scholz HC, Hubalek Z, Sedláček I, et al. Brucella microti sp. nov., isolated from the common vole Microtus arvalis[J]. Int J Syst Evol Microbiol, 2008, 58(Pt 2): 375-382. DOI:10.1099/ijs.0.65356-0 |
| [18] |
Scholz HC, Nöckler K, Göllner C, et al. Brucella inopinata sp. nov., isolated from a breast implant infection[J]. Int J Syst Evol Microbiol, 2010, 60(Pt 4): 801-808. DOI:10.1099/ijs.0.011148-0 |
| [19] |
Le Flèche P, Jacques I, Grayon M, et al. Evaluation and selection of tandem repeat loci for a Brucella MLVA typing assay[J]. BMC Microbiol, 2006, 6: 9. DOI:10.1186/1471-2180-6-9 |
| [20] |
Maquart M, Le Flèche P, Foster G, et al. MLVA-16 typing of 295 marine mammal Brucella isolates from different animal and geographic origins identifies 7 major groups within Brucella ceti and Brucella pinnipedialis[J]. BMC Microbiol, 2009, 9: 145. DOI:10.1186/1471-2180-9-145 |
| [21] |
Al Dahouk S, Le Flèche P, Nöckler K, et al. Evaluation of Brucella MLVA typing for human brucellosis[J]. J Microbiol Methods, 2007, 69(1): 137-145. DOI:10.1016/j.mimet.2006.12.015 |
| [22] |
Whatmore AM. Current understanding of the genetic diversity of Brucella, an expanding genus of zoonotic pathogens[J]. Infect Genet Evol, 2009, 9(6): 1168-1184. DOI:10.1016/j.meegid.2009.07.001 |
| [23] |
Tian GZ, Cui BY, Piao DR, et al. Multi-locus variable-number tandem repeat analysis of Chinese Brucella strains isolated from 1953 to 2013[J]. Infect Dis Poverty, 2017, 6: 89. DOI:10.1186/s40249-017-0296-0 |
| [24] |
Foster JT, Beckstrom-Sternberg SM, Pearson T, et al. Whole-genome-based phylogeny and divergence of the genus Brucella[J]. J Bacteriol, 2009, 191(8): 2864-2870. DOI:10.1128/JB.01581-08 |
| [25] |
Audic S, Lescot M, Claverie JM, et al. The genome sequence of Brucella pinnipedialis B2/94 sheds light on the evolutionary history of the genus Brucella[J]. BMC Evol Biol, 2011, 11: 200. DOI:10.1186/1471-2148-11-200 |
 2019, Vol. 40
2019, Vol. 40