文章信息
- 李吉, 方圆, 肖千一, 高莹, 徐望红.
- Li Ji, Fang Yuan, Xiao Qianyi, Gao Ying, Xu Wanghong.
- DNA甲基化在女性出生体重与成年期肥胖风险关联中的中介效应
- Mediation effect of DNA methylation in associations between birth weight and adulthood obesity in women in China
- 中华流行病学杂志, 2019, 40(5): 590-595
- Chinese Journal of Epidemiology, 2019, 40(5): 590-595
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2019.05.019
-
文章历史
收稿日期: 2018-10-23
2. 中国科学院上海生命科学研究院营养科学研究所 200031
2. Shanghai Institute of Nutrition and Health, Shanghai Institutes for Biological Sciences, Chinese Academy of Sciences, Shanghai 200031, China
肥胖已成为我国及世界各国的重大公共卫生问题。除膳食、体力活动等生活方式影响肥胖的发生外,生命早期尤其是胚胎时期的营养状况对成年期肥胖及其他代谢异常的发生有重要影响。作为反映宫内不良暴露的重要指标,低和高出生体重均与成年后全身性肥胖或腹部肥胖的高风险有关[1-6]。国外研究提示,高或低出生体重可能伴随DNA甲基化水平的改变,从而影响肥胖相关基因的表达[7-8]。一些基因特异位点的甲基化水平可能作为后期肥胖和MS等发生的预测因子[9]。本研究采用中介分析方法,探索DNA甲基化在女性出生体重与成年期肥胖风险关联中的作用。
对象与方法1.研究对象:2016年3-12月在上海市闵行区13家社区卫生服务中心招募志愿者及部分家庭成员开展横断面调查。纳入标准:上海市闵行区常住女性居民;有直系血缘关系的2~4代女性家庭。排除标准:无血缘关系(婆媳、养母和养女等)或无连续直系血缘关系(仅外祖母和外孙女参加);妊娠期女性;调查当时患有严重疾病者,包括已确诊患有恶性肿瘤、痴呆、智力低下、精神病、长期瘫痪卧床、身体极度虚弱或有严重的心脑血管疾病。共调查2 147名女性。本次调查通过了复旦大学公共卫生学院伦理委员会审查。所有研究对象均在进行实验室检查和问卷调查前签署知情同意书。
2.数据收集:数据采集由经过统一培训的社区医生,使用结构式问卷,采用一对一面访调查与身体测量相结合的方式进行。问卷调查收集的信息包括一般人口学信息(包括出生年月、文化程度等)、出生时信息(包括孕周、出生身长及出生体重)、当前生活方式状况(吸烟、饮酒、体育运动情况)及常见慢性疾病史(高血压、糖尿病、冠心病等)。对≥20岁的研究对象测量身高、体重、腰围(WC)和臀围。基于测量数据,分别计算BMI、腰臀比(WHR)和腰围身高比(WHtR)。
3.生物标本采集:在知情自愿情况下,使用抗凝管共抽取2 047名研究对象的静脉血,于4 ℃冰箱中临时保存,并于当日送至实验室分装,置-80 ℃冰箱中长期保存。
4.候选基因位置及基因序列:通过检索PubMed、Web of Science等数据库,寻找既往报道的与宫内营养不良相关的基因甲基化位点,综合考虑基因类型(父源印记、母源印记或非印记基因)、甲基化敏感转录因子的结合、DNA甲基化与基因表达的关联、印记位点差异甲基化区(differentially methylated region,DMR)等因素。最终选择鸟苷酸结合蛋白活性刺激肽反义RNA1(GNASAS)、胰岛素样生长因子2(IGF2)、胰岛素样生长因子2受体(IGF2-R)3个印记基因,白介素10(IL10)和瘦素(LEP)2个非印记基因进行分析。
5.实验检测:外周血DNA抽提使用天根生化科技(北京)有限公司DP348血液基因组DNA提取试剂盒(型号:DP348),按照试剂盒说明书进行操作。去除质量不符合要求的DNA样本后,最终对1 717人的DNA样本进行了甲基化检测,本研究仅对≥20岁的1 602名女性进行分析。DNA经过亚硫酸氢盐转化后,进行PCR扩增。扩增引物由上海境像生物科技有限公司设计提供,5对PCR引物序列见表 1。采用次世代定序(Next Generation Sequencing,NGS)测序方法,由上海境像生物科技有限公司对IL10基因CpG1~4、GNASAS基因CpG1~19、LEP基因CpG2~32、IGF2-R CpG1~22及IGF2基因DMR(IGF2-DMR)CpG1~8进行DNA甲基化水平测定。
样本转化效率为(99.26±2.12)%。测序错误率与碱基质量有关,受测序仪、测序试剂、样品等因素共同影响。通常测序序列(sequenced reads)5’端及3’端前几个碱基的错误率相对较高,本研究测序为2×150 bp双端测序(illumina hiSeq X)。利用FastQC工具分析测序获得原始序列的质量情况,并对所有样本的两端读序分别进行质控评估。采用TrimGalore工具进行序列过滤,去除接头引物以及低质量序列;采用Bowtie2 v2.3.4软件进行基因比对、Bismark v0.19.0软件对结果进行解读。
6.统计学分析:采用EpiData 3.0软件进行数据录入和逻辑校错、SAS 9.4软件进行数据分析、Mplus 7.4软件进行中介分析。将研究对象拆分成804对母女进行分析,其中792对来自二代户家庭,12对来自于6个三代户家庭。运用配对t检验与配对χ2检验比较母女两代的基本特征。采用结合了限制性立方样条函数(restricted cubic spline,RCS)的广义估计方程(generalized estimating equations,GEE)模型[10],分析出生体重与女性成年期BMI、WC、WHR和WHtR的关联。RCS采用5个百分位数(P5、P25、P50、P75、P95)作为节点。以家庭作为第二水平,个体为第一水平,采用多水平回归模型分别估计出生体重及肥胖指标与各特异CpG位点甲基化水平的关联。对与出生体重及成年期肥胖指标均有显著关联的甲基化位点进行基于多水平结构方程模型(multilevel structural equation model,MSEM)的中介效应分析。所有检验均为双侧,P<0.05为差异有统计学意义。
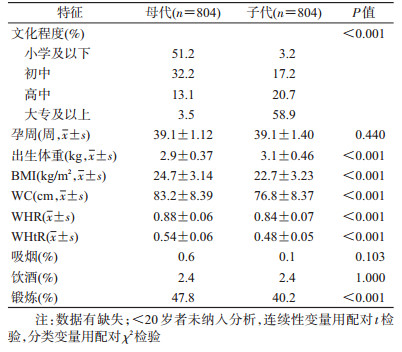
结果1.基本信息:母代年龄为(66.1±9.62)岁,子代为(40.8±9.74)岁。与子代相比,母代的平均出生体重较低,BMI、WC、WHR、WHtR较高、文化程度较低、参加锻炼的比例较高(P<0.05)。两代在孕周、吸烟和饮酒方面差异无统计学意义(表 2)。
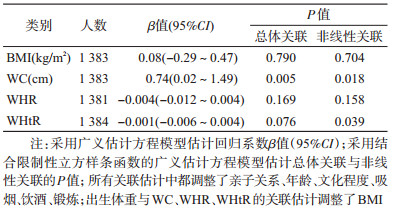
2.出生体重与肥胖、腹部肥胖的关联:采用结合RCS的GEE模型,调整亲子关系、年龄、文化程度、吸烟、饮酒、锻炼和BMI后,分析结果显示,出生体重与WC在总体上有显著关联(P=0.005),且呈显著的非线性关联(P=0.018)。当出生体重低于2 650 g时,WC随着出生体重的增加呈下降趋势,当出生体重在2 650~3 100 g之间时,WC呈上升趋势,当出生体重在3 101~3 500 g之间时,WC下降,出生体重高于3 500 g时又呈缓慢上升趋势。未提示出生体重与BMI、WHR和WHtR在总体上有统计学关联(表 3)。
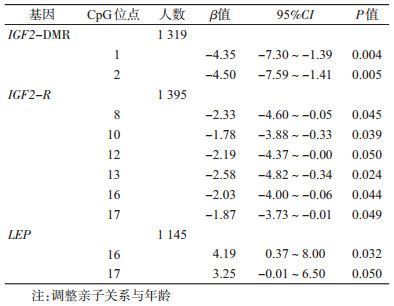
3.出生体重与特异性基因位点甲基化水平的关联:分别以5个基因上84个CpG位点的甲基化水平为因变量,采用多水平回归模型,分析出生体重与其的关联。出生体重与IGF2-DMR CpG1、2,IGF2-R CpG8、10、13、16、17的甲基化水平呈显著的负向关联,出生体重每增加1 kg,这些位点甲基化水平分别降低1.78%~4.50%。出生体重与LEP CpG16的甲基化水平呈显著的正向关联。见表 4。
进一步将研究对象按出生体重分为低(<2 500 g)、正常(2 500~3 999 g)和高(≥4 000 g)出生体重组,比较各组甲基化水平。分别以各基因特定CpG位点的甲基化水平为因变量,不同出生体重组为自变量,建立多水平回归模型。调整亲子关系与年龄后,分析结果显示,低出生体重组IGF2-DMR CpG2的甲基化水平明显高于正常体重组,回归系数β值为6.38(95%CI:0.43~12.34),其余位点各出生体重组甲基化水平差异无统计学意义(表 5)。
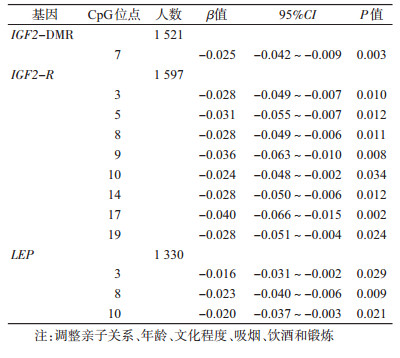
4.特异性基因位点甲基化水平与WC的关联:将5个基因中84个CpG位点的甲基化水平分别作为自变量纳入多水平回归模型,分析其与WC的关联。结果显示,共12个CpG位点的甲基化与WC有关,分别为IGF2-DMR CpG7,IGF2-R CpG3、5、8、9、10、14、17和19,以及LEP CpG3、8和10。见表 6。
5. DNA甲基化在出生体重与WC关联中的可能中介效应:IGF2-R CpG8和17的甲基化水平与出生体重及成年后WC大小均有显著关联,将其分别纳入基于MSEM的1-1-1模型进行多水平中介效应分析。调整亲子关系、年龄、文化程度、吸烟、饮酒和锻炼等可能的混杂因素,且区分了家庭因素的影响后,在个体层面上,出生体重与WC呈显著的正向关联[β=1.09(95%CI:0.55~1.63)],IGF2-R CpG8的甲基化水平与出生体重[β=-1.85(95%CI:-2.36~-1.34)]和WC[β=-0.02(95%CI:-0.03~-0.01)]均呈显著的负向关联。出生体重通过IGF2-R CpG8甲基化对WC产生的间接效应为0.04(95%CI:0.02~0.06),中介效应有统计学意义(P<0.001),IGF2-R CpG8甲基化可以解释出生体重与WC关联的3.3%。但这种中介效应在家庭层面上未达显著水平。
在IGF2-R CpG17介导的中介效应分析中,在个体层面上,出生体重与WC[β=1.07(95%CI:0.44~1.70)]及IGF2-R基因CpG17的甲基化水平[β=-2.02(95%CI:-3.70~-0.01)]均呈显著关联;在家庭层面,IGF2-R基因CpG17的甲基化水平与出生体重呈显著关联[β=-6.71(95%CI:-7.52~-5.90)];但无论在个体还是家庭层面,均未提示IGF2-R CpG17的甲基化水平在出生体重与WC关联中有显著的中介效应。
讨论中心性肥胖又称腹型肥胖,反映了身体脂肪的分布异常,常采用WC、WHR和WHtR作为指标进行衡量。国外有研究显示,出生体重与老年女性全身性肥胖(BMI)存在非线性关联[11]。前期我们在上海地区女性中的一项研究也显示,出生体重与全身性肥胖(BMI)和腹部肥胖(WC、WHR、WHtR)均呈显著的非线性关联[12]。本研究虽未显示出生体重与成年后BMI和WHR有显著关联,但关联方向与我们的前期研究一致[12]。
本研究结果未显示出生体重与GNASAS基因特异位点甲基化水平有显著关联,与Tobi等[13]的研究一致,也未显示出生体重与IL10基因特异位点甲基化水平有显著关联,但结果提示,出生体重与IGF2-DMR、LEP基因特异位点的甲基化水平有显著关联。关联方向与Steegers-Theunissen等[14]的研究一致,均观察到出生体重与IGF2-DMR甲基化水平呈负向关联。研究结果提示宫内营养可能影响IGF2-DMR的甲基化水平。
IGF2-R是一种印记基因,对人体出生前后的生长和发育都非常重要[15-16],动物实验研究提示,胚胎的过度生长与IGF2-R甲基化水平的下降有关[17-18]。本研究多水平中介效应分析结果显示,IGF2-R基因CpG8的甲基化可能是出生体重与WC关联的中介因素,甲基化作用可以解释两者的关联的3.3%。但这种中介作用只在个体水平有统计学意义,在家庭水平上的关联未达显著水平,提示该位点在母女两代之间可能不存在传递效应。对母女两代IGF2-R基因CpG8的甲基化水平进行相关分析,结果显示,两者呈正向关联,但未达显著水平,这与多水平中介分析的结论一致。若仅用单水平中介模型进行分析,则中介效应增加至8.3%。可见,尽管IGF2-R基因CpG8甲基化在家庭水平上无显著的中介效应,但家庭水平上间接效应的混杂作用不可忽视。此外,表观遗传漂移学说认为,DNA甲基化水平会随年龄发生改变,但这种变化通常具有基因或组织特异性[19]。Talens等[20]的研究结果显示,某些CpG位点具有时间稳定性,而且印记基因较非印记基因更加稳定。IGF2、IGF2-R都是印记基因,均具有一定的稳定性。本研究未发现这两个印记基因的甲基化水平在母女之间传递。
本研究存在局限性。首先,本研究属于横断面调查,在揭示甲基化作用与肥胖的因果关联可能存在一些局限性。其次,本研究中出生体重多为自我报告所得,可能存在较大的回忆偏倚。再者,本次调查人群年龄跨度比较大(20~93岁),而DNA甲基化会随年龄发生改变。不能排除年龄等因素对研究结果的混杂效应。此外,白细胞构成也可能影响甲基化检测。本研究未能检测白细胞构成情况,可能使研究结果发生偏倚。然而,尽管某些CpG甲基化与血细胞构成有关,但大多数CpG及全基因组甲基化似乎并不受影响[21]。Dick等[22]的研究结果显示,调整了淋巴细胞、中性粒细胞、单核细胞计数后并未减弱DNA甲基化与BMI的关联。Talens等[23]的研究结果显示,血液中性粒细胞比例与IGF2-R、IGF2、GNASAS的甲基化水平无关,但与IL10、LEP的甲基化水平有关。可见,本研究中检测的IGF2-R、IGF2、GNASAS甲基化水平可能并不受此影响。最后,DNA甲基化具有组织特异性,而本次检测了外周血DNA甲基化,未检测组织(如脂肪细胞)中DNA甲基化水平。Crujeiras等[24]的研究结果显示,只检测血液而非脂肪细胞的DNA甲基化水平可能会错过与肥胖真正关联的特异性位点。但也有研究显示血液可以代替脂肪组织用于研究某些特异位点[25]。Slieker等[26]的研究结果显示,血液和脂肪都来源于中胚层,许多位点的甲基化水平具有相关性。Tobi等[27]对12种死后组织进行分析,结果显示,血液与网膜、皮下脂肪、肾脏周围摄取脂肪中的DNA甲基化水平相关。Wahl等[28]的研究结果显示,血液与皮下和网膜脂肪、肝脏、肌肉、脾脏和胰腺细胞中DNA甲基化水平相关,脂肪组织和血液中DNA甲基化水平与BMI关联的方向一致。
综上所述,本研究对生命早期营养与成年期肥胖关联及其机制进行了探索,尽管存在诸多的局限性,研究结果提示了DNA甲基化可能介导生命早期营养状况对中国女性成年期肥胖的影响。
利益冲突 所有作者均声明不存在利益冲突
| [1] |
Yu ZB, Han SP, Zhu GZ, et al. Birth weight and subsequent risk of obesity:a systematic review and Meta-analysis[J]. Obes Rev, 2011, 12(7): 525-542. DOI:10.1111/j.1467-789X.2011.00867.x |
| [2] |
Araújo de França GV, Restrepo-Méndez MC, Loret de Mola C, et al. Size at birth and abdominal adiposity in adults:a systematic review and Meta-analysis[J]. Obes Rev, 2014, 15(2): 77-91. DOI:10.1111/obr.12109 |
| [3] |
Rønn PF, Smith LS, Andersen GS, et al. Birth weight and risk of adiposity among adult Inuit in Greenland[J]. PLoS One, 2014, 9(12): e115976. DOI:10.1371/journal.pone.0115976 |
| [4] |
Yuan ZP, Yang M, Liang L, et al. Possible role of birth weight on general and central obesity in Chinese children and adolescents:a cross-sectional study[J]. Ann Epidemiol, 2015, 25(10): 748-752. DOI:10.1016/j.annepidem.2015.05.011 |
| [5] |
Johnsson IW, Haglund B, Ahlsson F, et al. A high birth weight is associated with increased risk of type 2 diabetes and obesity[J]. Pediatr Obes, 2015, 10(2): 77-83. DOI:10.1111/ijpo.230 |
| [6] |
Robinson WR, Utz RL, Keyes KM, et al. Birth cohort effects on abdominal obesity in the United States:the Silent Generation, Baby Boomers and Generation X[J]. Int J Obes (Lond), 2013, 37(8): 1129-1134. DOI:10.1038/ijo.2012.198 |
| [7] |
Pan H, Lin XY, Wu YH, et al. HIF3A association with adiposity:the story begins before birth[J]. Epigenomics, 2015, 7(6): 937-950. DOI:10.2217/epi.15.45 |
| [8] |
Liu ZW, Zhang JT, Cai QY, et al. Birth weight is associated with placental fat mass-and obesity-associated gene expression and promoter methylation in a Chinese population[J]. J Matern Fetal Neonatal Med, 2016, 29(1): 106-111. DOI:10.3109/14767058.2014.987749 |
| [9] |
Yoo JY, Lee S, Lee HA, et al. Can proopiomelanocortin methylation be used as an early predictor of metabolic syndrome?[J]. Diabetes Care, 2014, 37(3): 734-739. DOI:10.2337/dc13-1012 |
| [10] |
Desquilbet L, Mariotti F. Dose-response analyses using restricted cubic spline functions in public health research[J]. Stat Med, 2010, 29(9): 1037-1057. DOI:10.1002/sim.3841 |
| [11] |
Newby PK, Dickman PW, Adami HO, et al. Early anthropometric measures and reproductive factors as predictors of body mass index and obesity among older women[J]. Int J Obes (Lond), 2005, 29(9): 1084-1092. DOI:10.1038/sj.ijo.0802996 |
| [12] |
Xia QH, Cai H, Xiang YB, et al. Prospective cohort studies of birth weight and risk of obesity, diabetes, and hypertension in adulthood among the Chinese population[J]. J Diabetes, 2019, 11(1): 55-64. DOI:10.1111/1753-0407.12800 |
| [13] |
Tobi EW, Heijmans BT, Kremer D, et al. DNA methylation of IGF2, GNASAS, INSIGF and LEP and being born small for gestational age[J]. Epigenetics, 2011, 6(2): 171-176. DOI:10.4161/epi.6.2.13516 |
| [14] |
Steegers-Theunissen RP, Obermann-Borst SA, Kremer D, et al. Periconceptional maternal folic acid use of 400μg per day is related to increased methylation of the IGF2 gene in the very young child[J]. PLoS One, 2009, 4(11): e7845. DOI:10.1371/journal.pone.0007845 |
| [15] |
Wang ZQ, Fung MR, Barlow DP, et al. Regulation of embryonic growth and lysosomal targeting by the imprinted Igf2/Mpr gene[J]. Nature, 1994, 372(6505): 464-467. DOI:10.1038/372464a0 |
| [16] |
Berkowicz EW, Magee DA, Berry DP, et al. Single nucleotide polymorphisms in the imprinted bovine insulin-like growth factor 2 receptor gene (IGF2R) are associated with body size traits in Irish Holstein-Friesian cattle[J]. Anim Genet, 2012, 43(1): 81-87. DOI:10.1111/j.1365-2052.2011.02211.x |
| [17] |
Young LE, Fernandes K, McEvoy TG, et al. Epigenetic change in IGF2R is associated with fetal overgrowth after sheep embryo culture[J]. Nat Genet, 2001, 27(2): 153-154. DOI:10.1038/84769 |
| [18] |
Ludwig T, Eggenschwiler J, Fisher P, et al. Mouse mutants lacking the type 2 IGF receptor (IGF2R) are rescued from perinatal lethality in Igf2 and Igf1r null backgrounds[J]. Dev Biol, 1996, 177(2): 517-535. DOI:10.1006/dbio.1996.0182 |
| [19] |
Kochmanski J, Marchlewicz EH, Savidge M, et al. Longitudinal effects of developmental bisphenol A and variable diet exposures on epigenetic drift in mice[J]. Reprod Toxicol, 2017, 68: 154-163. DOI:10.1016/j.reprotox.2016.07.021 |
| [20] |
Talens RP, Christensen K, Putter H, et al. Epigenetic variation during the adult lifespan:cross-sectional and longitudinal data on monozygotic twin pairs[J]. Aging Cell, 2012, 11(4): 694-703. DOI:10.1111/j.1474-9726.2012.00835.x |
| [21] |
van Dijk SJ, Molloy PL, Varinli H, et al. Epigenetics and human obesity[J]. Int J Obes (Lond), 2015, 39(1): 85-97. DOI:10.1038/ijo.2014.34 |
| [22] |
Dick KJ, Nelson CP, Tsaprouni L, et al. DNA methylation and body-mass index:a genome-wide analysis[J]. Lancet, 2014, 383(9933): 1990-1998. DOI:10.1016/S0140-6736(13)62674-4 |
| [23] |
Talens RP, Boomsma DI, Tobi EW, et al. Variation, patterns, and temporal stability of DNA methylation:considerations for epigenetic epidemiology[J]. FASEB J, 2010, 24(9): 3135-3144. DOI:10.1096/fj.09-150490 |
| [24] |
Crujeiras AB, Diaz-Lagares A, Sandoval J, et al. DNA methylation map in circulating leukocytes mirrors subcutaneous adipose tissue methylation pattern:a genome-wide analysis from non-obese and obese patients[J]. Sci Rep, 2017, 7: 41903. DOI:10.1038/srep41903 |
| [25] |
Huang YT, Chu S, Loucks EB, et al. Epigenome-wide profiling of DNA methylation in paired samples of adipose tissue and blood[J]. Epigenetics, 2016, 11(3): 227-236. DOI:10.1080/15592294.2016.1146853 |
| [26] |
Slieker RC, Bos SD, Goeman JJ, et al. Identification and systematic annotation of tissue-specific differentially methylated regions using the Illumina 450k array[J]. Epigenetics Chromatin, 2013, 6(1): 26. DOI:10.1186/1756-8935-6-26 |
| [27] |
Tobi EW, Slieker RC, Luijk R, et al. DNA methylation as a mediator of the association between prenatal adversity and risk factors for metabolic disease in adulthood[J]. Sci Adv, 2018, 4(1): eaao4364. DOI:10.1126/sciadv.aao4364 |
| [28] |
Wahl S, Drong A, Lehne B, et al. Epigenome-wide association study of body mass index, and the adverse outcomes of adiposity[J]. Nature, 2017, 541(7635): 81-86. DOI:10.1038/nature20784 |
 2019, Vol. 40
2019, Vol. 40