文章信息
- 梁文娟, 崔聪聪, 段广才, 刘慧莹, 徐亚珂, 郗园林, 杨海燕, 陈帅印.
- Shigella Liang Wenjuan, Cui Congcong, Duan Guangcai, Liu Huiying, Xu Yake, Xi Yuanlin, Yang Haiyan, Chen Shuaiyin.
- CRISPR1上游侧翼序列用于大肠埃希菌和志贺菌鉴定的效果评价
- Identification and evaluation on methods with upstream flank sequences of CRISPR1, regarding Escherichia coli and Shigella
- 中华流行病学杂志, 2018, 39(12): 1607-1610
- Chinese Journal of Epidemiology, 2018, 39(12): 1607-1610
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2018.12.013
-
文章历史
收稿日期: 2018-06-14
2. 450001 郑州大学公共卫生学院流行病与卫生统计学系;
3. 453003 新乡医学院分子诊断与医学检验技术河南省协同创新中心
2. Department of Epidemiology, College of Public Health, Zhengzhou University, Zhengzhou 450001, China;
3. Henan Innovation Center of Molecular Diagnosis and Laboratory Medicine, Xinxiang Medical University, Xinxiang 453003, China
致病大肠埃希菌和志贺菌感染给公众健康带来严重危害[1-2]。快速鉴定大肠埃希菌和志贺菌具有重要意义。成簇规律间隔的短回文重复序列(clustered regularly interspaced short palindromic repeats,CRISPR)由一段不连续的重复序列和高度可变的间隔序列组成,和CRISPR相关基因(CRISPR-associated,cas)及前导序列共同组成CRISPR/Cas系统,存在于大多数古细菌(84%)和近一半(46%)的细菌基因组中[3]。在大肠埃希菌中存在I-E型(由CRISPR1及相邻的cas和CRISPR2共同组成)或I-F型CRISPR/Cas系统(CRISPR3及下游的cas和CRISPR4位点)[4]。已有研究显示,志贺菌仅存在I-E型CRISPR/Cas[5]。CRISPR上游侧翼序列包含转录启动子,在获得新间隔序列的过程中发挥重要作用[6]。本研究选取经过全基因组测序的大肠埃希菌203株和志贺菌9株,分析其CRISPR/Cas与种系关系;建立CRISPR1上游侧翼序列对大肠埃希菌和志贺菌鉴定方法,然后在266株实验室保存的不同种类的细菌中进行PCR验证并评价其效果。
材料与方法1.菌株:选取经过全基因组测序的大肠埃希菌(203株)、志贺菌(9株)和实验室保存的266株不同种属的细菌,其中大肠埃希菌(130株),志贺菌(59株),沙门菌(38株),其他种属菌株1~7个不等,实验室菌株均经过细菌鉴定国际金标准(API)鉴定。
2. CRISPR/Cas类型识别:具体方法参照文献[7]。
3.大肠埃希菌种系分析:部分全基因组测序的大肠埃希菌的种系通过NCBI数据库搜索相关文献获得,其他没有明确种系的菌株通过BLAST获取鉴定种系的chuA、yjaA和TSPE4.C2基因判定。实验室大肠埃希菌种系使用两步三重PCR的方法,判定结果标准均参照文献[8]。
4. CRISPRs侧翼序列比对:选取大肠埃希菌和志贺菌CRISPR位点上、下游各500 bp序列,使用Clustal X进行侧翼序列的比对,获得不同CRISPRs侧翼序列的特征。
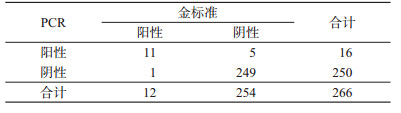
5.大肠埃希菌和志贺菌鉴定引物序列及PCR鉴定和评价:本研究设计引物扩增鉴定大肠埃希菌和志贺菌特异序列,见表 1。为评价其效果,与金标准(API和种系分类法)相比,计算其灵敏度,特异度,符合率和Kappa值。
PCR扩增体系(25 μl):Taq Mix 12.5 μl,引物各1 μl,模板DNA 2 μl,ddH2O 8.5 μl;PCR反应条件:94 ℃预变性5 min;94 ℃变性30 s,退火30 s,72 ℃延伸45 s,共32个循环;72 ℃延伸10 min。PCR产物用2%的琼脂糖凝胶电泳30 min,凝胶成像仪观察结果,拍照保存。
结果1.大肠埃希菌CRISPR/Cas的分布及与种系关系:CRISPR/Cas的分布显示73.4%(149/203)的大肠埃希菌存在I-E型CRISPR/Cas,包含了A、B1、D种系的大肠埃希菌。含有I-F型CRISPR/Cas和仅有CRISPR3-4(无CRISPR/Cas)的大肠埃希菌均属于B2种系。见表 2。9株志贺菌均存在I-E型CRISPR/Cas。
2. CRISPRs的侧翼序列比对:研究分别选取确定CRISPRs的上、下游500 bp的侧翼序列进行比对,显示大肠埃希菌的CRISPR2上游侧翼序列和下游侧翼序列、CRISPR3的上游侧翼序列和CRISPR4的下游侧翼序列一致性均超过98%,经BLAST分析,不具有种属特异性。
在大肠埃希菌(B2种系外)和志贺菌CRISPR1的上游61 bp侧翼序列存在99%的一致性,且经BLAST分析,具有很高的种属特异性,提示其可以成为大肠埃希菌(B2种系外)和志贺菌的分子鉴定靶标。
大肠埃希菌B2种系虽然没有CRISPR1位点,但是在其等位基因区域上游存在61 bp侧翼序列显示99%的一致性,且经BLAST分析,具有很强的种属特异性,提示可以成为大肠埃希菌(B2种系)分子鉴定靶标。
3. CRISPR1上游侧翼序列用于大肠埃希菌和志贺菌鉴定和评价:本研究验证纳入266株不同种属的细菌。
(1)大肠埃希菌种系:在130株大肠埃希菌中,A群共61株(46.9%);其次B1群共27株(20.8%);D群共30株(23.1%);B2群共12株(9.2%)。
(2)评价大肠埃希菌(B2种系除外)和志贺菌鉴定的效果:PCR检测大肠埃希菌(B2种系除外)和志贺菌的侧翼序列,见图 1。为评价CRISPR1上游特异序列对大肠埃希菌(B2种系除外)和志贺菌的鉴定效果,共选用266份不同种属的菌株。与金标准API和种系鉴定结果相比,PCR扩增CRISPR1上游特异序列鉴定大肠埃希菌(B2种系除外)和志贺菌的灵敏度为97.2%,特异度为96.6%,符合率为97.0%,Kappa值为0.93(表 3)。
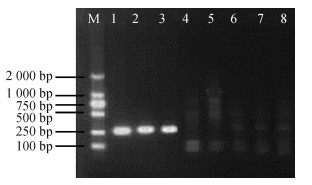
|
| 注:M:DL2000 Marker;1:大肠埃希菌2014001;2:大肠埃希菌2002002;3:志贺菌2014030;4:沙门菌2014001;5:肺炎克雷伯杆菌2015001;6:铜绿假单胞菌2014002;7:柠檬酸杆菌2015001;8:金黄色葡萄球菌2014001 图 1 大肠埃希菌和志贺菌特异序列的电泳图 |
(3)评价大肠埃希菌B2种系菌株鉴定效果:PCR检测大肠埃希菌B2种系菌株的CRISPR1等位基因区域,见图 2。为评价CRISPR1等位基因区域上游特异序列鉴定大肠埃希菌(B2种系)的效果,共选用266份菌株。PCR扩增结果显示,与金标准API和种系鉴定结果相比,CRISPR1上游特异序列鉴定大肠埃希菌(B2种系)灵敏度为91.7%,特异度为98.0%,符合率为97.8%,Kappa值为0.77(表 4)。
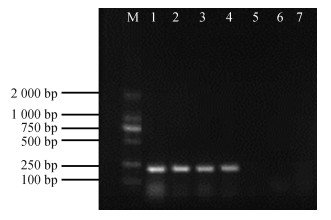
|
| 注:M:DL2000 marker;1:大肠埃希菌2014009;2:大肠埃希菌2014010;3:大肠埃希菌2014041;4:大肠埃希菌2014099;5:沙门菌2014001;6:志贺菌2014030;7:铜绿假单胞菌2014002 图 2 大肠埃希菌(B2种系)特异序列电泳图 |
大肠埃希菌和志贺菌均属于革兰阴性杆菌,由少数致病大肠埃希菌和志贺菌感染所引起的感染性腹泻是全球范围内重要的传染病之一,严重者可导致死亡,给公众健康带来了严重危害。
本研究显示,73.4%(149/203)全基因组测序大肠埃希菌携带I-E型CRISPR/Cas,Touchon等[9]研究263株不同来源的大肠埃希菌,结果显示,163株(62.0%)拥有完整的I-E型CRISPR系统。本研究结果显示,8.4%的大肠埃希菌存在I-F型CRISPR/Cas,17.2%大肠埃希菌既不存在I-E型CRISPR/Cas的CRISPR1和CRISPR2,也不存在I-F型CRISPR/Cas的CRISPR3和CRISPR4,仅在I-F型CRISPR/Cas的位置留有一段序列CRISPR3-4,此类菌株暂称之为无CRISPR/Cas菌株。Touchon和Rocha[4]及García- Gutiérrez等[10]研究显示I-E型CRISPR/Cas或者I-F型CRISPR/Cas只能单独存在大肠埃希菌中,本研究发现在大多数大肠埃希菌确实如此,但也发现2株大肠埃希菌B7A和Co6114具有完整的I-E和I-F系统,这一结果丰富了大肠埃希菌CRISPR/Cas的结构类型。因I-E型CRISPR/Cas在大肠埃希菌中具有数量优势,所以暂时将B7A和Co6114划分为I-E型CRISPR/Cas的细菌。研究显示9株志贺菌均含有I-E型CRISPR/Cas。
种系进化分析结果显示,存在I-E型CRISPR/Cas的大肠埃希菌包含了种系进化的大多数型别(A、B1和D系),而含有I-F型CRISPR/Cas和无CRISPR/Cas的大肠埃希菌均属于B2系。Touchon等[9]的研究结果也显示,无CRISPR/Cas的大肠埃希菌是B2系,这也许暗示CRSIPR进化过程中的选择性。进一步分析显示,I-F型CRISPR/Cas和无CRISPR/Cas的大肠埃希菌大多数来源于人类尿液、血液和禽,属于人类肠外致病菌和禽致病菌。大多数肠道外致病性大肠埃希菌的B2群处于该物种遗传进化最古老的种系[11],但同是B2系菌株,一部分菌株存在I-F型CRISPR/Cas,而另一部分菌株无CRISPR/Cas,其差异有待于进一步研究。
本研究结果显示,大肠埃希菌CRISPR2上游侧翼序列和下游侧翼序列、CRISPR3的上游侧翼序列和CRISPR4的下游侧翼序列一致性均超过98%,与王鹏飞[12]的研究结果一致,体现细菌在进化过程中的保守型。但是本研究分析CRISPR1的上游序列时,在大肠埃希菌(B2种系外)和志贺菌的CRISPR1的上游、大肠埃希菌(B2种系)与CRISPR1等位基因区域上游存在均有61 bp侧翼序列分别有几乎99%的一致性,经BLAST分析,其具有种属特异性。该序列具有大肠埃希菌的种属特异性,推测其与鉴定大肠埃希菌种系的chuA、yjaA和TSPE4.C2基因一样,是大肠埃希菌进化过程保守性的选择。根据此区域设计引物,PCR扩增,显示鉴定的灵敏性和特异性均>91%,与API菌株鉴定和种系分类结果比较,具有很高的一致性。研究也显示沙门菌的CRISPR1的上游61 bp的特异序列也可称为其分子鉴定靶标(待发表)。由此提示在细菌的非编码区序列有许多潜在的功能,有待开发。本研究显示大肠埃希菌的种系在基因水平并不属于真正的同一自然族群,与B2系大肠埃希菌相比,志贺菌与具有I-E型CRISPR/Cas的大肠埃希菌或许具有更近的遗传学关系。梁文娟[13]及龙金照等[14]的研究结果显示,产志贺毒素大肠埃希菌也均存在I-E型CRISPR/Cas,且不存在I-F型CRISPR/Cas,与志贺菌类似。这也许显示产志贺毒素的前噬菌体的插入细菌有一定的倾向性,这种特征有待进一步的研究。
研究显示大肠埃希菌CRISPRs的间隔序列可以作为其血清型的分子靶标[15],本研究为大肠埃希菌和志贺菌的鉴定提供了一种简单、容易操作的手段,和CRISPRs的间隔序列可共同组成一套鉴定大肠埃希菌及其血清型的系统,可广泛应用于农产品企业、水源、肉制品等大肠埃希菌和志贺菌的快速检测,预计将在农业、检验检疫、食品工业及卫生防疫部门具有较广的应用前景。
利益冲突 无
| [1] |
Cooper KK, Mandrell RE, Louie JW, et al. Comparative genomics of enterohemorrhagic Escherichia coli O145:H28 demonstrates a common evolutionary lineage with Escherichia coli O157:H7[J]. BMC Genomics, 2014, 15: 17. DOI:10.1186/1471-2164-15-17 |
| [2] |
Wang Y, Song C, Duan G, et al. Transposition of ISEcp1 modulates blaCTX-M-55-mediated Shigella flexneri resistance to cefalothin[J]. Int J Antimicrob Agents, 2013, 42(6): 507-512. DOI:10.1016/j.ijantimicag.2013.08.009 |
| [3] |
Grissa I, Vergnaud G, Pourcel C. CRISPRFinder:a web tool to identify clustered regularly interspaced short palindromic repeats[J]. Nucleic Acids Res, 2007, 35(Web Server issue): W52-57. |
| [4] |
Touchon M, Rocha EP. The small, slow and specialized CRISPR and anti-CRISPR of Escherichia and Salmonella[J]. PLoS One, 2010, 5(6): e11126. DOI:10.1371/journal.pone.0011126 |
| [5] |
Guo X, Wang Y, Duan G, et al. Detection and analysis of CRISPRs of Shigella[J]. Curr Microbiol, 2015, 70(1): 85-90. DOI:10.1007/s00284-014-0683-8 |
| [6] |
Kiro R, Goren MG, Yosef I, et al. CRISPR adaptation in Escherichia coli subtypeI-E system[J]. Biochem Soc Trans, 2013, 41(6): 1412-1415. DOI:10.1042/BST20130109 |
| [7] |
梁文娟, 张荣光, 段广才, 等. 基于CRISPR/Cas的大肠埃希菌分子标志物的监测研究[J]. 中华流行病学杂志, 2016, 37(8): 1080-1086. Liang WJ, Zhang RG, Duan GC, et al. A surveillance study on CRISPR/Cas molecular biomarker in Escherichia coli[J]. Chin J Epidemiol, 2016, 37(8): 1080-1086. DOI:10.3760/cma.j.issn.0254-6450.2016.08.005 |
| [8] |
Clermont O, Bonacorsi S, Bingen E. Rapid and simple determination of the Escherichia coli phylogenetic group[J]. Appl Environ Microbiol, 2000, 66(10): 4555-4558. DOI:10.1128/AEM.66.10.4555-4558.2000 |
| [9] |
Touchon M, Charpentier S, Pognard D, et al. Antibiotic resistance plasmids spread among natural isolates of Escherichia coli in spite of CRISPR elements[J]. Microbiology, 2012, 158(12): 2997-3004. DOI:10.1099/mic.0.060814-0 |
| [10] |
García-Gutiérrez E, Almendros C, Mojica FJ, et al. CRISPR Content Correlates with the Pathogenic Potential of Escherichia coli[J]. PLoS One, 2015, 10(7): e131935. |
| [11] |
Tenaillon O, Skurnik D, Picard B, et al. The population genetics of commensal Escherichia coli[J]. Nat Rev Microbiol, 2010, 8(3): 207-217. DOI:10.1038/nrmicro2298 |
| [12] |
王鹏飞.志贺菌、大肠杆菌和沙门菌CRISPR结构特征生物信息学分析[D].郑州大学, 2015. Wang PF. Bioinformatics analysis of CRISPR structures and feature in Shigella, Escherichia coli and Salmonella[D]. Zhengzhou: Zhengzhou University, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10459-1015302504.htm |
| [13] |
梁文娟.基于CRISPRs的大肠埃希菌分型方法及其与耐药和毒力关系[D].郑州大学, 2017. Liang WJ. The genotyping method based on CRISPRs and the relationship between the CRISPR/Cas and the virulence and resistant in Escherichia coli[D]. Zhengzhou: Zhengzhou University, 2017. http://cdmd.cnki.com.cn/Article/CDMD-10459-1018058648.htm |
| [14] |
龙金照, 徐亚珂, 段广才, 等. O26:H11及NM大肠埃希菌CRISPR的分子分布特征及其与stx噬菌体的关系[J]. 中华流行病学杂志, 2017, 38(7): 944-949. Long JZ, Xu YK, Duan GC, et al. Association between phage-mediated Shiga toxin and molecular distribution of CRISPR in Escherichia coli O26:H11 or NM[J]. Chin J Epidemiol, 2017, 38(7): 944-949. DOI:10.3760/cma.j.issn.0254-6450.2017.07.019 |
| [15] |
Delannoy S, Beutin L, Fach P. Use of clustered regularly interspaced short palindromic repeat sequence polymorphisms for specific detection of enterohemorrhagic Escherichia coli strains of serotypes O26:H11, O45:H2, O103:H2, O111:H8, O121:H19, O145:H28, and O157:H7 by real-time PCR[J]. J Clin Microbiol, 2012, 50(12): 4035-4040. DOI:10.1128/JCM.02097-12 |
 2018, Vol. 39
2018, Vol. 39