文章信息
- 张慧, 刘宇鹏, 葛安琪, 王玄, 孙鸿儒, 毕皓然, 庞达, 赵亚双.
- Zhang Hui, Liu Yupeng, Ge Anqi, Wang Xuan, Sun Hongru, Bi Haoran, Pang Da, Zhao Yashuang.
- 外周血白细胞AOX1和IRF4基因甲基化与乳腺癌关系的病例对照研究
- Association between AOX1, IRF4 methylation in peripheral blood leukocyte DNA and the risks of breast cancer: a case-control study
- 中华流行病学杂志, 2018, 39(9): 1265-1269
- Chinese Journal of Epidemiology, 2018, 39(9): 1265-1269
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2018.09.023
-
文章历史
收稿日期: 2018-03-29
2. 150081 哈尔滨医科大学附属肿瘤医院乳腺外科
2. Department of Breast Surgery, Cancer Hospital, Harbin Medical University, Harbin 150081, China
乳腺癌是最常见的女性恶性肿瘤之一[1]。研究显示,表观遗传学的改变尤其是DNA启动子区甲基化是肿瘤发展的一个重要原因[2-4]。Øster等[5]在结直肠癌的研究中发现醛氧化酶1(aldehyde oxidase 1,AOX1)在癌组织中呈现高甲基化水平,在一项乳腺癌患者组织全基因组甲基化图谱检测研究中发现一组与细胞增殖、分化和转录相关的候选基因包括干扰素调节因子4(interferon regulatory factor 4,IRF4)的DNA甲基化水平高于正常组织[6]。目前关于DNA甲基化与癌症发病关系的研究主要集中于组织来源的生物样本,本研究基于外周血白细胞中AOX1、IRF4甲基化状态及其与相关环境因素的交互作用分析其与乳腺癌的关系。
对象与方法1.研究对象:采用病例对照的研究方法探讨外周血中AOX1和IRF4基因甲基化与乳腺癌的关系。患者为2010-2014年在哈尔滨医科大学附属肿瘤医院首次就诊,经病理诊断确诊为乳腺癌且无其他癌症史的女性患者401例,年龄(51.73 ± 9.39)岁。同期共纳入555例女性对照,年龄(51.89±10.31)岁,其中哈尔滨医科大学附属第二医院骨科及眼科就诊患者258例,年龄(53.57±9.84)岁;哈尔滨市香坊社区进行健康体检的社区居民297例,年龄(50.43±10.51)岁,对照人群无乳腺癌病史及其他癌症病史。排除无法完成问卷调查及无法采集外周静脉血的研究对象。采集病例及对照组外周静脉血5 ml。住院患者的血液在手术前采集,并立即分装、储存于-80 ℃冰箱备用。
2.问卷调查:主要内容为过去1年的信息,包括基本资料(年龄、身高、体重、BMI、民族、文化程度、癌症家族史等)、饮食习惯(蔬菜、水果、肉类、奶制品等食用量)、行为习惯(吸烟、饮酒、体力活动、职业类型等)以及月经史、生育史、服用避孕药史及激素使用情况等。
3.检测方法:
(1)DNA提取及重亚硫酸盐修饰:使用德国Qiagen公司QIAamp DNA Blood Mini Kit,按照说明书对血样进行DNA提取。使用Nanodrop 2000 Spectrophotometer(美国Thermo Scientific公司)检测DNA浓度和质量。使用EpiTect重亚硫酸盐修饰试剂盒(德国Qiagen公司),按照说明书将提取到的DNA进行重亚硫酸盐修饰处理。
(2)标准品制备:完全甲基化及完全非甲基化DNA(美国ZYMO公司)进行重亚硫酸盐修饰转换,二者按照比例进行梯度稀释,制备100%、2%、1%、0.5%和0%甲基化水平的标准品模板(100%表示完全甲基化,0%表示完全非甲基化),在后续高分辨率熔解曲线法(methylation sensitive-high resolution melting curve analysis,MS-HRM)检测样本甲基化水平时用于半定量评估分析。
(3)PCR和MS-HRM检测:使用Light Cycler 480(Roche Applied Science,Mannheim,Germany)进行PCR扩增及MS-HRM基因甲基化水平检测。AOX1基因扩增引物:5′-GAA CGT TGG ATT TTA ATT AAG GTT TTT-3′,5′-CTA AAA AAT AAC GAA CAC CTA AAA CC-3′,扩增片段位于2号染色体:201 450 661~201 450 785,包括10个CG位点;IRF4基因扩增引物:5′-CGT TGT AGT TTA GTG ATT GAT TG-3′,5′-GCT TCG AAA ACT ATC ACT AAA AC-3′,扩增片段位于6号染色体:391 379~391 489,包括10个CG位点。PCR反应体系:Mix试剂5 μl、引物各0.25 μl、Mg2+溶液1.20 μl、重亚硫酸盐修饰后的DNA 0.60 μl,补充双蒸水至10 μl。DNA样本检测采用复孔,保证结果的可重复性,并以双蒸水作空白对照。AOX1基因HRM检测:梯度退火64~56 ℃ 15 s,共55个循环,熔解温度67~93 ℃;IRF4基因HRM检测:梯度退火63~54 ℃ 30 s,共50个循环,熔解温度64~88 ℃。将样本的熔解曲线与甲基化标准模板进行比较确定甲基化水平。
4.统计学分析:采用χ2检验分析分类变量在病例组和对照组中的差异。采用单因素和多因素非条件logistic回归计算AOX1和IRF4基因甲基化与乳腺癌发病的关系。相乘交互作用采用非条件logistic回归分析。相加交互作用采用Andersson等[7]编制的Excel软件分析,采用交互作用超额相对危险度(RERI)、交互作用归因比(AP),效应指数(SI)3个指标评价交互作用,若RERI和AP的95%CI包含0,SI的95%CI包含1,提示两因素间无相加交互作用。使用SPSS 24.0软件进行数据分析。
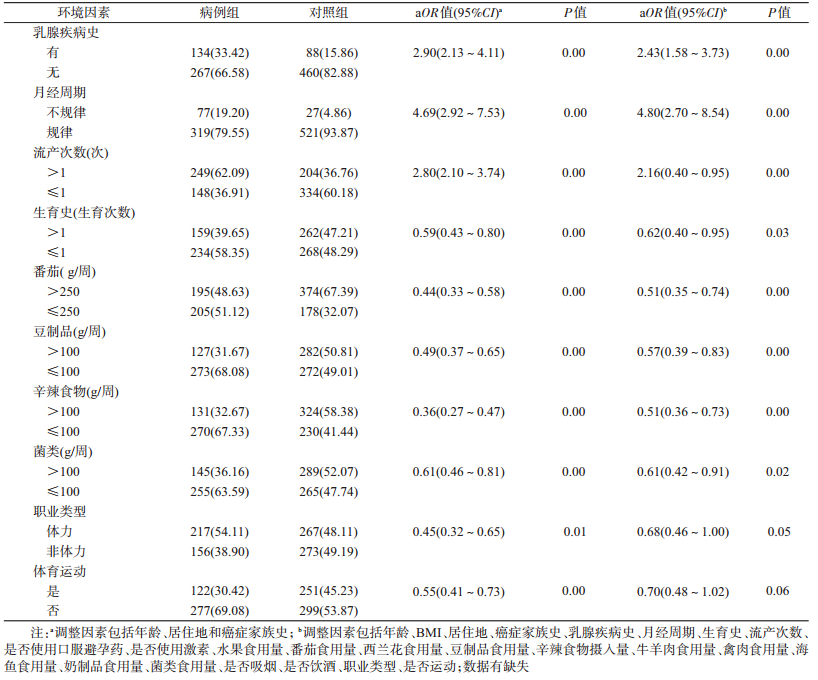
结果1.一般情况:病例组和对照组的年龄、居住地和癌症家族史之间差异有统计学意义(P < 0.05),后续统计学分析将其纳入作为调整因素。见表 1。
2.基因甲基化与乳腺癌的关系:调整年龄、居住地、癌症家族史,非条件logistic回归结果显示,AOX1非甲基化是乳腺癌发病的危险因素。IRF4非甲基化是乳腺癌发生的危险因素。见表 2。
3.基因甲基化和环境因素的交互作用与乳腺癌的关系:多因素logistic回归分析显示,乳腺疾病史、月经周期、流产次数、生育次数、食用番茄、豆制品、辛辣食物、菌类、职业类型和体育运动10个因素与乳腺癌的发病有关。见表 3。进一步分析AOX1、IRF4基因甲基化与上述环境因素之间的相加和相乘交互作用与乳腺癌的关系,结果显示,AOX1基因非甲基化与菌类食用量少(< 100 g/周)和缺乏运动存在相乘交互作用,即不携带AOX1甲基化时,每周食用菌类 < 100 g的个体发生乳腺癌的风险增加(相乘交互作用);不携带AOX1甲基化时,缺乏体育运动的个体发生乳腺癌的风险增加(相乘交互作用)。IRF4基因甲基化与上述环境因素不存在相乘和相加交互作用。见表 4,5。
癌症的特征是不受控的生长以及细胞机制的紊乱,具有基因突变和染色体异常等特征。随着表观遗传学尤其是DNA甲基化研究的深入,为癌症复杂机制的解释提供了新的思路。Hansen等[8]在大肠癌、肺癌和乳腺癌组织中均检测到DNA异常甲基化,并且结果显示由于DNA甲基化异常引起表观遗传的不稳定性,促进了肿瘤的异质性。
近年,有研究者将AOX1与C1orf114(Chromosome 1 open reading frame 114)、GSTP1(Glutathione s-transferase pi 1)等基因在肿瘤组织中甲基化情况进行联合研究,用于预测大肠癌、前列腺癌等疾病复发风险及预后情况[9-10];Yagi等[11]在大肠癌甲基化基因的研究中发现AOX1表达下调。Wojdacz等[12]在外周血白细胞中检测到BRCA(Breast cancer susceptibility gene)、APC(Adenomatous polyposis coli)等乳腺癌常见基因启动子甲基化的存在;Xu等[13]的前瞻性队列研究采用Infinium Human Methylation27 Bead Chips检测全血样本,结果发现250个差异甲基化位点,并且75.20%的乳腺癌患者发生低甲基化,该研究结果提示血液中基因甲基化图谱具有潜在诊断及预测乳腺癌风险的作用。在本研究中,病例组和对照组外周血样本均检测到AOX1基因的低甲基化,调整年龄、居住地和癌症家族史后OR值为1.37,即不携带AOX1基因甲基化能够增加乳腺癌发病风险。
世界癌症报告结果显示生活方式、体力活动、饮食习惯等均与癌症相关[14]。我们分析环境因素与外周血白细胞中DNA甲基化交互作用与乳腺癌发生的关系,结果显示,AOX1非甲基化与菌类食用量少(< 100 g/周)以及缺乏运动之间存在相乘交互作用,提示不携带AOX1甲基化时,菌类食用量少及缺乏运动,乳腺癌的发病风险增加。一项病例对照研究发现食用≥10 g/d的新鲜蘑菇或≥4 g/d的干蘑菇能够降低乳腺癌发病风险[15]。国内有研究显示菌类多糖具有诱导肿瘤细胞凋亡的作用,其中多糖类物质或通过增强机体免疫功能发挥抗肿瘤作用。最新发表的一项随访研究发现每周运动超过6.36 h的乳腺癌女性患者癌组织中APC、HIN1(Cytokine high in normal-1)启动子发生甲基化与APC、HIN1没有发生甲基化的患者相比死亡风险更低,获得了与原假设相反的结果,可能是由于运动降低了乳腺癌发展过程中抑癌基因失活所产生的负面效应[16]。
本研究局限性。环境因素收集为回顾性内容,可能存在一定的回忆偏倚。本研究中AOX1非甲基化为乳腺癌的危险因素,与以往癌组织中甲基化的研究结果不一致[5, 10],然而在肿瘤组织生长之前基因组DNA就可能已经发生改变,另外,甲基化具有组织特异性,因此肿瘤组织甲基化检测结果可能并不能够代表表观遗传信息的改变;除此之外,AOX1基因对外周血白细胞功能的影响及其对肿瘤发病机制的影响还不清楚,需要进一步的研究。IRF4基因甲基化与乳腺癌发病关系研究结果具有临界统计学意义,需要更大的样本量进行后续分析。
利益冲突: 无
| [1] |
Siegel RL, Miller KD, Jemal A. Cancer statistics, 2017[J]. CA Cancer J Clin, 2017, 67(1): 7-30. DOI:10.3322/caac.21387 |
| [2] |
Fazzari MJ, Greally JM. Epigenomics:beyond CpG islands[J]. Nat Rev Genet, 2004, 5(6): 446-455. DOI:10.1038/nrg1349 |
| [3] |
Adorján P, Distler J, Lipscher E, et al. Tumour class prediction and discovery by microarray-based DNA methylation analysis[J]. Nucleic Acids Res, 2002, 30(5): e21. DOI:10.1093/nar/30.5.e21 |
| [4] |
Cho YH, Yazici H, Wu HC, et al. Aberrant promoter hypermethylation and genomic hypomethylation in tumor, adjacent normal tissues and blood from breast cancer patients[J]. Anticancer Res, 2010, 30(7): 2489-2496. |
| [5] |
Øster B, Thorsen K, Lamy P, et al. Identification and validation of highly frequent CpG island hypermethylation in colorectal adenomas and carcinomas[J]. Int J Cancer, 2011, 129(12): 2855-2866. DOI:10.1002/ijc.25951 |
| [6] |
Lindqvist BM, Wingren S, Motlagh PB, et al. Whole genome DNA methylation signature of HER2-positive breast cancer[J]. Epigenetics, 2014, 9(8): 1149-1162. DOI:10.4161/epi.29632 |
| [7] |
Andersson T, Alfredsson L, Källberg H, et al. Calculating measures of biological interaction[J]. Eur J Epidemiol, 2005, 20(7): 575-579. DOI:10.1007/s10654-005-7835-x |
| [8] |
Hansen KD, Timp W, Bravo HC, et al. Increased methylation variation in epigenetic domains across cancer types[J]. Nat Genet, 2011, 43(8): 768-775. DOI:10.1038/ng.865 |
| [9] |
Varisli L. Identification of new genes downregulated in prostate cancer and investigation of their effects on prognosis[J]. Genet Test Mol Biomarkers, 2013, 17(7): 562-566. DOI:10.1089/gtmb.2012.0524 |
| [10] |
Haldrup C, Mundbjerg K, Vestergaard EM, et al. DNA methylation signatures for prediction of biochemical recurrence after radical prostatectomy of clinically localized prostate cancer[J]. J Clin Oncol, 2013, 31(26): 3250-3258. DOI:10.1200/JCO.2012.47.1847 |
| [11] |
Yagi K, Akagi K, Hayashi H, et al. Three DNA methylation epigenotypes in human colorectal cancer[J]. Clin Cancer Res, 2010, 16(1): 21-33. DOI:10.1158/1078-0432.ccr-09-2006 |
| [12] |
Wojdacz TK, Thestrup BB, Cold S, et al. No difference in the frequency of locus-specific methylation in the peripheral blood DNA of women diagnosed with breast cancer and age-matched controls[J]. Future Oncol, 2011, 7(12): 1451-1455. DOI:10.2217/fon.11.123 |
| [13] |
Xu ZL, Bolick SCE, DeRoo LA, et al. Epigenome-wide association study of breast cancer using prospectively collected sister study samples[J]. J Natl Cancer Inst, 2013, 105(10): 694-700. DOI:10.1093/jnci/djt045 |
| [14] |
McGuire S. World cancer report 2014. Geneva, Switzerland:world health organization, international agency for research on cancer, WHO Press, 2015[J]. Adv Nutr, 2016, 7(2): 418-419. DOI:10.3945/an.116.012211 |
| [15] |
Zhang M, Huang J, Xie X, et al. Dietary intakes of mushrooms and green tea combine to reduce the risk of breast cancer in Chinese women[J]. Int J Cancer, 2009, 124(6): 1404-1408. DOI:10.1002/ijc.24047 |
| [16] |
McCullough LE, Chen J, Cho YH, et al. Modification of the association between recreational physical activity and survival after breast cancer by promoter methylation in breast cancer-related genes[J]. Breast Cancer Res, 2017, 19: 19. DOI:10.1186/s13058-017-0811-z |
 2018, Vol. 39
2018, Vol. 39