文章信息
- 石丽媛, 丁奕博, 谭红丽, 郭英, 张海鹏, 段存娟, 李伟, 王鹏.
- Shi Liyuan, Ding Yibo, Tan Hongli, Guo Ying, Zhang Haipeng, Duan Cunjuan, Li Wei, Wang Peng.
- 云南省鹤庆县2017年分离鼠疫菌分子溯源
- Source tracing of the Yersinia pestis strains isolated from Heqing county, Yunnan province in 2017
- 中华流行病学杂志, 2018, 39(7): 983-987
- Chinese Journal of Epidemiology, 2018, 39(7): 983-987
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2018.07.022
-
文章历史
收稿日期: 2018-01-31
2. 102206 北京, 中国疾病预防控制中心传染病预防控制所 传染病预防控制国家重点实验室
2. State Key Laboratory for Communicable Diseases Control and Prevention, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
据史料记载[1],1772-1937年鹤庆县流行鼠疫50年次,累计死亡15 000人,此后再无疫情报道。新中国成立以后,鹤庆县于1985年在马厂村、新蜂村的动物血清中发现鼠疫抗体放射免疫阳性,1990年开始每月进行鼠疫监测。2017年4-5月云南省鹤庆县从350多份活鼠及其寄生蚤中分离到10株鼠疫菌,本研究对这10株鼠疫菌进行分子分型。鼠疫分子分型常用方法包括差异片段(Different region,DFR)、规律成簇的间隔短回文重复序列(Clustered regularly interspaced short palindromic repeats,CRISPRs)及多位点可变数目串联重复序列分析(Multiple-locus variable-number tandem repeat analysis,MLVA)[2]。本团队前期已建立了云南省各鼠疫疫源地200余株鼠疫菌的DFR-CRISPRs-MLVA数据库[3-5],本研究采用上述3种分型方法对鹤庆地区分离菌株进行分型,并与数据库菌株进行比较和聚类分析。
材料与方法1.菌株及其DNA制备:2017年分离自鹤庆县草海镇马厂村的鼠疫菌10株,其中分离自大绒鼠6株、齐氏姬鼠1株、蚤3株。EV76作为PCR反应的阳性对照。菌株DNA提取制备采用德国Qiagen公司DNeasy Blood & Tissue Kit进行。
2. DFR分型:包括23对DFR(DFR01~23)分型引物和质粒验证引物(PMT1),引物序列参照文献[6]设计。扩增条件:95 ℃ 3 min;94 ℃ 30 s,60 ℃(DFR18、DFR19用56 ℃)30 s,72 ℃ 1 min,30个循环;72 ℃ 5 min。琼脂糖凝胶电泳检测PCR产物。
3. CRISPRs分型:共有YPa、YPb及YPc 3对引物,引物序列参照文献[7]设计。扩增条件:95 ℃ 5 min;95 ℃ 40 s,58 ℃ 40 s,72 ℃ 40 s,30个循环;72 ℃ 5 min。对产物进行测序,利用在线工具(http://crispr.i2bc.paris-saclay.fr/)查找spacers[8],再对应spacers库[7]识别出spacers类型。
4. MLVA分型:采用MLVA“14+12”分型策略的26个VNTR位点,引物序列参照文献设计[9],每对引物的上游用荧光标记。扩增条件:95 ℃ 5 min;95 ℃ 1 min,52/55/60 ℃ 1 min(不同引物退火温度不同),72 ℃ 1 min,30个循环;72 ℃ 5 min。利用毛细管电泳检测产物大小,将产物碱基数与上下游序列碱基数的差值除以核心序列的长度得出重复数,将数据整理成Excel表格,导入BioNumerics 6.6软件。
将鹤庆县分离的10株鼠疫菌以及邻近疫源地分离的93株鼠疫菌(玉龙县12株、古城区1株、剑川县18株、祥云县1株、普洱市17株、临沧市34株、保山市10株)共计103株鼠疫菌和2株假结核耶尔森菌IP32953和IP31758纳入聚类分析。运行Clustering下的Advanced cluster analysis中的MST for categorical data生成最小生成树(Minimum spanning tree analysis,MST)。
结果1. DFR分型:鹤庆县分离的10株菌DFR型别均为DFR 01、02、13、23位点缺失。将其与数据库DFR分型数据比较(表 1),结果显示,与丽江菌株有相同的DFR型别[3],按照杨晓艳等[10]的定义,为Genomovar 05型。
2. CRISPRs分型:鹤庆县10株菌CRISPRs分型中,YPa排序为“a1-a2-a3-a4-a5-a6-a7”,YPb为“b1-b2-b3-b4”,YPc为“c1-c2-c3”。将其与数据库CRISPRs分型数据比较(表 2),结果显示与丽江菌株有着相同的型别[4],按照Cui等[7]的定义,为基因簇Ca7(基因型22)。
3. MLVA分型:103株鼠疫菌被分为3个簇,分别是丽江野鼠鼠疫簇、剑川野鼠鼠疫簇及家鼠鼠疫簇(分离自祥云县的1株菌与邻近菌株位点差异数较大未包含在簇内)。鹤庆县分离的10株菌位于丽江野鼠鼠疫簇内,其中9株菌MLVA基因型相同,与另外1株菌(HQ1)有1个位点(M22)的差异。丽江菌株2006-7号位于整个丽江野鼠鼠疫簇的核心位置,鹤庆县分离的9株菌与之同源性最高,仅有2个位点(N2117,M23)的差异。见图 1。
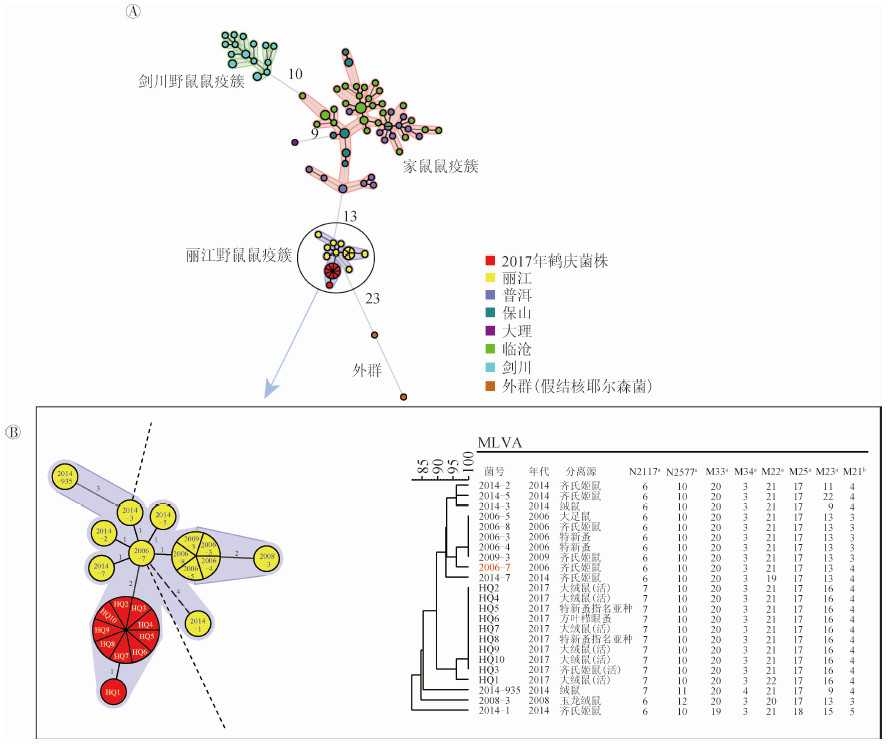
|
| 注:红色圆圈内为鹤庆县分离株;A.103株鼠疫菌和2株假结核耶尔森菌的最小生成树(MST),以菌株分离地点创建Group赋予不同颜色,圆的大小代表菌株数量的多少,同一圆内的菌株具有相同的MLVA基因型,有相同底色的圆为一个簇,同一簇内的位点差异数不超过6;B.丽江野鼠鼠疫簇的MST和丽江野鼠鼠疫簇有差异的VNTR位点;a为MLVA12位点,b为MLVA14位点 图 1 103株鼠疫菌和2株假结核耶尔森菌基于MLVA26个位点的聚类分析 |
鹤庆县位于云南省西北部,大理白族自治州北端,与之毗邻的剑川县、丽江市恰好分别是剑川野鼠鼠疫疫源地和丽江野鼠鼠疫疫源地的范畴。这两块疫源地虽然从地理生境、主要宿主和媒介都较相似,但疫源地菌株在生化特性[11]、分子分型特征上[12-13]有着质的差异,是两块不同的疫源地。鹤庆县处于两地之间,其分离菌株为何种性质的菌株必然成为我们关注的问题,本研究通过分离菌株的基因分型初步给出了答案。
细菌基因组多态性产生有着不同的遗传机制,而DFR、CRISPRs、MLVA这3种分型方法对应不同的产生机制:DFR对应基因水平转移和缺失;MLVA对应短重复序列,CRISPRs极性化记录噬菌体入侵事件,3种分型方法从不同角度描述细菌基因组多态性。本研究中,鹤庆县分离的鼠疫菌株在DFR、CRISPRs分型上与丽江野鼠疫源地菌株一致,在MLVA聚类分析中与丽江菌株聚为同一个簇。从遗传的角度看,鹤庆菌株与丽江野鼠疫源地菌株属于同一种性质的菌株,3种分型方法得出一致的结论,相互印证。
以往的研究表明[3-5],云南省鼠疫菌DFR、CRISPRs、MLVA分型能区分菌株生态型,且MLVA(尤其是MLVA12)蕴含大量多态信息,具有很高的分辨率,不仅能区分生态型,而且能识别个体特征,适用于溯源。鹤庆县所分离的10株菌中,有9株为同一个MLVA基因型,与另1株菌仅有1个MLVA位点的差异,表明这起发生在云南省鹤庆县草海镇马厂村北坡脚、西坡脚,历时近半月,波及齐氏姬鼠、大绒鼠及其蚤的鼠间疫情属于同一次流行。
MLVA聚类分析还表明,2006年分离的菌株是丽江野鼠疫源地较古老的菌株,其随着时间的推移及地点的变迁,发生VNTR位点或多或少(1~4)的改变,这种改变以MLVA12位点为主(仅有1个MLV14位点),这与Li等[9]的研究结果一致;同时,丽江疫源地大部分菌株包括新分离的鹤庆菌株都与2006年菌株同源,表明2006年菌株的基因特征是该疫源地基因的主特征。鹤庆菌株与2006年丽江菌株在N2117及M23位点上有差异,这两个位点的核心序列分别为“ATGTGCCTG”和“CAAATAG”,前者位于鼠疫菌基因组编码区,后者位于非编码区,两者重复数增加对鼠疫菌将产生何种影响有待进一步研究确认。所有菌株的MST表明,在云南省3种疫源地菌株中,丽江野鼠鼠疫菌与外群假结核耶尔森菌的关系最近,侧面说明从进化的角度来说,丽江鼠疫菌出现得最早,这与CRISPRs分析的结果相吻合[4]。
自2006年从玉龙县鹿子村分离到鼠疫菌以来,丽江野鼠鼠疫疫源地范围逐年扩大[14],覆盖了玉龙县的鹿子、满下、满上、满中及汝南化,以及古城区的木梳。鹤庆疫情可能是丽江鼠疫进一步向南扩散的结果。丽江疫源地菌株从生化特性、基因分型比对等均表明与喜马拉雅旱獭鼠疫青藏高原型相似[15-16],认为其毒力强,可引发肺鼠疫流行,对人类威胁较大。鹤庆菌株与丽江菌株具有相同的基因型,对人类也存在着同样的威胁。鹤庆鼠疫能在活鼠中流行,疫情较为隐秘,且菌株分离地分布于马厂村外300 m林地,属于人类生产生活的范围,因此,应高度重视鹤庆县的鼠疫防控,并加强不明原因肺炎的监测、报告。
值得注意的是,虽然鹤庆菌株和丽江菌株的分离宿主都是野鼠(齐氏姬鼠、大绒鼠),但前者从活鼠中检出而后者均从自毙鼠里发现[17],既然两者是同样类型的菌株,又怎么解释两地的宿主动物对菌株有着不同的耐受性?对于鹤庆鼠间疫情,尚有许多方面亟待研究,比如分离菌株的生化特性和毒力、主要宿主和主要媒介及疫源地生态系统等。通过全面系统的研究,将有利于更深刻地理解鹤庆菌株遗传、进化及保存机制,促进对云南省鼠疫流行演化规律的认识。
利益冲突: 无
| [1] |
中国医学科学院流行病学微生物学研究所. 中国鼠疫流行史[M]. 北京: 人民卫生出版社, 1981: 749–926.
Institute of Epidemiology and Microbiology, Chinese Academy of Medical Sciences.Epidemiological History of Plague in China[M]. Beijing: People's Health Publishing House, 1981: 749–926. |
| [2] | Qi ZZ, Cui YJ, Zhang QW, et al. Taxonomy of Yersinia pestis[C]//Yang R, Anisimov A. Yersinia pestis: Retrospective and Perspective. Dordrecht: Springer, 2016, 918: 35-78. DOI: 10.1007/978-94-024-0890-4_3. |
| [3] |
朱俊洁, 王鹏, 李伟, 等. 云南省鼠疫菌株差异片段基因分型及其流行病学特征[J]. 中华地方病学杂志, 2013, 32(6): 599–601.
Zhu JJ, Wang P, Li W, et al. Genotyping of Yersinia pestis by different regions and its epidemiological characteristics in Yunnan province[J]. Chin J Endemiol, 2013, 32(6): 599–601. DOI:10.3760/cma.j.issn.2095-4255.2013.06.004 |
| [4] |
杨光璨, 石丽媛, 郭英, 等. 云南省鼠疫耶尔森菌规律成簇间隔短回文重复序列分型[J]. 中华流行病学杂志, 2014, 35(8): 974.
Yang GC, Shi LY, Guo Y, et al. Genotyping on Yersinia pestis isolated from Yunnan province by clustered-regularly-interspaced-short palindromic-repeats[J]. Chin J Epidemiol, 2014, 35(8): 974. DOI:10.3760/cma.j.issn.0254-6450.2014.08.022 |
| [5] |
朱俊洁, 王鹏, 张蓉, 等. 云南省鼠疫耶尔森菌多位点可变数目串联重复序列分析[J]. 疾病监测, 2013, 28(10): 848–852.
Zhu JJ, Wang P, Zhang R, et al. Multiple-locus variable-number tandem-repeat analysis on Yersinia pestis in Yunnan[J]. Dis Surveill, 2013, 28(10): 848–852. DOI:10.3784/j.issn.1003-9961.2013.10.016 |
| [6] | Li YJ, Dai E, Cui YJ, et al. Different region analysis for genotyping Yersinia pestis isolates from China[J]. PLoS One, 2008, 3(5): e2166. DOI:10.1371/journal.pone.0002166 |
| [7] | Cui YJ, Li YJ, Gorgé O, et al. Insight into microevolution of Yersinia pestis by clustered regularly interspaced short palindromic repeats[J]. PLoS One, 2008, 3(7): e2652. DOI:10.1371/journal.pone.0002652 |
| [8] | Grissa I, Vergnaud G, Pourcel C. CRISPR= Finder:a web tool to identify clustered regularly interspaced short palindromic repeats[J]. Nucleic Acids Res, 2007, 35(W1): W52–57. DOI:10.1093/nar/gkm360 |
| [9] | Li YJ, Cui YJ, Cui BZ, et al. Features of variable number of tandem repeats in Yersinia pestis and the development of a hierarchical genotyping scheme[J]. PLoS One, 2013, 8(6): e66567. DOI:10.1371/journal.pone.0066567 |
| [10] |
杨晓艳, 魏柏青, 靳娟, 等. 中国鼠疫耶尔森菌差异区段分型及其地理分布特征[J]. 中华流行病学杂志, 2014, 35(8): 943–948.
Yang XY, Wei BQ, Jin J, et al. Regional genotyping and the geographical distribution regarding Yersinia pestis isolates in China[J]. Chin J Epidemiol, 2014, 35(8): 943–948. DOI:10.3760/cma.j.issn.0254-6450.2014.08.015 |
| [11] |
郭英, 张丽云, 夏连续, 等. 云南省玉龙县鼠疫菌的生化特性[J]. 地方病通报, 2008, 23(3): 12–14.
Guo Y, Zhang LY, Xia LX, et al. Biochemical characters of Yersinia pestis isolated from Yulong county of Yunnan province in China[J]. Endem Dis Bull, 2008, 23(3): 12–14. DOI:10.3969/j.issn.1000-3711.2008.03.004 |
| [12] |
石丽媛, 叶蕊, 董珊珊, 等. 云南省鼠疫菌FseⅠ酶切分型及其流行病学意义[J]. 中华流行病学杂志, 2014, 35(2): 182–185.
Shi LY, Ye R, Dong SS, et al. Genotyping and its epidemiological significance on Yunnan Yersinia pestis under FseⅠ enzyme digestion method[J]. Chin J Epidemiol, 2014, 35(2): 182–185. DOI:10.3760/cma.j.issn.0254-6450.2014.02.018 |
| [13] |
方喜业, 周冬生, 崔玉军, 等. 中国鼠疫自然疫源地分型研究Ⅲ.鼠疫耶尔森菌DFR/MLVA主要基因组型生物学特征[J]. 中华流行病学杂志, 2012, 33(5): 536–539.
Fang XY, Zhou DS, Cui YJ, et al. Eco-geographicl and scapes of natural plague foci in China Ⅲ. Biological charactedsfics of major DFR/MLVA-based genotypes of Yersinia pestis, China[J]. Chin J Epidemiol, 2012, 33(5): 536–539. DOI:10.3760/cmaj.issn.0254-6450.2012.05.021 |
| [14] |
张正飞, 杨焕, 张福新, 等. 丽江野鼠鼠疫自然疫源地10年扩散趋势研究[J]. 国外医学医学地理分册, 2017, 38(4): 328–331.
Zhang ZF, Yang H, Zhang FX, et al. A 10-year study of plague natural foci diffusion trend in Lijiang[J]. For Med Sci Sect Medgeogr, 2017, 38(4): 328–331. DOI:10.3969/j.issn.1001-8883.2017.04.005 |
| [15] | Zhang XA, Hai R, Wei JC, et al. MLVA distribution characteristics of Yersinia pestis in China and the correlation analysis[J]. BMC Microbiol, 2009, 9: 205. DOI:10.1186/1471-2180-9-205 |
| [16] |
申小娜, 王琪, 夏连续, 等. 我国新发现的鼠疫自然疫源地鼠疫菌基因组序列测定及分析[J]. 中国地方病学杂志, 2011, 30(5): 476–480.
Shen XN, Wang Q, Xia LX, et al. Comparison of Yersinia pestis genomes from a new natural foci in China[J]. Chin J Endemiol, 2011, 30(5): 476–480. DOI:10.3760/cma.j.issn.1000-4955.2011.05.002 |
| [17] |
张正飞, 张福新, 和映天, 等. 丽江野鼠鼠疫疫源地6年疫情发生监测及防治措施研究[J]. 国外医学医学地理分册, 2012, 33(3): 154–158.
Zhang ZF, Zhang FX, He YT, et al. Research on the six-year monitoring and control measures in epidemic wild rodent plague foci in Lijiang[J]. For Med Sci Sect Medgeogr, 2012, 33(3): 154–158. DOI:10.3969/j.issn.1001-8883.2012.03.004 |
 2018, Vol. 39
2018, Vol. 39




