文章信息
- 黄涛, 李立明.
- Huang Tao, Li Liming.
- 系统流行病学
- Systems epidemiology
- 中华流行病学杂志, 2018, 39(5): 694-699
- Chinese Journal of Epidemiology, 2018, 39(5): 694-699
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2018.05.031
-
文章历史
收稿日期: 2018-03-11
流行病学通常被认为是公共卫生的基础科学。人类疾病遗传与环境的病因学研究一直是流行病学的重要任务。然而,传统黑箱子流行病学往往侧重于识别单一危险因素,很少聚焦网络对疾病的影响,这种单一的研究有严重的局限性。随着新发现的危险因素越来越多,通过研究疾病的单一风险因素,不太可能深入了解风险因素及其交互作用所导致的网络动态变化在疾病发生发展中的作用[1]。我们渴望打开黑箱子,阐明致病因素如何通过病因链环节而导致疾病发生、发展。因此,新的系统分析方法必将在复杂疾病病因的研究中起到越来越重要的作用。
高通量组学技术的发展成熟,使得流行病学家有机会将系统生物学理论方法与流行病学有机结合,借助于丰富的组学数据、临床医学大数据,系统阐明暴露到疾病结局的致病机制,这有助于将复杂数据转化为与医学和公共卫生相关的知识[2]。从而催生了一个崭新的流行病学分支学科——系统流行病学。虽然系统思维已被生物医学所接受,但在公共卫生的应用尚未形成。因此,本文详细介绍了系统流行病学的理论基础、概念、研究目的、研究内容、研究意义、研究设计、分析方法及其在公共卫生领域的应用。
二、系统理论、系统生物学以及系统医学在认识系统流行病学之前,先介绍系统理论、系统生物学和系统医学的主要概念及其演变过程,再详细讨论系统流行病学。
系统科学起源于20世纪上半叶出现的系统理论,是由美籍奥地利人理论生物学家贝塔朗菲(Ludwig von Bertalanffy)(1901-1972)创立的。他在1932年提出“开放系统理论”和“系统论”(system approach)的思想,1937年提出了一般系统论原理,奠定了这门科学的理论基础。真正确立这门科学学术地位的是1968年贝塔朗菲发表的专著《一般系统理论:基础、发展和应用》。贝塔朗菲认为任何系统都是一个有机的整体,不是各个部分的机械组合或简单相加,系统的整体功能是各要素在孤立状态下所没有的性质,即整体大于部分之和。系统论把所研究和处理的对象当作一个整体,分析其结构和功能,研究系统、要素、环境三者的相互关系和变动(互动)的规律性,用优化系统观点看问题。系统论正渗透到每个领域,但在公共卫生领域尚未形成,利用系统论的思想解决我国公共卫生问题具有现实意义。
系统论已在生物学中有广泛应用。系统方法能用动态的分析识别生物系统在复杂的环境因素里产生的动态行为过程[2]。随着现代生物技术的发展和计算数学的进步,一门新的学科“系统生物学”(systems biology)随之产生。最早,贝塔朗菲将系统生物学称为“有机生物学”。美国生物学家Leroy Hood博士于2004把系统生物学定义为:研究一个生物系统中所有组成成分的构成,以及在特定条件下这些组分间的相互关系并通过计算生物学建立一个数学模型来定量其相互关系,预测生物的功能、表型和行为的学科。系统生物学是生物学的一个新领域,其目的是实现从系统水平上认识“基因、蛋白质和代谢物的相互关系(组织和结构)和交互作用(动态或行为)”,在系统层面上理解生物系统。现代生物技术以及计算机技术构成系统生物学发展的技术基础。系统生物学的发展依赖新技术进步的同时,也会推动技术发展、多学科协作。
系统生物学原理在复杂疾病病因学研究和诊断中的应用被称为“网络医学”,也有人将其称为系统医学(systems medicine)。系统医学采用系统论方法研究生物医学,使用分子诊断来分层患者和疾病,以更好地理解疾病的复杂性,也就是系统生物学的医学应用研究。系统医学全方位、立体化、多视角研究生命与疾病全过程,揭示人体的生理和病理机制,并提出疾病的诊断和防治措施与方法。系统医学或能克服目前药物研发的局限性。系统医学的研究不仅能帮助我们理解药物在不同表型的个体中发挥的药效不同,还可以帮助我们根据个体的药物代谢特点为其定制个性化治疗方案。
三、系统流行病学的定义和内涵传统流行病学关注疾病单个危险因素的研究,难以发现危险因素与由此产生的动态网络改变间潜在的重要交互作用。危险因素间的网络关系可能无法被发现。尤其是以心脑血管疾病和肿瘤为代表的慢性病,其病因复杂、遗传和环境交互作用常见,对于想要揭示复杂生化通路的研究者,需要的是新的分析性和系统性的方法,系统流行病学(systems epidemiology)应运而生。
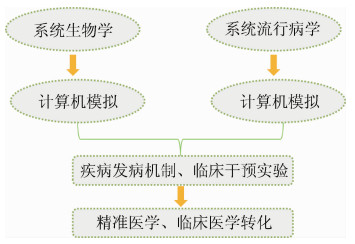
系统流行病学与系统生物学、系统医学都是建立在系统论的基础之上,但又有区别。系统生物学是典型的多学科交叉研究,需要将分子、细胞、组织及器官等多层次的生物功能进行整合,借助计算模型进一步加深对生物体一般规律的理解。系统医学更加关注疾病的发生、发展及其干预。系统流行病学在系统医学的基础上,更加关注疾病的危险因素(图 1)。在病因学和预防医学研究中,这种系统性的思维模式与多因多果的病因模型更为契合,因为它可以在多水平发现疾病的危险因素,还可以探究其交互作用的网络关系。
|
| 图 1 系统生物学与系统流行病学的比较 |
系统流行病学是一种进行疾病危险因素风险识别的流行病学方法,不是传统流行病学方法的替代,而是一种补充和完善,且具有传统流行病学所不具备的优势。系统流行病学是传统遗传学和分子流行病学的整合,是机体内环境与疾病发生之间关系的研究;是传统行为危险因素流行病学和健康环境决定因素的整合,寻找疾病危险因素与环境的交互作用;是数据计算方法研究与流行病学研究的整合,通过建模反映观察到的危险因素之间、危险因素与环境因素之间、因素与疾病之间的复杂作用与关系,为疾病预防、预警、预测提供科学依据。其研究内容包括:系统水平(如各种组学)的暴露测量;涉及可能影响健康和疾病的行为、社会人口学和群体水平以及生态环境和进化因素等多层面因素的分析;危险因素之间相互关系的网络分析;疾病发生风险的统计学模型的建立以及未来风险状况的计算模拟和预警预测[2]。人体是众多网络构成的网络(Network of Networks),传统流行病学的方法是在单一维度上的建模,而系统流行病学的方法适应多维度的数据、多样的数据类型,构建整合的模型,允许信息在不同维度上流动,全面理解生物系统。
四、系统流行病学的研究内容系统流行病学研究内容的核心是整合,这里的整合包括多重含义。第一,把系统内不同性质的构成要素(DNA、mRNA、蛋白质、生物小分子等)整合在一起进行研究。第二,对于多细胞生物,要实现从基因到细胞、器官、组织甚至是个体的各个层次的整合。第三,研究思路和方法的整合:①垂直型研究:经典的分子生物学研究采用多种手段研究个别的基因和蛋白质;②水平型研究:基因组学、蛋白质组学和其他各种“组学”是以单一的手段同时研究成千上万个基因或蛋白质;③“三维”研究:把水平型研究和垂直型研究整合起来。第四,整合生物学研究、暴露组学研究和临床、人群健康和疾病结局的研究,并通过大数据、多水平的计算模型建立,对疾病发生风险进行预警预测。
系统流行病学对多学科整合有较高要求。由于基本假设、思维方式以及不同学科的实验室使用的科学语言极其不同,系统流行病学中各学科间的理解与沟通、甚至学术语言的理解与共识都存在着困难。然而,改善实验家和系统生物学建模者之间、流行病学家和系统流行病学建模者之间、以及4类研究者之间的沟通交流,将会产生通用词汇表和互利的启示。
五、系统流行病学的研究意义系统流行病学研究有着重要的临床和公共卫生意义。系统流行病学方法可以通过流行病学观察研究,更深入地了解分子途径,并指导早期疾病的筛检、临床诊断和预后,并有助于个性化预防和治疗;将揭开暴露与结局之间的“黑箱子”,补充和完善传统流行病学研究方法的局限性;不仅包括分子标记物的测量,还包括对可能影响健康和疾病的行为、社会人口学和群体水平以及生态环境和进化因素的研究;不仅包括各种暴露组学的研究,还包括计算机疾病模型或健康风险模型的建立。这种多水平、多层次因素研究的整合对发现疾病的致病因素和机制会起到积极的推动作用;数据计算研究将为系统流行病学家提供模拟观察到的危险因素间相互作用的能力,从而阐明暴露和导致人类疾病的结局进程之间极其复杂的相互作用。
在病因和预防研究中,系统性思考多重因果关系将使流行病学家能够在多个层面上识别疾病的病因及其相互作用。在公共卫生领域,系统流行病学将有助于改善疾病监测方法。因此,我们应该鼓励流行病学方法和计算模拟的结合,并创建复杂的计算模拟模型,整合疾病病因,病原性数据以及来自不同学科的研究者的专业知识的信息。
六、系统流行病学的研究方法系统流行病学方法不仅涉及测量人类疾病的分子基础,还涉及测量多种环境相互作用成分,包括可能影响健康和疾病的行为、社会人口学和群体水平的环境因素(图 2)。系统流行病学能够整合多组学新技术产生的组学数据、临床医学大数据以及观察性流行病学的环境大数据,在分子、细胞、组织、人群和社会环境等多水平、多组学上深入研究人类的健康和疾病[1]。

|
| 图 2 系统流行病学的原理 |
1.系统流行病学的多层次、全组学设计:系统流行病学研究中的多层次、全组学设计为复杂疾病病因研究提供了新方法。全组学研究设计以流行病学前瞻性队列研究为基础,利用基因数据库中有关DNA、RNA和血浆的信息以及队列研究开始到出现结局之间的问卷信息,对诊断前血液样品进行分析;可以观察到复杂的真实生活条件下的基因表达谱,不同的暴露与遗传易感性交互作用和患病风险[3]。随着生物技术的不断成熟和系统生物学的发展,全组学设计不断将基因变异、基因表达和修饰、蛋白质以及信号和代谢途径等多方面的信息在不同层次上加以整合,并与具有人群特征的流行病学前瞻性研究设计相结合,形成一个新颖的系统流行病学的设计方法,为更加全面和深入地认识暴露与结局之间的生物学机制提供了新的方法。
2.系统流行病学分析方法:系统流行病学数据来源的多样性、复杂性以及大数据的特征,为系统流行病学的设计和分析方法提出了新的挑战。目前,系统流行病学应用较多的方法是网络分析法。网络分析方法为系统流行病学中的数据整合和生物标志物选择提供了计算框架[1]。因此,有必要将人群水平的多组学大数据进行整合并试图寻找到新的跨组学的新的标志物。
(1)网络差异法:系统流行病学强调用高通量全组学大数据、临床医学大数据以及环境大数据,这对统计分析方法提出更高要求。系统流行病学的统计分析的核心是在人群水平上通过“暴露因素-组学标志物-疾病结局”的网络/通路的组间差异统计学比较,推断危险因素导致疾病发生、发展的网络效应和网络结构。在实际工作中,常用2种不同研究策略来推断网络差异:①基于高通量组学技术的数据驱动的研究策略(Data-Driven):该策略在当今大数据时代应用较多,在不受任何假设限制的情况下,充分利用系统生物学的网络分析方法构建“暴露因素-组学标志物-疾病结局”网络模型,并检验对比组间网络差异及其效应。为进一步的实验验证、药物靶点确定、制定精准的预防或诊疗措施提供理论依据。②基于流行病学方法的假设驱动的研究策略(Hypothesis-Driven):可在理解疾病的机制上,综合以往实验的结果,借助网络数据库通路信息,推断出一个假定合理的致病网络机制,并加以验证。该方法可应用到设计药物靶点、预测疾病发生预后、制定精准的预防或诊疗策略等。但是无论是数据驱动还是假设驱动的研究策略,仍存在或多或少的缺点。由于数据来源的复杂性、多样性,我们急需发展和应用更加科学的系统流行病学设计方法和分析方法,这也为系统流行病学家提出了新的挑战和机遇。
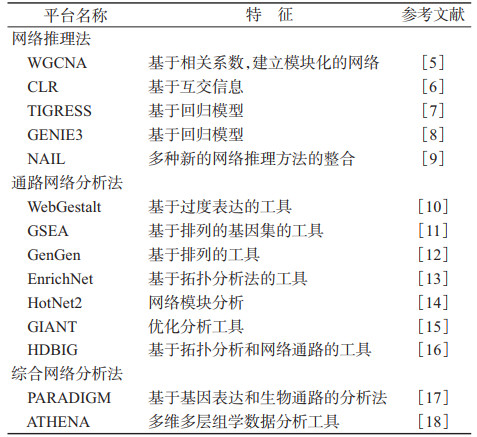
(2)可视化多层网络分析法:多组学数据的整合分析是相当复杂的,需要基于网络的系统分析方法等强大技术。计算网络法在整合多组学数据上有很好的应用前景。近年来,尽管越来越多的网络分析方法得以应用(表 1),复杂的多层多组学数据仍然需要更优化的方法才能被有效利用。更加先进的计算网络分析方法正在不断的更新完善中。例如,MONGKIE(模块化网络生成和知识集成的可视化环境平台)是基于生物网络的组学数据的可视化和分析的通用平台,集成了网络可视化组学数据分析工具的无缝连接。此外,该平台还集成了多样的内部算法、网络分析,包括网络聚类等分析[4]。
随着多组学技术的不断发展,在未来人类疾病研究中,开发和应用更先进的网络处理数据的技术以及分析更多层的组学数据的方法是非常有必要的。与传统的单一生物层次分析相比,系统的多组学分析是一门新的学科,具有更高的维度和复杂性,适合于复杂疾病的研究。有效的多组学研究需要科学家的广泛合作,包括疾病专家、计算机科学家、生物信息学家、生物学家和其他多科学家。新的网络分析方法和策略正在不断出现,在不久的将来,我们一定能更好地理解复杂疾病背后的重要生物过程。
(3)多层次、多组学数据的整合方法:目前的网络分析方法越来越受到关注。然而,大多数研究仍然采用单组学层内分析,并且没有解决多组学层之间的连接和网络。尽管通路和网络分析使用了一个以上的组学数据,但在一定程度上,没有将多层多组学数据组合成一个有机的整体框架,不能全面诠释生物系统。因此,缺乏综合分析的研究对疾病的认识仍然有限。
如何整合多层多组学数据?仍然是系统流行病学面临的一个重要问题。已经出现了若干整合多维多层组学数据的方法,比如TCGA分析平台。TCGA平台综合利用30多种肿瘤的多维度的基因组学数据(包括基因变异数据、拷贝数基因、表观遗传学数据、基因表达数据、miRNA测序数据等),挖掘与肿瘤相关的基因网络。TCGA应用多组学综合分析方法,发现了3种与子宫癌相关的独特分子生物学特征组合。同样的方法也在肺癌、卵巢癌、前列腺癌的研究中得到应用。尽管该方法仍存在争议,但这些研究结果使我们更加深入的认识到癌症的发病机制。要充分利用大数据时代的复杂数据,开发用于整合数据的方法仍然是系统流行病学的重要任务。
七、系统流行病学的应用1.传染病系统流行病学:传染病研究者提出了系统的流行病学发展更全面的结核病理解的作用[19-20]。系统生物学注重传染病的生物学机制,但系统流行病学包括流行病学、社会学、进化生物学和生态学等方面。计算研究能为传染病流行病学家提供系统模型来研究多层次危险因素之间的网络关系以及相互作用,从而阐明暴露和传染病之间复杂的相互关系。事实上,将数学建模/仿真集成在一起的想法在传染病流行病学研究已经有广泛应用[21]。
以结核病为例,结核分枝杆菌的生物学机制、结核病原体及其与人类宿主相互作用的机制仍不清楚。系统流行病学将有助于填补这些空白。新的高通量技术已经被引入到传染病的研究中。要全面了解结核病并在全球范围内控制该疾病,不仅要考虑系统生物学的因素,还要考虑宿主和病原体多样性、人口统计学变化、病原耐药改变以及社会经济和环境因素的影响。这种跨学科的方法被描述为“传染病系统流行病学”以克服传统生物学和传统流行病学之间的界限,更全面更系统的方法能够了解结核病的复杂性,为推动结核病新药和疫苗的发展至关重要。
2.癌症系统流行病学:癌症流行病学的前瞻性研究一直沿用了过去几十年的传统研究设计。研究基因与环境的相互作用与癌症的流行病学是基于遗传变异和生物标志物等数据的生物样本库。这些研究是使用了高通量组学技术,而不是开发新的设计策略。Lund和Dumeaux[3]提出了全组学设计的理念,覆盖了来自于血样和癌症组织的全组学数据,并将前瞻性队列设计的数据与组学数据如转录组学数据整合在一起。因此,开辟了一条研究癌症流行病学中因果关系的新途径。通过全组学设计,mRNA和miRNA分析能够打开黑箱子,并深入阐明生物学通路的机制,甚至增加对癌症流行病学因果关系的理解。这种全组学设计理念为系统的整合多层次多组学的数据提供了新思路,可以打开系统流行病学在癌症流行病学中应用的新研究领域。
3.营养系统流行病学:系统流行病学在营养学研究中的应用能够更加全面系统的研究食物与健康的关系及其机制,并有可能激发新的营养学研究方法的产生。应用于2型糖尿病和相关病症研究中的系统方法发现了可能通过饮食调控的新通路。展望未来,如果能充分利用现有生物大样本、大型前瞻性研究的数据以及多次测量的饮食和生活方式等资源并系统整合各层次数据,营养系统流行病学的结果将大大改善个性化的营养预防和治疗,最终推动营养学走向更加精准的方向。Cornelis和Hu[22]建议,将代谢组学数据纳入糖尿病流行病学研究。将高通量分析技术应用到新型代谢生物标志物的人类观察研究,有利于使流行病学的研究手段从传统的黑箱子战略到系统方法学的转变。但它是否可以改善早期疾病的监测、临床诊断、预后以及个性化预防和治疗仍有待进一步研究。
综上所述,系统流行病学作为流行病学的分支新学科,其在公共卫生领域的应用将越来越受到重视。在今后的系统流行病学应用中,为了弥合系统生物学和传统流行病学之间的差距,必须将人类丰富的组学数据和社会经济、环境及其相互作用的数据紧密的结合起来。
八、系统流行病学的思考与展望计算建模对系统流行病学研究至关重要,计算建模研究有助于我们更进一步了解疾病的发生与发展。最重要的是,计算建模的模拟结果能让系统流行病学家深入了解到不同层面危险因素交互作用,从而阐明复杂的相互作用导致人类疾病的整个过程。然而,详细地模拟多层次的研究设计是非常困难的。目前,仍有待解决的问题是如何设计和进行层内交互作用的网络分析[21]。
系统流行病学面临的另一个重要挑战可能是缺乏组学数据如生物标志物等的标准化和统一。随着先进的计算建模技术的使用,需要良好的集成平台和数据集来融合高度动态的数据参数,并评估组学级表型与其他表型之间的关系。此外,所应用的研究设计要能够进行动态暴露因子的评估,并需要将实验数据与理论建模之间进行紧密的融合[23]。
最重要的是,如何整合系统生物学与系统流行病学?我们期望系统生物学的致病机制的计算模型能很快应用到模拟人类疾病发生的计算病因学模型中。然而,整合的关键在于跨学科的支持合作。只有系统生物学家、流行病学家以及计算机学家加强合作,才能有助于将不同学科的基本假设、思维模式和科学语言有机的结合在一起,建立更接近现实的计算模型来模拟人类疾病发生发展的病因学模型,并最终揭示疾病发病机制[21]。
九、结论系统流行病学使我们能够更好地描述影响复杂疾病的各种因素及其相互关系的网络。实现这一目标的关键是广泛来源的大数据、来自不同层级的结构和功能组织模型的整合以及大数据的网络分析处理方法的应用。因此,发展新的设计方法和先进的统计分析方法是系统流行病学的重要任务。
利益冲突: 无
| [1] | Haring R, Wallaschofski H. Diving through the "-Omics":the case for deep phenotyping and systems epidemiology[J]. OMICS A J Int Biol, 2012, 16(5): 231–234. DOI:10.1089/omi.2011.0108 |
| [2] | Dammann O, Gray P, Gressens P, et al. Systems epidemiology:what's in a name?[J]. Online J Public Health Inform, 2014, 6(3): e198. DOI:10.5210/ojphi.v6i3.5571 |
| [3] | Lund E, Dumeaux V. Systems epidemiology in cancer[J]. Cancer Epidemiol Biomarkers Prev, 2008, 17(11): 2954–2957. DOI:10.1158/1055-9965.EPI-08-0519 |
| [4] | Jang Y, Yu N, Seo J, et al. MONGKIE:an integrated tool for network analysis and visualization for multi-omics data[J]. Biol Direct, 2016, 11(1): 10. DOI:10.1186/s13062-016-0112-y |
| [5] | Langfelder P, Horvath S. WGCNA:an R package for weighted correlation network analysis[J]. BMC Bioinformatics, 2008, 9: 559. DOI:10.1186/1471-2105-9-559 |
| [6] | Faith JJ, Hayete B, Thaden JT, et al. Large-scale mapping and validation of Escherichia coli transcriptional regulation from a compendium of expression profiles[J]. PLoS Biol, 2007, 5(1): e8. DOI:10.1371/journal.pbio.0050008 |
| [7] | Haury AC, Mordelet F, Vera-Licona P, et al. TIGRESS:trustful inference of gene regulation using stability selection[J]. BMC Syst Biol, 2012, 6: 145. DOI:10.1186/1752-0509-6-145 |
| [8] | Huynh-Thu VA, Irrthum A, Wehenkel L, et al. Inferring regulatory networks from expression data using tree-based methods[J]. PLoS One, 2010, 5(9): e12776. DOI:10.1371/journal.pone.0012776 |
| [9] | Hurley DG, Cursons J, Wang YK, et al. NAIL, a software toolset for inferring, analyzing and visualizing regulatory networks[J]. Bioinformatics, 2015, 31(2): 277–278. DOI:10.1093/bioinformatics/btu612 |
| [10] | Wang J, Duncan D, Shi ZA, et al. WEB-based gene set analysis toolkit (webgestalt):update 2013[J]. Nucleic Acids Res, 2013, 41(W1): W77–83. DOI:10.1093/nar/gkt439 |
| [11] | Mootha VK, Lindgren CM, Eriksson KF, et al. PGC-1α-responsive genes involved in oxidative phosphorylation are coordinately downregulated in human diabetes[J]. Nat Genet, 2003, 34(3): 267–273. DOI:10.1038/ng1180 |
| [12] | Wang K, Li MY, Bucan M. Pathway-based approaches for analysis of genomewide association studies[J]. Am J Hum Genet, 2007, 81(6): 1278–1283. DOI:10.1086/522374 |
| [13] | Glaab E, Baudot A, Krasnogor N, et al. EnrichNet:network-based gene set enrichment analysis[J]. Bioinformatics, 2012, 28(18): i451–457. DOI:10.1093/bioinformatics/bts389 |
| [14] | Leiserson MDM, Vandin F, Wu HT, et al. Pan-cancer network analysis identifies combinations of rare somatic mutations across pathways and protein complexes[J]. Nat Genet, 2015, 47(2): 106–114. DOI:10.1038/ng.3168 |
| [15] | Greene CS, Krishnan A, Wong AK, et al. Understanding multicellular function and disease with human tissue-specific networks[J]. Nat Genet, 2015, 47(6): 569–576. DOI:10.1038/ng.3259 |
| [16] | Yan JW, Du L, Kim S, et al. Transcriptome-guided amyloid imaging genetic analysis via a novel structured sparse learning algorithm[J]. Bioinformatics, 2014, 30(17): i564–571. DOI:10.1093/bioinformatics/btu465 |
| [17] | Vaske CJ, Benz SC, Sanborn JZ, et al. Inference of patient-specific pathway activities from multi-dimensional cancer genomics data using PARADIGM[J]. Bioinformatics, 2010, 26(12): i237–245. DOI:10.1093/bioinformatics/btq182 |
| [18] | Holzinger ER, Dudek SM, Frase AT, et al. ATHENA:the analysis tool for heritable and environmental network associations[J]. Bioinformatics, 2014, 30(5): 698–705. DOI:10.1093/bioinformatics/btt572 |
| [19] | Comas I, Gagneux S. A role for systems epidemiology in tuberculosis research[J]. Trends Microbiol, 2011, 19(10): 492–500. DOI:10.1016/j.tim.2011.07.002 |
| [20] | Fenner L, Egger M, Gagneux S. Annie Darwin's death, the evolution of tuberculosis and the need for systems epidemiology[J]. Int J Epidemiol, 2009, 38(6): 1425–1428. DOI:10.1093/ije/dyp367 |
| [21] | Dammann O, Gray P, Gressens P, et al. Systems Epidemiology:What's in a Name?[J]. Online J Public Health Info, 2014, 6(3): e198. DOI:10.5210/ojphi.v6i3.5571 |
| [22] | Cornelis MC, Hu FB. Systems epidemiology:a new direction in nutrition and metabolic disease research[J]. Curr Nutr Rep, 2013, 2(4): 225–235. DOI:10.1007/s13668-013-0052-4 |
| [23] | Haring R, Wallaschofski H. Diving Through the "-Omics":The Case for Deep Phenotyping and Systems Epidemiology[J]. OMICS, 2012, 16: p. 3. DOI:10.1089/omi.2011.0108 |
 2018, Vol. 39
2018, Vol. 39