文章信息
- 孙一鑫, 裴正存, 詹思延.
- Sun Yixin, Pei Zhengcun, Zhan Siyan.
- 呼吸系统疾病专病队列研究的标准制定与数据共享
- Data harmonization and sharing in study cohorts of respiratory diseases
- 中华流行病学杂志, 2018, 39(2): 233-239
- Chinese Journal of Epidemiology, 2018, 39(2): 233-239
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2018.02.019
-
文章历史
收稿日期: 2017-07-10
慢性呼吸系统疾病是严重危害我国居民健康的疾病之一,已经与心脑血管病、癌症并列成为我国居民的主要死因[1],导致巨大的经济负担与疾病负担。其中,COPD、哮喘、间质性肺疾病和肺血栓栓塞症是常见的重大呼吸疾病,其发病机制、病因、个体化防治策略等尚不明确,亟需建立呼吸专病队列开展研究。
队列研究是目前国际上公认的研究暴露因素与疾病结局之间关联的流行病学研究方法,是探索现代医学问题的重要研究手段。目前,研究者倾向于开展大型人群队列研究,大规模人群调查虽然有利于提升结果的可靠性,但却需要耗费大量人力、物力及时间成本,在探索特殊问题上也存在局限性,因此,对现有多个小样本数据整合为大样本研究的需求日益突出[2]。充分利用现有研究资源,在对多源异构数据进行整合和规范的基础上,进一步开展科学研究,以获得准确可靠、外推性强的研究结果就显得很有必要。
在对多个项目资源之间进行数据整合与共享方面,国际上已经进行了多次尝试,但国内尚缺乏成功经验与模式。数据整合与共享方式按照时序性可分为回顾性、前瞻性与双向整合3种。①回顾性整合是在项目数据收集已经完成的情况下,开展的数据整合与共享工作,例如美国Fred Hutchinson癌症研究中心的COMPASS部门,针对多个癌症数据库开展的数据整合工作[3],由于不同研究对于变量的定义、分类等方面不一致,因此这种数据整合方式最关键的步骤就是评估多源异构数据的相容性与一致性,从而识别、确定标准的通用数据元素,再将各项目数据分别进行映射与转化,形成最小数据集[4]。②前瞻性整合是指在项目开始之初就有数据共享的意愿,因此针对某个特定领域定义最小数据集,使不同项目以相同的定义和标准来收集核心数据[4],国际上开展的前瞻性多中心合作项目大多采用的是这种方式。③双向整合结合了前两者的优点,适用于数据收集已经开始但尚未完成的情况,基于现有资源建立标准化数据模型,一方面对已收集的数据进行映射与转化,充分利用已有数据资源;另一方面在未来可采用统一标准收集数据,为数据整合扫除障碍,节约研究成本,更有利于多源数据的共享。
本文主要介绍的“呼吸系统疾病专病队列研究”已获国家科技部立项支持(2016YFC0901100),项目基于我国5个较为完备的大型社区队列和4个已有良好基础的呼吸疾病临床队列,将系统整合现有队列资源,建立呼吸系统疾病专病队列(呼吸专病队列),项目分课题“呼吸疾病专病队列标准化与质控的方法学研究”(2016YFC0901105)将在回顾性整合数据的同时,针对研究队列的多源异构现状,前瞻性确定最小数据集,建立呼吸系统疾病通用数据模型与各专病队列特异数据模型,以统一的标准收集数据,并在此基础上开展长期随访,实现各队列资源的数据统一与信息共享。呼吸专病队列项目有助于充分挖掘我国现有数据的科研价值,为今后多源异构的大规模队列数据整合与利用提供模式和方法上的重要参考,现将其数据标准制定的思路与阶段性成果介绍如下。
资料与方法针对呼吸系统疾病领域建立通用数据标准的步骤:首先,学习与参考国际标准,其中临床数据交换标准协会(Clinical Data Interchange Standards Consortium,CDISC)的clinical data acquisition standards harmonization(CDASH)模型,与观察性医疗结果合作组织(Observational Medical Outcomes Partnership,OMOP)的通用数据模型(common data model,CDM),对于呼吸专病队列研究的标准制定最具有借鉴意义;其次,整理、归类现有呼吸专病队列资源,评估各队列资源与国际标准的匹配与差异情况;最终建立呼吸专病队列数据标准。标准制定的思路与技术路线见图 1。
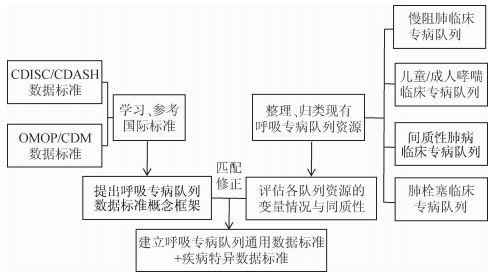
|
| 图 1 呼吸专病队列数据标准制定思路与技术路线图 |
1.学习、参考国际标准:在数据标准化和整合方面,国际上已有组织提出了针对不同领域的标准数据集,以及通用数据模型的概念,目前最受关注的是CDISC[5]针对临床试验提出的CDASH模型和OMOP[6-7]针对观察性数据定义的CDM。二者在其应用领域均制定了适用的数据标准,提出了标准化调查模块与调查条目,并对条目进行明确的定义与说明,对本项目数据标准制定有极高的参考价值,现进行简要介绍。
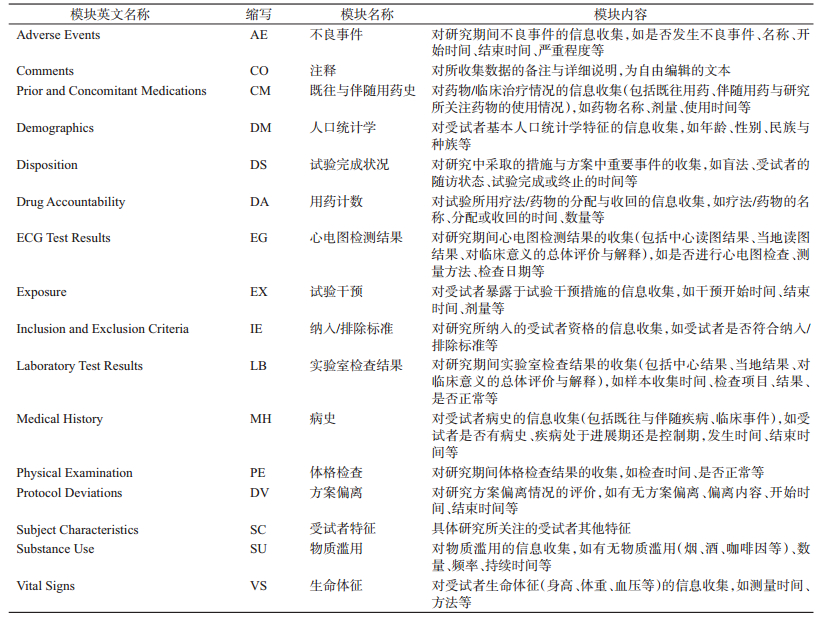
(1)CDISC/CDASH标准:CDISC是一个开放的、多学科参加的非盈利性组织,其针对临床试验的各个环节,建立了一套涵盖试验方案设计、操作,数据收集、分析、提交等方面的完整ODM(operational data model)数据标准[8]。CDASH模型作为该标准中的一部分,主要用于临床试验的数据收集,有助于建立标准的病例报告表(case report form,CRF),从而支持临床研究从数据收集到数据提交的标准化[9]。
CDASH模型[10]包括10个主要模块(Domain),涵盖受试者基线特征、干预、事件等多个方面,定义及设定模块的目的是将CRF中的问题或研究变量归类,统一制定适用的数据标准,具体介绍见表 1。
(2)OMOP/CDM标准:OMOP组织一直致力于合理利用多来源的观察性电子医疗数据库,开展医疗产品的安全性与有效性研究[11]。由于多源数据往往结构各异,若电子医疗数据的结构化程度高则更有利于数据的提取与使用,因此,OMOP组织创建了一套统一的框架,即CDM,来标准化观察性数据的格式,支持多源异构的观察性医疗数据通过提取、转化与加载(extract transform load,ETL)来形成标准化数据仓库,从而进行数据的分析与利用[12]。多中心合作研究可以通过使用所定义的CDM,从不同的数据中心获取研究所需的数据,实现数据共享[13]。
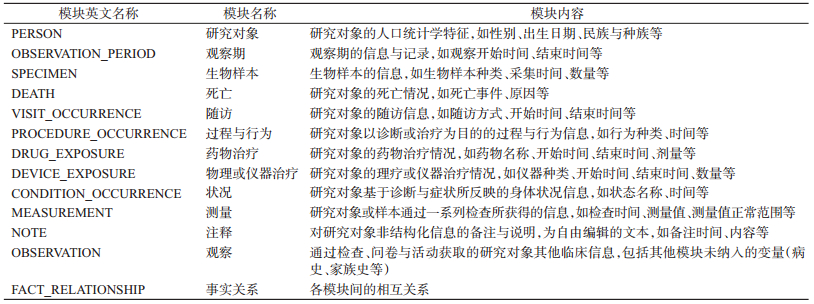
CDM目前已经发展到第五版[14],包括标准化术语表、标准化临床数据表、标准化健康系统数据表、标准化卫生经济数据表等6大模块,见图 2。
其中,标准化临床数据模块(standardized clinical data table)对临床数据进行了归纳整理,并按数据的特点将具体的问题就研究变量归类至不同的表格中,即为不同的数据模块,主要内容见表 2。
2.整理、归类现有呼吸专病队列资源:本研究依托于4个已有良好基础的呼吸疾病临床队列:COPD专病队列、儿童/成人哮喘专病队列、间质性肺疾病专病队列和肺栓塞专病队列,将各队列现有的数据资源及CRF进行初步整理、归纳,评估现有队列资源的变量模块等基本情况,以及与国际标准的匹配与差异情况,从而评估各队列资源间的同质性与整合的可能性。
3.建立呼吸专病队列数据标准:结合前述国际数据标准的模块与适用条件,以及各专病队列的数据特征及匹配情况,针对呼吸疾病领域定义最小数据集,建立呼吸专病队列数据标准,包括呼吸队列通用数据标准和疾病特异数据标准两部分。
结果1.现有呼吸专病队列资源基本情况:对于COPD、哮喘、间质性肺疾病和肺栓塞临床专病队列,整理、归纳各队列现有的数据资源及CRF,统计其变量模块的基本情况见表 3。经评估可以发现,各队列资源的变量模块同质性较好,基本结构相似,具有数据整合的可行性。
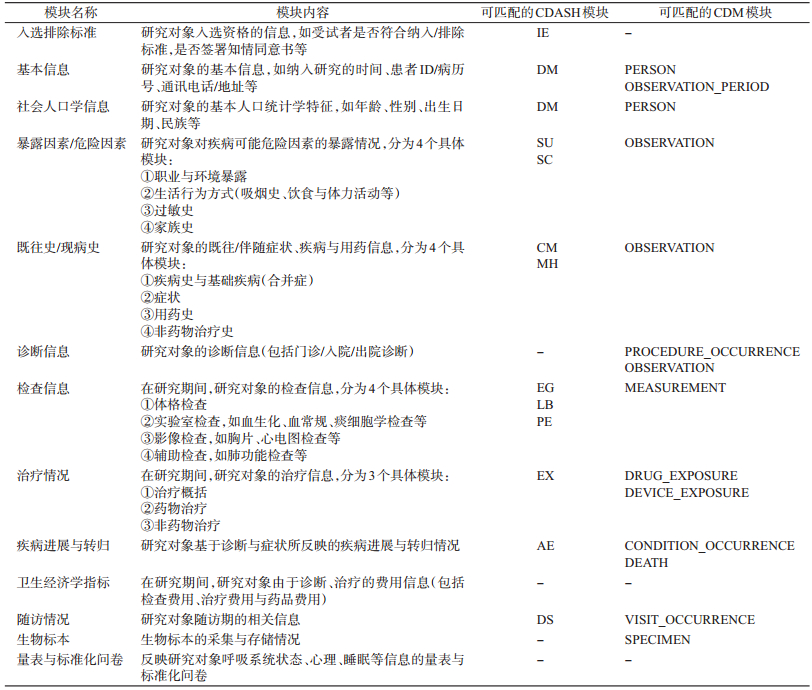
2.呼吸专病队列数据标准:参考国际标准与各专病队列现有变量基本情况,经呼吸疾病临床专家、流行病学专家讨论,本研究提出呼吸专病队列数据标准概念框架,由呼吸队列通用数据标准及疾病特异数据标准构成。其中,通用数据标准针对各专病队列中均有涉及、且能够统一标准的问题或研究变量,如基本信息、社会人口学信息、危险因素等;疾病特异数据标准是针对各专病特有的问题或研究变量所设定的数据标准,如间质性肺疾病涉及的多个特异性检查、肺栓塞涉及的抗凝治疗情况调查等。
(1)呼吸队列通用数据标准:通过分析各专病队列的数据特征,同时匹配国际标准已有模块,研究组已初步拟定包含“基本信息”、“社会人口学信息”、“暴露因素/危险因素”等13个模块的变量表单,以此构成呼吸队列通用数据标准,各专病队列的CRF设计及优化应参照本标准,以实现数据标准统一及数据共享。通用数据标准具体内容见表 4。
(2)疾病特异数据标准:各专病队列特有的、无法列为通用数据标准的问题及研究变量,亦按照通用数据标准制定的流程,将特有数据匹配至国际标准模块或重新定义成适用于本项目的模块,最终汇总形成疾病特异数据标准。
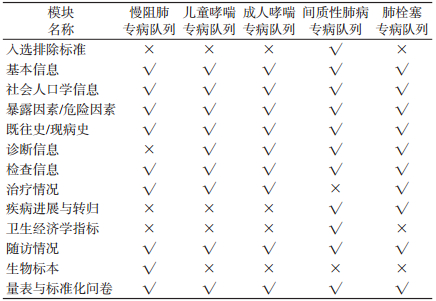
(3)各专病队列变量模块与通用数据标准的匹配情况:将各专病队列的变量模块与构建的通用数据标准进行匹配,见表 5。结果显示匹配程度良好,说明研究构建的通用数据标准适用于以上4个呼吸专病队列,研究组将继续细化通用数据标准的变量,并使各专病队列参照本标准进行CRF设计与优化,在未来以相同的标准与定义收集数据,开展随访。
目前,国际上开展了多项多中心参与的队列资源整合与共享项目,例如BioSHaRE项目(Biobank Standardization and Harmonization for Research Excellence in the European Union),该项目基于8项欧洲人群研究,旨在为欧洲现有队列及生物样本库建立数据整合工具与标准化系统[15];再例如德国开展的LCPD项目(Lung Cancer Phenotype Database),确定肺癌通用数据集,进行数据整合,从而建立关于肺癌的中心数据库[16]等。多源数据的整合能够获得大样本量,提高研究结果的外推性,多中心合作的开展与现有数据资源的高效利用,具有单项研究不可比拟的优势[15]。但是,大量数据的整合与管理过程也存在很多问题与挑战,其中最关键的在于多源数据由于设计、收集等方面的不同导致的异质性问题[17-18]。因此,在项目开展之前就建立标准化数据模型,在变量定义、数据收集、平台搭建等多个方面均考虑后期数据共享的需要,这种前瞻性的数据整合方式更有意义。
本研究建立呼吸专病队列数据标准,所参考的现有数据标准为CDISC/CDASH模型与OMOP/CDM模型。CDASH模型是针对临床试验所建立的标准,定义了数据收集阶段应包括的基本模块[10],参考该标准有助于本项目建立标准的CRF。但是本项目为观察性队列研究而非临床试验研究,与该标准存在一定程度的偏差与不适用性,因此不能直接运用该标准。OMOP的CDM模型虽然是针对观察性数据库所建立的标准,但其关注的更多是药品、疫苗等医疗产品上市后的安全性与有效性研究[11],与本项目临床队列研究也不完全匹配。
项目所建立的呼吸专病队列数据标准特点:首先,针对COPD、哮喘、间质性肺疾病和肺栓塞的疾病特征与临床诊治规律,确定了呼吸系统疾病研究所需的特异性变量模块。为挖掘呼吸疾病相关信息,探究暴露与疾病的发生、发展关系,以及开展精准化诊治研究奠定坚实的数据基础。第二,针对队列研究的特点,确定了无人为干预及随机分组的观察性研究所需的变量模块。有利于观察疾病的自然进程与演变规律,为后期观察性研究开展分析奠定基础。第三,针对本项目双向性研究的特征,确定了与现有队列资源和未来研究所需数据均匹配良好的变量模块。既能充分利用现有队列数据,进行映射分析,又能以统一标准随访收集数据,整合与共享不同队列之间的数据,发挥数据的巨大作用。国际上缺乏特异性针对呼吸系统疾病队列研究建立的数据标准,因此,本研究所建立的通用数据标准具有一定意义。
本项呼吸疾病专病队列研究,在充分利用现有队列资源的同时,针对呼吸系统疾病领域建立通用数据标准,下一步,研究组将结合社区队列的变量情况,进一步细化、优化通用数据标准,使不同专病队列未来可以采用相同的定义和标准来收集核心数据,为开展数据共享,建立中国呼吸系统疾病专病队列中央数据库奠定基础;同时也为多地区、多中心数据资源的整合和利用提供重要参考。
利益冲突: 无
| [1] |
顾景范. 《中国居民营养与慢性病状况报告(2015)》解读[J]. 营养学报, 2016(6): 525–529.
Gu JF. 2015 report on Chinese nutrition and chronic disease[J]. J Nutri, 2016(6): 525–529. DOI:10.3969/j.issn.0512-7955.2016.06.005 |
| [2] | Griffith LE, van den Heuvel E, Raina P, et al. Comparison of standardization methods for the harmonization of phenotype data:an application to cognitive measures[J]. Am J Epidemiol, 2016, 184(10): 770–778. DOI:10.1093/aje/kww098 |
| [3] | Rolland B, Reid S, Stelling D, et al. Toward rigorous data harmonization in cancer epidemiology research:one approach[J]. Am J Epidemiol, 2015, 182(12): 1033–1038. DOI:10.1093/aje/kwv133 |
| [4] |
周光迪, 吴美琴, 赵丽, 等. 中国和加拿大合作出生队列研究数据统一及共享方法[J]. 中国医药生物技术, 2015, 10(6): 494–497.
Zhou GD, Wu MQ, Zhao L, et al. Data harmonization and sharing of birth cohort research cooperated between China and Canada[J]. Chin Med Biotechnol, 2015, 10(6): 494–497. DOI:10.3969/j.issn.1673-713X.2015.06.004 |
| [5] | Clinical Data Interchange Standards Consortium (CDISC)[EB/OL]. (2017-01-01)[2017-06-10]. http://www.cdisc.org. |
| [6] | Observational Medical Outcomes Partnership(OMOP)[EB/OL]. (2017-01-01)[2017-06-10]. http://omop.org/. |
| [7] | OMOP. About Us[EB/OL]. (2017-01-01)[2017-06-10]. http://omop.org/node/22. |
| [8] | Hume S, Aerts J, Sarnikar S, et al. Current applications and future directions for the CDISC Operational Data Model standard:A methodological review[J]. J Biomed Inform, 2016, 60: 352–362. DOI:10.1016/j.jbi.2016.02.016 |
| [9] |
黎燕兰, 胡镜清, 刘保延. CDISC标准与中医药临床研究数据标准化[J]. 世界科学技术-中医药现代化, 2012, 14(6): 2109–2114.
Li YL, Hu JQ, Liu BY. CDISC standards and clinical research data standardization of traditional Chinese medicine[J]. World Sci Technol Modernizat Tradit Chin Med, 2012, 14(6): 2109–2114. DOI:10.3969/j.issn.1674-3849.2012.06.001 |
| [10] | Clinical Data Acquisition Standards Harmonization (CDASH version 1. 1)[EB/OL]. (2017-01-01)[2017-06-10]. https://www.cdisc.org/standards/foundational/cdash. |
| [11] | Trifirò G, Coloma PM, Rijnbeek PR, et al. Combining multiple healthcare databases for postmarketing drug and vaccine safety surveillance:why and how?[J]. J Intern Med, 2014, 275(6): 551–561. DOI:10.1111/joim.12159 |
| [12] | Rijnbeek PR. Converting to a common data model:what is lost in translation?:Commentary on "fidelity assessment of a clinical practice research datalink conversion to the OMOP common data model"[J]. Drug Saf, 2014, 37(11): 893–896. DOI:10.1007/s40264-014-0221-4 |
| [13] |
王玲. 美国观察医疗结果合作项目中数据组织及通用数据模型的应用研究[J]. 中国药物警戒, 2015, 12(6): 341–346.
Wang L. Study on application of data organization and common data model of the observational medical outcomes partnership in US[J]. Chin J Pharm, 2015, 12(6): 341–346. |
| [14] | OMOP Common Data Model Specifications Version 5. 0[EB/OL]. (2017-01-01)[2017-06-10]. http://omop.org/CDM. |
| [15] | Doiron D, Burton P, Marcon Y, et al. Data harmonization and federated analysis of population-based studies:the BioSHaRE project[J]. Emerg Themes Epidemiol, 2013, 10(1): 12. DOI:10.1186/1742-7622-10-12 |
| [16] | Firnkorn D, Ganzinger M, Muley T, et al. A generic data harmonization process for cross-linked research and network interaction. Construction and application for the lung cancer phenotype database of the German center for lung research[J]. Methods Inf Med, 2015, 54(5): 455–460. DOI:10.3414/ME14-02-0030 |
| [17] | Fortier I, Doiron D, Little J, et al. Is rigorous retrospective harmonization possible? Application of the DataSHaPER approach across 53 large studies[J]. Int J Epidemiol, 2011, 40(5): 1314–1328. DOI:10.1093/ije/dyr106 |
| [18] | Bennett SN, Caporaso N, Fitzpatrick AL, et al. Phenotype harmonization and cross-study collaboration in GWAS consortia:the GENEVA experience[J]. Genet Epidemiol, 2011, 35(3): 159–173. DOI:10.1002/gepi.20564 |
 2018, Vol. 39
2018, Vol. 39