文章信息
- 刘志国, 王妙, 刘日宏, 尚修建, 崔步云.
- Liu Zhiguo, Wang Miao, Liu Rihong, Shang Xiujian, Cui Buyun.
- 内蒙古自治区乌兰察布市羊种布鲁氏菌HOOF基因分型特征
- HOOF genotyping characteristics of Brucella melitensis strains isolated in Ulanqab of Inner Mongolia Autonomous Region
- 中华流行病学杂志, 2017, 38(7): 954-958
- Chinese journal of Epidemiology, 2017, 38(7): 954-958
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.07.021
-
文章历史
收稿日期: 2016-11-29
2. 830001 乌鲁木齐, 新疆维吾尔自治区疾病预防控制中心布鲁氏菌病防治科;
3. 102206 北京, 中国疾病预防控制中心传染病预防控制所布鲁氏菌病室 传染病预防控制国家重点实验室布鲁氏菌病组
2. Department of Brucellosis, Xinjiang Uygur Autonomous Region Center for Diseases Control and Prevention, Urumqi 830001, China;
3. Department of Brucellosis, National Institute of Communicable Diseases Prevention and Control, Chinese Center for Disease Prevention and Control, State Key Laboratory for Communicable Disease Prevention and Control, Beijing 102206, China
布鲁氏菌病(布病)是由布鲁氏菌属细菌引起的一种重要的人兽共患传染病[1]。人因直接或间接接触染疫动物及其产品被感染[2]。内蒙古自治区是布病历史疫区,人畜间布病时有发生[3]。但该地区布鲁氏菌分子分型特征研究鲜见报道。HOOF-Prints分型方法不仅分辨高、重复性好且具有高通量、省时和成本低的特点,已用于布鲁氏菌分子流行病学调查。为此本研究应用该方法对临床分离的83株布鲁氏菌进行基因分型,以阐述菌株的分子分型特征。
材料与方法1.菌株:83株布鲁氏菌于2012-2015年分离自内蒙古自治区乌兰察布市10个旗(县、市、区)以及周边邻近地区。其中四子王旗1株,化德县3株,察哈尔右翼中旗3株,卓资县3株,兴和县4株,未知地点4株(乌兰察布),察哈尔右翼后旗5株,商都县6株,察哈尔右翼前旗(前旗)14株,丰镇县16株,凉城县20株以及锡林郭勒盟(锡盟)1株,河北省尚义县3株。按标准方法鉴定菌株的生物型。试验标准参考菌株羊种菌16M、牛种菌544、猪种菌1330(均由中国CDC传染病预防控制所布病室提供)为对照菌株。
2.仪器与试剂:仪器主要包括ClassⅡ A2型生物安全柜、CO2培养箱[赛默飞世尔科技(中国)有限公司]、Gene Amp PCR仪(美国Applied Bio Systems Inc.),DYY-6C-电泳仪(北京六一生物科技有限公司),Gel Doc XR自动凝胶成像系统(美国Bio-Rad公司)。试剂包括布氏琼脂和肉汤培养基[赛默飞世尔科技(中国)有限公司];布鲁氏菌阳性血清,A、M因子血清,Tb、Wb、BK2噬菌体(均由中国CDC传染病预防控制所布病室提供);2×EasyTaq PCR Super mix、DNA Marker 2000(TranS 2K)、6×Loading Buffer均由北京全氏金生物技术有限公司提供;细菌核酸提取试剂盒购自德国Qiagen公司。HOOF-PCR引物名称、序列见表 1;羊种,牛种1、2、4型,猪种1型和绵羊附睾种布鲁氏菌鉴别方法(AMOS-PCR)引物参照文献[4]。引物均由北京擎科新业生物技术有限公司合成。
3.试验方法:
(1)菌株基因组DNA提取:将传代培养布鲁氏菌灭活,并严格按照QIAamp DNA Mini Kit(250次)(细菌核酸提取试剂盒)操作步骤进行,提取完毕后用紫外分光光度计测定DNA浓度及纯度,-20 ℃保存备用。
(2)AMOS-PCR扩增:扩增条件、参数以及电泳结果检测同文献[5]。
(3)HOOF-PCR扩增及数据分析:扩增采用20 μl体系,包括2×EasyTaq PCR Super mix 18.2 μl、上下游引物各0.4 μl(10 μmol/L)、DNA 1.0 μl。扩增参数为预变性95 ℃ 3 min,95 ℃ 30 s;56 ℃ 30 s;72 ℃ 50 s;终末延伸72 ℃ 3 min。PCR产物经2.0%琼脂糖凝检测。获得预期条带的扩增结果送北京擎科新业生物技术有限公司进行微卫星扫描(STR),并与GenBank公布的布鲁氏菌HOOF分型标准等位基因比对,将扩增片段转换为串联重复数(U)。
4.数据处理:用Hunter-Gaston分辨指数(HGDI)评估各VNTR位点的多态性和数目可变串联重复序列(VNTR)基因分型方法对菌株的综合辨别能力(http://www.hpa-bioinformatics.org.uk/cgi-bin/DICI/DICI.pl)。用BioNumerics v5.0软件采用UPGMA(unweighted pair group method using arithmetic averages)进行聚类分析,获得布鲁氏菌的聚类图。
结果1.菌株鉴定:83株布鲁氏菌(待测菌株)CO2需要和H2S产生试验结果均阴性;血清凝集试验均阳性,其中67株A、M因子血清凝集均阳性,其余16株仅M因子血清凝集阳性;染料抑菌试验均为阳性;试验菌株均可被噬菌体BK2裂解(表 2)。
2. AMOS-PCR扩增:阳性对照牛、羊、猪标准参考菌株均获得了预期结果,扩增条带分别为牛种498 bp、羊种731 bp和猪种285 bp;阴性对照未见扩增。待测菌株的产物均为731 bp(图 1)。

|
| 注:M:Marker;1~3分别为牛、羊、猪标准参考菌株对照;4为阴性对照;5~23为羊种布鲁氏菌 图 1 布鲁氏菌的AMOS-PCR扩增结果 |
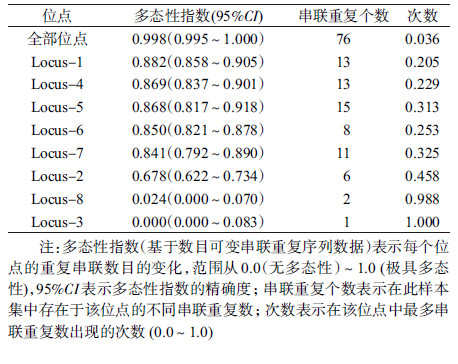
3.分型位点的多态性:8个位点的多态性指数为0.998(95%CI:0.995~1.000),其分辨力由高到低依次为Locus-1(0.882)、Locus-4(0.869)、Locus-5(0.868)、Locus-6(0.850)、Locus-7(0.841)、Locus-2(0.678)、Locus-8(0.024)、Locus-3(0.000),其中6个位点(Locus-1、2、4~7)的多态性指数均≥0.678,Locus-8的多态性指数为0.024,仅Locus-3的多态性指数为0.000(无多态性);6个高分辨力位点的等位基因型依次分别为13、13、15、8、11和6(表 3)。
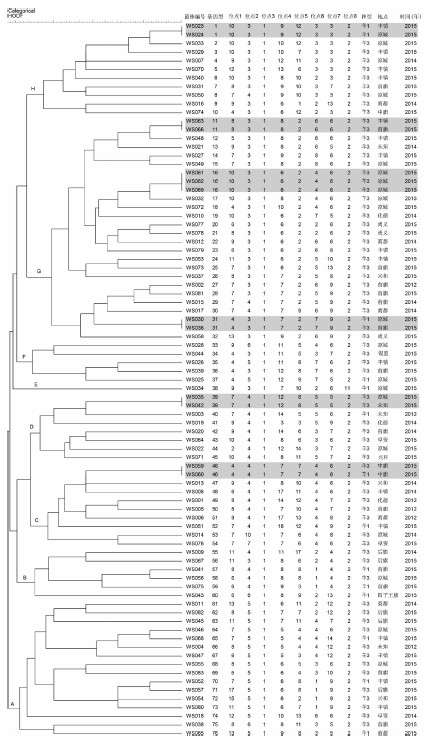
4.聚类分析:83株布鲁氏菌聚为8类(依次命名为A~H)76个基因型;其中6个为共享基因型,70个为独特基因型。在6个共享基因型中,有2个基因型(16和46)分别包含3和2株分离时间地点相同的菌株;其余4个基因型分别包括2株来自不同地区的菌株(图 2)。
|
| 注:标注阴影为共享基因型 图 2 83株羊种布鲁氏菌HOOF分型聚类结果 |
本研究分离的83株布鲁氏菌中16株为羊种1型菌,67株为羊种3型菌。常规鉴定和AMOS-PCR鉴定结果相同,均为羊种布鲁氏菌,其种型与我国西北地区其他省份的种型一致[6-8]。90%以上患者与羊有直接或间接的接触史,证实了疫羊是该地区人间布病的主要传染源。与疫羊直接或间接接触也被其他研究证实是人感染布病最重要的方式[9-10]。因此,强化疫羊的管控是控制人间布病最有效的措施。
布鲁氏菌的核心基因组序列有极高的同源性(核苷酸相似性>99%),但本实验所用8个位点的多态性指数为0.998,表明该方法对试验菌株有极高的分辨力,是一种适用的布鲁氏菌分型技术。在8个多态性位点中有6个多态性位点呈现出极好的分辨力,而Locus-3的多态性指数为0.000,无分辨能力。对该地区菌株的分辨力主要来自6个多态性指数较高的位点。多态性指数的相似性或差异性可用于评估临床分离株的遗传相关性。分离自乌兰察布市周边的锡盟及河北省尚义县菌株,虽未见与乌兰察布市分离株形成共享基因型,但仅在1~3个高变位点存在差异,提示这些菌株来自同一克隆群,具有较近的亲缘关系。HOOF分型虽是一种分辨力较高的基因分型方法,但数据分析和结果解读均有难度,在一定程度上限制了其在基层的推广应用。
本文83株羊种布鲁氏菌聚为8类76个基因型,包括6个共享基因型和70个独特基因型。70株布鲁氏菌呈现独特的基因型提示该地区布病具有零星和流行病学无关联的特点;6个共享基因型包括13株菌表明菌株存在流行病学关联。从分子病原学角度证实该地区布病具有零星散发和暴发共存的发病特点,但以零星散发为主,偶有暴发。本研究结果与该地区疫情特点基本一致[5]。先前的研究证实流行病学相关的菌株具有相同或相似的基因型[11-12]。在6个共享基因型中,有4个基因型(1、11、31和39)每个包括2株分离时间相同而地点相邻的菌株;结合患者信息,凉城县与前旗、丰镇县接壤相邻地区多有患病家畜流动的交叉感染病例,这与流行病学调查结果相吻合。提示交叉感染病例是由该地区缺乏流通检疫和管控受染疫动物感染所致。另2个共享基因型(16、46)包括5株布鲁氏菌,分别来自相同的时间地点,证实了引起多点暴发。乌兰察布市及周边地区人畜间布病较为严重,患病家畜无序转运和跨区域流动极为频繁。本研究结果证实了当前该区域布病的流行特点,为人畜间布病防控提供了有意义的流行病学线索。
利益冲突: 无
| [1] | van Straten M, Bardenstein S, Keningswald G, et al. Brucella abortus S19 vaccine protects dairy cattle against natural infection with Brucella melitensis[J]. Vaccine, 2016, 34(48): 5837–5839. DOI:10.1016/j.vaccine.2016.10.011 |
| [2] | Shevtsov A, Ramanculov E, Shevtsova E, et al. Genetic diversity of Brucella abortus and Brucella melitensis in Kazakhstan using MLVA-16[J]. Infect Genet Evol, 2015, 34: 173–180. DOI:10.1016/j.meegid.2015.07.008 |
| [3] | Chen YF, Ke YH, Wang YF, et al. Changes of predominant species/biovars and sequence types of Brucella isolates, Inner Mongolia, China[J]. BMC Infect Dis, 2013, 13: 514. DOI:10.1186/1471-2334-13-514 |
| [4] | Shome R, Krithiga N, Shankaranarayana PB, et al. Genotyping of Indian antigenic, vaccine, and field Brucella spp. using multilocus sequence typing[J]. J Infect Dev Ctries, 2016, 10(3): 237–244. DOI:10.3855/jidc.6617 |
| [5] |
刘志国, 王妙, 刘日宏, 等.
内蒙古乌兰察布布氏菌分离株种型鉴定及流行病学特征分析[J]. 中国人兽共患病学报, 2016, 32(7): 618–622, 631.
Liu ZG, Wang M, Liu RH, et al. Species identification and epidemiological characteristics analysis of Brucella in Ulanqab, Inner Mongolia, China[J]. Chin J Zoonoses, 2016, 32(7): 618–622, 631. DOI:10.3969/j.issn.1002-2694.2016.07.006 |
| [6] | Xiao P, Yang HX, Di DD, et al. Genotyping of human Brucella melitensis biovar 3 isolated from Shanxi province in China by MLVA16 and HOOF[J]. PLoS One, 2015, 10(1): e0115932. DOI:10.1371/journal.pone.0115932 |
| [7] | Sun MJ, Di DD, Li Y, et al. Genotyping of Brucella melitensis and Brucella abortus strains currently circulating in Xinjiang, China[J]. Infect Genet Evol, 2016, 44: 522–529. DOI:10.1016/j.meegid.2016.07.025 |
| [8] | Jiang H, Fan MG, Chen JD, et al. MLVA genotyping of Chinese human Brucella melitensis biovar 1, 2 and 3 isolates[J]. BMC Microbiol, 2011, 11: 256. DOI:10.1186/1471-2180-11-256 |
| [9] | Tan ZM, Huang Y, Liu GY, et al. A familial cluster of human brucellosis attributable to contact with imported infected goats in Shuyang, Jiangsu province, China, 2013[J]. Am J Trop Med Hyg, 2015, 93(4): 757–760. DOI:10.4269/ajtmh.15-0149 |
| [10] | Roushan MRH, Ebrahimpour S. Human brucellosis:an overview[J]. Caspian J Intern Med, 2015, 6(1): 46–47. DOI:10.1016/S1201-9712(03)90049-X |
| [11] | KılıçS, Ivanov IN, Durmaz R, et al. Multiple-locus variable-number tandem-repeat analysis genotyping of human Brucella isolates from Turkey[J]. J Clin Microbiol, 2011, 49(9): 3276–3283. DOI:10.1128/JCM.02538-10 |
| [12] | Allen A, Breadon E, Byrne A, et al. Molecular epidemiology of Brucella abortus in Northern Ireland-1991 to 2012[J]. PLoS One, 2015, 10(9): e0136721. DOI:10.1371/journal.pone.0136721 |
 2017, Vol. 38
2017, Vol. 38