文章信息
- 龙金照, 徐亚珂, 段广才, 梁文娟, 刘慧莹, 陈帅印, 郗园林, 王鹏飞, 王颖芳.
- Long Jinzhao, Xu Yake, Duan Guangcai, Liang Wenjuan, Liu Huiying, Chen Shuaiyin, Xi Yuanlin, Wang Pengfei, Wang Yingfang.
- O26: H11及NM大肠埃希菌CRISPR的分子分布特征及其与stx噬菌体的关系
- Association between phage-mediated shiga toxin and molecular distribution of CRISPR in Escherichia coli O26: H11 or NM
- 中华流行病学杂志, 2017, 38(7): 944-949
- Chinese journal of Epidemiology, 2017, 38(7): 944-949
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.07.019
-
文章历史
收稿日期: 2016-12-21
2. 453003 新乡医学院分子诊断与医学检验技术河南省协同创新中心;
3. 471023 洛阳, 河南科技大学校医院;
4. 471023 洛阳, 预防医学教研室
2. Henan Innovation Center of Molecular Diagnosis and Laboratory Medicine, Xinxiang Medical University, Xinxiang 453003, China;
3. University Health Service, Henan University of Science and Technology, Luoyang 471023, China;
4. Department of Preventive Medicine, Henan University of Science and Technology, Luoyang 471023, China
成簇规律间隔短回文重复序列(Clustered Regularly Interspaced Short Palindromic Repeat,CRISPR)是一类在原核生物中发现的能够抵御外来遗传物质入侵的一种特殊结构,由正向重复序列(Direct Repeats,DR)和插入其中的间隔序列(Spacer)构成[1]。间隔序列作为核心元件,具有高度多态性,可反映外来遗传物质的入侵过程,这种特征使其成为监测新发高毒高致病性病原菌的有效分子靶标[2]。O26: H11或NM血清型是产志贺毒素大肠埃希菌中一种非常重要的血清型,可引起水样腹泻、溶血性尿毒症、出血型结肠炎等疾病[3]。河南省于2005年从1名腹泻患者中分离到1株不产志贺毒素但产超广谱β内酰胺酶的O26血清型大肠埃希菌[4]。本研究对O26: H11或NM血清型大肠埃希菌中CRISPR的分子分布进行分析。
资料与方法1.数据来源:O26: H11及NM血清型大肠埃希菌的全基因组序列及鸟枪法测序序列从NCBI获取。
2.生物信息学工具及方法:利用Serotype Finder(https://cge.cbs.dtu.dk/services/SerotypeFinder/)在线工具对O26: H11及NM血清型大肠埃希菌进行筛选,再用Virulence Finder(https://cge.cbs.dtu.dk/services/VirulenceFinder/)获取菌株的毒力信息,并用IS Finder(https://www-is.biotoul.fr//)在线工具确认插入序列。CRISPR位点信息通过CRT软件或者分析CRISPR位点上下游序列的保守性获得,并用CRISPR Finder软件进行确认。
间隔序列的同源性搜索使用CRISPR Target(http://nhjy.hzau.edu.cn/kech/swxxx/jakj/dianzi/Bioinf4/ miRNA/miRNA1.html)。重复序列的二级结构预测使用RNA fold(http://nhjy.hzau.edu.cn/kech/swxxx/jakj/dianzi/Bioinf4/miRNA/miRNA1.htm)。利用Excel表对间隔序列进行图谱绘制及CRISPR分型。
3.统计学检验:用SPSS 21.0软件进行统计学分析、Mann-Whitney进行两样本总体分布的比较,检验水准α=0.05。
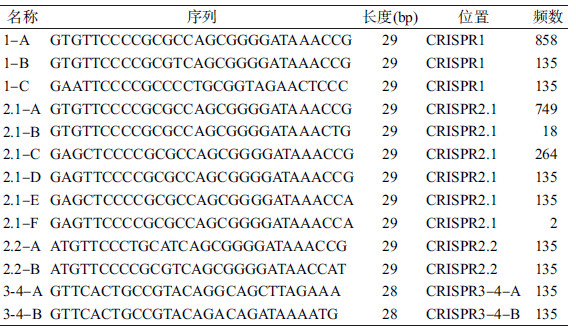
结果1. O26: H11或NM血清型大肠埃希菌CRISPR位点的分布:从NCBI数据库共筛选出135株血清型为O26: H11或NM的大肠埃希菌,共识别出4种CRISPR位点,为Ⅰ-E CRISPR/Cas系统,具体位置见图 1。根据CRISPR位点在基因组中的位置,将其分别命名为CRISPR1、CRISPR2.1、CRISPR2.2、CRISPR3-4,4种CRISPR位点的阳性率均为100%。其中CRISPR1位点中间隔序列数目为3~11个,CRISPR2.1位点中间隔序列数为5~12个,CRISPR2.2中间隔序列数为1个,CRISPR3-4中间隔序列数为1个,其中CRISPR1、CRISPR2.1、CRISPR2.2中重复序列均为29 bp,间隔序列均为32 bp,CRISPR3-4中重复序列为28 bp,间隔序列为32 bp。
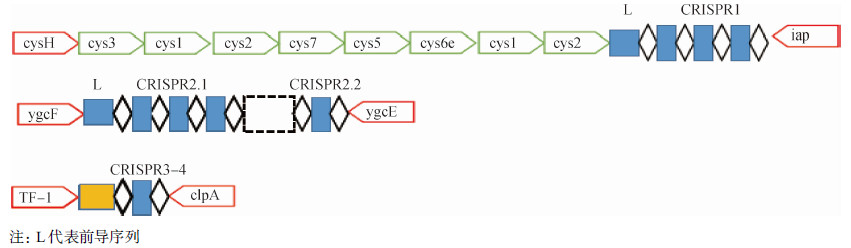
|
| 图 1 O26: H11或NM血清型大肠埃希菌CRISPR位点 |
2.重复序列:CRISPR1位点中共识别出3种重复序列,分别命名为1-A、1-B、1-C。CRISPR2.1中识别出5种重复序列,分别命名为2.1-A、2.1-B、2.1-C、2.1-D、2.1-E、2.1-F。CRISPR2.2中识别出2种重复序列,分别命名为2.2-A、2.2-B。CRISPR3-4中识别出2种重复序列,分别命名为3-4-A、3-4-B。其中CRISPR1中的重复序列1-A与CRISPR2.1中的重复序列2.1-A拥有完全一致的序列,而其他重复序列与这两种重复序列均存在不同程度的碱基差异(表 1)。CRISPR1中的重复序列与CRISPR2.1中的重复序列除1-C外,其余的均能形成稳定的RNA二级结构,其最小自由能(Minimum Free Energy,MFE)均小于-15 kcal/mol(图 2)。
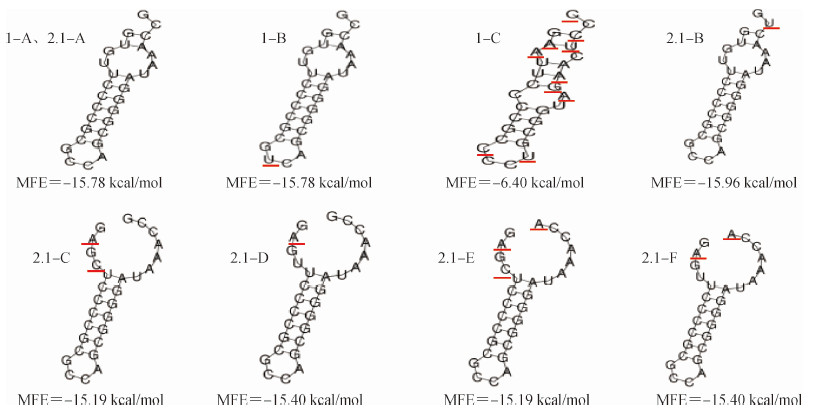
|
| 图 2 O26: H11或NM血清型大肠埃希菌CRISPR1及CRISPR2.1位点重复序列的二级结构预测 |
3.间隔序列:O26: H11或NM血清型大肠埃希菌CRISPR1位点中共识别出10个间隔序列,CRISPR2.1位点中识别出14个间隔序列和1个插入序列(Insertion Sequence,IS),CRISPR2.2中共识别出1个间隔序列,CRISPR3-4中只存在1个间隔序列,未发现独特的间隔序列,同时对间隔序列进行同源性搜索,未发现外源同源性序列(表 2)。
4. O26: H11或NM血清型大肠埃希菌CRISPR中间隔序列的亚型分析:根据CRISPR中间隔序列的排列情况,CRISPR1位点包括19个亚型,CRISPR2.1位点包括22个亚型,CRISPR2.2包括1个亚型,CRISPR3-4包括1个亚型。因CRISPR2.2与CRISPR3-4均只包括一个亚型,且其在所有菌株中都存在,故不作分析,仅分析CRISPR1与CRISPR2.1中的间隔序列。若将CRISPR1与CRISPR2.1的亚型结果结合起来,可将135株大肠埃希菌分为40个亚型,其中以亚型ST-9菌株数目最多,为32株,其次是亚型ST-22,为18株,亚型ST-20为16株,其余亚型菌株数目均小于10株(图 3)。
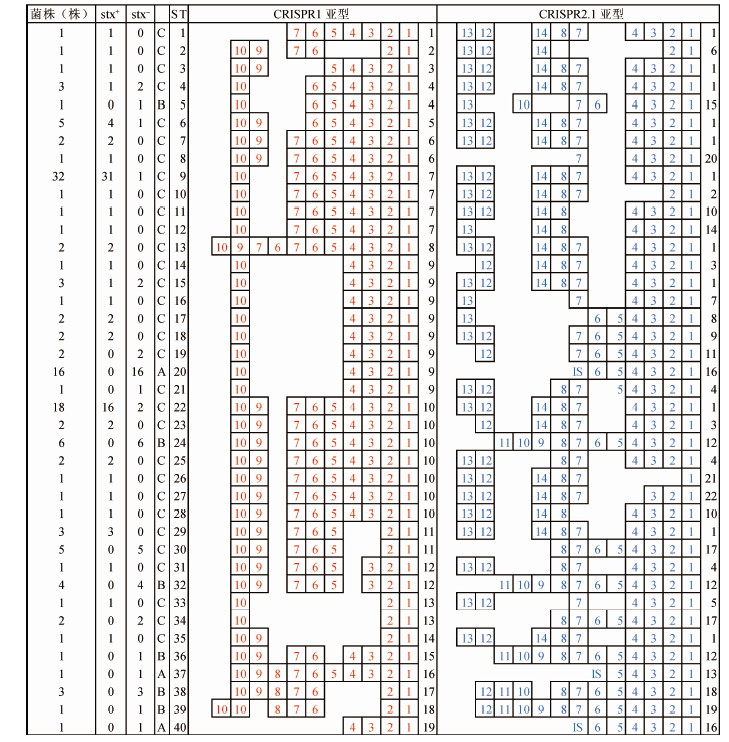
|
| 注:红色数字1~10分别代表CRISPR1中的间隔序列S1-1~S1-10;蓝色数字1~14分别代表CRISPR2.1中的间隔序列S2.1-1~S2.1-14;间隔序列从右至左离前导序列越来越近 图 3 O26: H11或NM血清型大肠埃希菌CRISPR1及CRISPR2.1的分子分布特征 |
在CRISPR2.1位点处出现一个IS序列,经IS Finder在线工具中的BLAST功能比对,结果显示其为ISEc17,属于IS3家族。IS序列只出现在ST-20、ST-37、ST-40这3个CRISPR亚型中,并且这3个亚型CRISPR1中离前导序列最远的4个间隔序列完全一致,CRISPR2.1也是如此,见图 3。
根据CRISPR2.1中间隔序列的排布方式,将菌株进一步分为3个群组,A组中含有插入序列,B组至少含有S2.1-9、S2.1-10、S2.1-11这3个间隔序列中的一个,C组中不含插入序列也不含有B组所描述的3个间隔序列中的任何一个,由S2.1-1~S2.1-8、S2.1-12~S2.1-14这11个间隔序列所构成。
5. O26: H11或NM血清型大肠埃希菌CRISPR群组与stx噬菌体的关系:根据stx噬菌体在O26: H11或NM血清型大肠埃希菌中的存在情况可将其分为2个亚组:第1亚组包含3种类型(亚型1:stx1+、stx2+;亚型2:stx1+、stx2-;亚型3:stx1-、stx2+),均为stx+菌株;第2亚组为stx1-、stx2-,为stx-菌株。135株大肠埃希菌中,83株为stx+菌株,52株为stx-菌株。A组共18株,均为stx-菌株,B组共16株,也均为stx-菌株,而C组中共101株细菌,仅有11株为stx-菌株,其余90株均为stx+菌株。stx+菌株只在CRISPR群组C组中出现,在A组及B组中并未出现stx+菌株,均为stx-菌株,见图 3。
6.在CRISPR群组C组中stx噬菌体与间隔序列数目的关系:经统计学检验,stx+菌株与stx-菌株相比,在CRISPR1、CRISPR2.1、CRISPR1+CRISPR2.1位点上间隔序列数目的分布上差异均有统计学意义(均P<0.05),stx+菌株比stx-菌株拥有更多的间隔序列(表 3)。
CRISPR/Cas系统广泛存在于古生菌和细菌中[5],大肠埃希菌已被研究确认存在4个CRISPR位点,其中CRISPR1、CRISPR2及其临近的Cas基因构成Ⅰ-E型CRISPR/Cas系统,CRISPR3、CRISPR4及其临近的Cas基因共同构成Ⅰ-F型CRISPR/Cas系统[6]。本研究通过生物信息学的方法发现,存在于O26: H11或NM血清型中的大肠埃希菌为Ⅰ-E型CRISPR/Cas系统,其存在4个CRISPR位点,CRISPR1、CRISPR2.1、CRISPR2.2、CRISPR3-4阳性率均为100%。根据间隔序列的排布方式,可获得多个CRISPR亚型,CRISPR1由10个间隔序列组成,CRISPR2.1中由一个插入序列和14个间隔序列组成,但并未发现独特的间隔序列,提示O26: H11或NM血清型大肠埃希菌虽然在CRISPR位点上存在着多样性,但其在间隔序列的获取上仍然具有一定的相似性和保守性。
CRISPR/Cas系统中的间隔序列靶向外源遗传物质,在原核生物中发挥着免疫防御及免疫记忆功能,记录了细菌在进化过程中的外来遗传物质入侵过程[7],在一定程度上可反映菌株间的进化关系,也可作为鉴定和判断强毒株的潜在分子靶标[8]。Delannoy等[9]基于CRISPR2.1位点建立了一种qPCR方法,用于特异性检测O26: H11、O45: H2、O103: H2、O111: H8、O121: H19、O145: H28等肠出血性大肠埃希菌。梁文娟等[10]基于CRISPR1及CRISPR2.1位点建立了一种能够特异性检测O157: H7的PCR方法,其特异度分别可达到100.0%及99.6%。Yin等[11]基于CRISPR位点建立了一种从O55: H7进化到O157: H7的模型,揭示了O55: H7与O157: H7之间的进化关系。本研究收集了NCBI数据库中O26: H11或NM血清型的大肠埃希菌,发现产志贺毒素大肠埃希菌只在CRISPR群组C组中出现,CRISPR群组A组与B组中的大肠埃希菌均为stx-型。分析原因可能在于O26: H11或NM血清型大肠埃希菌在进化过程中,stx噬菌体与CRISPR群组C组中细菌相互作用时,其为温和性噬菌体,可发生溶源现象,stx噬菌体可整合到细菌基因组中,而与CRISPR群组A组及B组中细菌相互作用时,其为烈性噬菌体,不能整合到细菌基因组中,而是使细菌发生裂解。但CRISPR群组C组中仍有18株细菌未检测到stx噬菌体的存在,原因可能在于其在进化过程中未遭遇过stx噬菌体的入侵或者曾经入侵过但在进化过程中丢失了stx噬菌体,但仍保留着获取stx噬菌体的能力。进而进化成高毒的新发高致病性病原菌的潜能,CRISPR群组C组的大肠埃希菌应该作为公共卫生监测的重点对象。
间隔序列作为外源遗传物质入侵后所遗留的产物,其数目多少可间接反映细菌在进化过程中遭遇可移动元件入侵的情况[12]。stx+菌株比stx-菌株拥有更多的间隔序列,提示产志贺毒素的O26: H11或NM血清型大肠埃希菌在进化过程中接受了更多可移动元件的入侵及整合过程。但进行间隔序列同源性搜索时,并未获得同源的外源遗传物质,可能在于现有发现及认知的质粒或噬菌体的局限性及本研究样本量的限制。
利益冲突: 无
| [1] | Díez-Villaseñor C, Almendros C, García-Martínez J, et al. Diversity of CRISPR loci in Escherichia coli[J]. Microbiology, 2010, 156(5): 1351–1361. DOI:10.1099/mic.0.036046-0 |
| [2] | Touchon M, Rocha EPC. The small, slow and specialized CRISPR and anti-CRISPR of Escherichia and Salmonella[J]. PLoS One, 2010, 5(6): e11126. DOI:10.1371/journal.pone.0011126 |
| [3] | Jiang Y, Yin S, Dudley EG, et al. Diversity of CRISPR loci and virulence genes in pathogenic Escherichia coli isolates from various sources[J]. Int J Food Microbiol, 2015, 204: 41–46. DOI:10.1016/j.ijfoodmicro.2015.03.025 |
| [4] |
张濛, 廖兴广, 张锦, 等.
河南省首次从人体内分离出E. coli O26: H11[J]. 中国人兽共患病学报, 2005, 21(2): 108–109, 104.
Zhang M, Liao XG, Zhang J, et al. Isolation of E. coli O26: H11 from human body for the first time in Henan province[J]. Chin J Zoon, 2005, 21(2): 108–109, 104. DOI:10.3969/j.issn.1002-2694.2005.02.004 |
| [5] | Sorek R, Kunin V, Hugenholtz P. CRISPR-a widespread system that provides acquired resistance against phages in bacteria and archaea[J]. Nat Rev Microbiol, 2008, 6(3): 181–186. DOI:10.1038/nrmicro1793 |
| [6] | Touchon M, Charpentier S, Clermont O, et al. CRISPR distribution within the Escherichia coli species is not suggestive of immunity-associated diversifying selection[J]. J Bacteriol, 2011, 193(10): 2460–2467. DOI:10.1128/JB.01307-10 |
| [7] |
薛泽润, 王颖芳, 段广才, 等.
志贺菌中成簇规律间隔短回文重复序列的分子分布特征[J]. 中华流行病学杂志, 2015, 36(8): 875–878.
Xue ZR, Wang YF, Duan GC, et al. Molecular characteristics of clustered regularly interspaced short palindromic repeat in Shigella[J]. Chin J Epidemiol, 2015, 36(8): 875–878. DOI:10.3760/cma.j.issn.0254-6450.2015.08.023 |
| [8] |
梁文娟, 张荣光, 段广才, 等.
基于CRISPR/Cas的大肠埃希菌分子标志物的监测研究[J]. 中华流行病学杂志, 2016, 37(8): 1080–1086.
Liang WJ, Zhang RG, Duan GC, et al. A Surveillance study on CRISPR/C molecular biomarker in Escherichia coli[J]. Chin J Epidemiol, 2016, 37(8): 1080–1086. DOI:10.3760/cma.j.issn.0254-6450.2016.08.005 |
| [9] | Delannoy S, Beutin L, Fach P. Use of clustered regularly interspaced short palindromic repeat sequence polymorphisms for specific detection of enterohemorrhagic Escherichia coli strains of serotypes O26: H11, O45:H2, O103:H2, O111:H8, O121:H19, O145:H28, and O157:H7 by real-time PCR[J]. J Clin Microbiol, 2012, 50(12): 4035–4040. DOI:10.1128/JCM.02097-12 |
| [10] |
梁文娟, 张荣光, 段广才, 等.
基于CRISPR对大肠埃希菌O157:H7的检测[J]. 西安交通大学学报:医学版, 2016, 37(5): 748–753.
Liang WJ, Zhang RG, Duan GC, et al. Detection of Escherichia coli O157:H7 based on CRISPR locus[J]. J Xi'an Jiaotong Univ, 2016, 37(5): 748–753. DOI:10.7652/jdyxb201605027 |
| [11] | Yin S, Jensen MA, Bai JW, et al. The evolutionary divergence of Shiga toxin-producing Escherichia coli is reflected in clustered regularly interspaced short palindromic repeat (CRISPR) spacer composition[J]. Appl Environ Microbiol, 2013, 79(18): 5710–5720. DOI:10.1128/AEM.00950-13 |
| [12] | Almendros C, Mojica FJM, Díez-Villaseñor C, et al. CRISPR-Cas Functional module exchange in Escherichia coli[J]. mBio, 2014, 5(1): e00767–13. DOI:10.1128/mBio.00767-13 |
 2017, Vol. 38
2017, Vol. 38