文章信息
- 谭红丽, 王鹏, 李伟.
- Tan Hongli, Wang Peng, Li Wei.
- 鼠疫噬菌体基因组研究进展
- Advance on genome research of Yersinia pestis bacteriophage
- 中华流行病学杂志, 2017, 38(4): 561-564
- Chinese journal of Epidemiology, 2017, 38(4): 561-564
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.04.030
-
文章历史
收稿日期: 2016-09-23
2. 671000 大理, 云南省地方病防治所云南省自然疫源性疾病防控技术重点实验室;
3. 102206 北京, 中国疾病预防控制中心传染病预防控制所鼠疫室 传染病预防控制国家重点实验室
2. Yunnan Key Laboratory for Disease of Natural Focus Prevention and Control, Yunnan Institute for Endemic Disease Prevention and Control, Dali 671000, China;
3. State Key Laboratory for Infectious Disease Prevention and Control, National Institute for Communicable Disease Control and Prevention, Chinese Center for Disease Control and Prevention, Beijing 102206, China
有其踪迹,反之如果在检材中检出噬菌体,则提示可能有鼠疫菌或曾被鼠疫菌感染。许多研究者从鼠疫者尸体、染疫动物死尸、恢复期患者粪便和血液、动物排泄物、污水以及鼠疫菌培养物中分离出[2]。依据噬菌体感染宿主菌的生物学后果可将其分为溶源噬菌体和裂解噬菌体,不同来源的鼠疫噬菌体在抗原性、颗粒形态、毒力、基因组结构和对鼠疫菌特异程度上有差异,大部分的鼠疫噬菌体在18~22 ℃时对鼠疫菌具有较强的特异性,但在30~37 ℃时还能裂解某些假结核耶尔森菌、大肠埃希菌、志贺痢疾菌和沙门菌。
研究鼠疫噬菌体基因组,有助于认识鼠疫噬菌体的进化及其在宿主细菌-鼠疫菌的共生进化中的作用,了解它在鼠疫菌保存机制中的作用,为研发治疗鼠疫的特效药、对抗鼠疫生物战剂、噬菌体实验室诊断应用以及建立鼠疫噬菌体生物学检测技术提供帮助。现对鼠疫噬菌体基因组最新研究结果,做简要综述。
1.已测序的鼠疫噬菌体及其基本特征:迄今,在美国国立生物信息中心(NCBI)数据库里收录的鼠疫噬菌体共12株(表 1)。在免疫学上被分为4个血清型[2],其中血清型Ⅰ最为常见,以所有已测序的8株烈性鼠疫噬菌体Berlin、ΦA1122、Yepe2、YpP-G、YepΦ、YpP-Y、YpP-R及YpsP-G为代表,均属于T7噬菌体家族,有尾噬菌体目,短尾噬菌体科(Podoviridae),C1形态群。具有同样大小的六边形结构的头部和一个短小非伸缩性圆锥形尾部,全基因组大小很相近,为37.71~39.41 kb,预测基因产物个数也相近,均是双链DNA病毒,GC含量47.10~48.30 mol%,与宿主菌鼠疫菌(47.6 mol%)相近。能裂解74%~100%的鼠疫耶尔森菌,25%~65%的假结核耶尔森菌、部分大肠埃希菌、志贺菌和沙门菌。血清Ⅱ型是P2样噬菌体,属于肌尾噬菌体科(Myoviridae),与常见的肠杆菌噬菌体P1、P2和P4同属一科,包括已测序的L-413C和大多数鼠疫温和噬菌体(或溶原性噬菌体)及噬菌体ΦJA1。血清Ⅲ型目前只有温和噬菌体P。血清Ⅳ型以另2株温和噬菌体Tal和513为代表。
(1)YepΦ:为我国分离的鼠疫诊断用噬菌体[3],在不同温度下均能特异裂解鼠疫菌,而不裂解其他菌。
(2)YepE2和YpP-G:是1929年俄国科学家Pokrovskaya从花金鼠感染组织中分离到特异性作用于鼠疫菌的裂解性噬菌体[4],被认为是特异性比较高的鼠疫诊断噬菌体,能裂解98%~100%的鼠疫耶尔森菌及19%~27%的假结核菌。
(3)ΦA1122:为美国CDC诊断用噬菌体[5],由Advier在1933年从一个腺鼠疫患者血液中分离出,在20 ℃能裂解鼠疫菌及37 ℃裂解假结核耶尔森菌。
(4)YpP-Y和YpP-R:是1964年由Knapp和Zwillenberg[6]发现,能裂解鼠疫噬菌体及部分假结核噬菌体。
(5)PY100:为2007年Schwudke等[7]从德国一个养猪场饲料中分离到,37 ℃裂解鼠疫耶尔森菌、假结核耶尔森菌及小肠结肠炎耶尔森菌。电镜下观察,PY100有一个20面体衣壳(70 nm)的头部和一个可伸缩的尾部(80 nm)。
(6)YpsP-PST:具有一个20面体衣壳的头部(90~120 nm)和一个长的纤维尾部,属肌尾噬菌体科,由双链DNA组成,是假结核菌诊断用噬菌体,也能裂解部分鼠疫菌[8]。
(7)L-413C:分离自溶源的鼠疫毒力菌413(中世纪型),该株菌是1964年从中亚荒漠鼠疫疫源地中的一只红尾沙鼠上分离获得[9],具有很高的特异性,只裂解鼠疫耶尔森菌,而不能裂解假结核耶尔森菌和大肠埃希菌属,其裂解特异性比Pokrovskaya噬菌体更高,成为一个能很好区分鼠疫菌和假结核耶尔森菌的工具,电镜下观察,具有一个等面积的多角形头部(53~63 nm)和一个长的收缩性尾部(110~160 nm)。
(8)YpfΦ:为细丝状病毒,在鼠疫菌CO92(GenBank AL590842.1)基因组的YPO2271~2281位置[10],基因组大小是8.7 kb,在电镜下呈细丝状长1 200 nm,直径8 nm,内含环状正面的单链DNA分子。对于YpfΦ的感染,鼠疫菌是最敏感的(菌株中99%),其次是小肠结肠炎菌株(50%),假结核菌被感染仅有30%。
(9)ΦJA1:系污水中分离出[11],可裂解大多数鼠疫菌而不裂解假结核菌,在一定程度与L-413C同源,在qPCR中可以用L-413C的两对特异性引物中的一对扩增出。
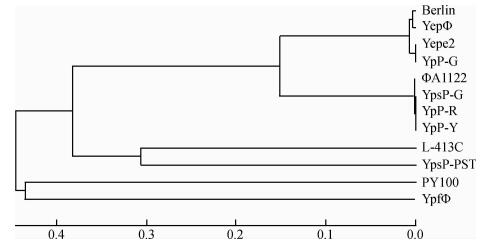
2.鼠疫噬菌体全基因组比对:用Mega 6进化树分析软件分析12株噬菌体进化的结果见图 1。结合BLAST比对,提示12株噬菌体可明显分为6个亚群,即YepΦ亚群(YepΦ、Berlin、Yepe2和YpP-G)、ΦA1122亚群(YpP-Y、YpP-R、YpsP-G和ΦA1122)、L-413 C亚群、PY100亚群、YpsP-PST亚群和YpfΦ亚群。YepΦ和ΦA1122两个亚群间的同源性为78%,其余亚群间无同源性。
|
| 注:距离越近,噬菌体间亲缘关系越近;YepΦ、Berlin、Yepe2、YpP-G之间及YpP-Y、YpP-R、YpsP-G和ΦA1122之间亲缘关系最近 图 1 12株鼠疫菌噬菌体基因组系统发育的构建进化树分析 |
Berlin、YepΦ、Yepe2和YpP-G之间有较高的同源性,YepΦ与Berlin同源性为98%,YepΦ和Yepe2同源性为95.9%,Yepe2和YpP-G的同源性>99%。使用RDP3 Beta42软件在YepΦ亚群中预测出至少4个潜在的重组事件,说明YepΦ亚群的4株噬菌体在进化上关系密切。另外,YpP-Y、YpP-R、YpsP-G及ΦA1122间有较高(99%)同源性。对ΦA1122和L-413C、ΦA1122和YpsP-PST比较无同源性。
虽然鼠疫菌噬菌体在基因序列上有很高的同源性,但在溶解鼠疫菌和假结核菌株的能力上很不相同。如ΦA1122和YpP-G不能裂解的一些假结核菌株,YpP-Y、YpP-R、YpsP-G和YpsP-PST则能裂解。另外,YpP-Y、YpP-R、YpsP-G、YpsP-PST和ΦA1122还能裂解大肠埃希菌ATCC35401、肺炎克雷伯菌env17、志贺菌sonneiS43-46,其原因很可能是志贺菌属与鼠疫耶尔森菌属有某些共同的受体。尽管YpsP-G和ΦA1122基因组有99%相似,但在ΦA1122中缺少YpsP-G存在的4个编码序列,这些密码子编码4个氨基酸,而在ΦA1122中是由另外的密码子编码这些氨基酸,这些编码序列还能编码额外10个氨基酸,而两噬菌体的尾丝蛋白和宿主特异性也不同,YpsP-G能裂解的部分假结核耶尔森菌,ΦA1122不能裂解[8]。
3.鼠疫噬菌体基因组的功能:目前国内外仅对ΦA1122、YepΦ、L-413C、PY100、YpfΦ 5株鼠疫噬菌体有较详细的研究。
(1)ΦA1122:ΦA1122和大肠埃希菌噬菌体T7有高度相似性(89%),两基因组共线性[5]。在ΦA1122基因组特定的1/4区域内,尤其是形态发生和成熟功能区一半的区域内更接近大肠埃希菌T3噬菌体。一些学者用小肠结肠炎耶尔森菌噬菌体Ye03-12重组噬菌体ΦA1122,产生了子代噬菌体;其中一株正好是Demerec和Fano于1945年用于大肠埃希菌分类的T3噬菌体。在分析其基因序列数据后,提示大肠埃希菌T3噬菌体是从2种鼠疫噬菌体的重组过程中演化而来。ΦA1122和T7都能用基因4和10合成gp4 B和gp10 B蛋白质;T7和ΦA1122均有同源预测基因4.1,是基因4的重叠部分,因为ΦA1122的开放阅读框(ORF)缺少一个可用的核糖体结核位点,所以基因4.1在ΦA1122中为不表达。ΦA1122和T7都有基因4.2,是基因4的另一个重叠部分,因为ΦA1122又缺乏可用的核糖体结核位点和一个起始密码子,所以基因4.2在ΦA1122也不表达,但在T7和YeO3-12中可以表达。ΦA1122的基因组长度比T7短2.5 kb,主要差异是缺失1.75 kb片段包括融合了T7的基因0.3和0.7,缺少基因1.4、2.8、5.3和1.7的前120个密码子。
(2)YepΦ:YepΦ共编码45个ORF,其中有38个得到功能注释[12]。对其蛋白质组进行质谱鉴定,共鉴定了10个结构蛋白,ORF 27~38分别编码头蛋白gp6.7,尾部组装蛋白gp7.3,头尾连接蛋白gp8,衣壳组装蛋白gp9,衣壳蛋白gp10,尾管蛋白gp11和gp12以及内核心蛋白gp13、gp14、gp15、gp16、gp17,其中ORF 38产物gp17与T7的尾丝蛋白gp17序列有较强的同源性,尤其表现在N末端序列。T7噬菌体尾丝蛋白的N末端负责将尾丝连接至噬菌体颗粒上,因此预测gp17是YepΦ的尾丝蛋白。ORF39编码穿孔素属于Ⅱ型穿孔素,有2个跨膜区,在合适的裂解时间允许溶菌酶到达细胞壁,ORF40和ORF43分别编码末端酶的大小亚基,负责对基因组单体进行包装并进入衣壳前体。
(3)L-413C:L-413C基因组与大肠埃希菌噬菌体p2的蛋白质有很高的相似性[7],在L-413C编码的39个ORF中,其中有34个ORF与P2噬菌体具有显著的相似性(>87%)。只在P2噬菌体中缺少5个基因(ORF20、Int、C、Cox及ORF40)。和P2完全相似的有4个基因(O、K、W、ORF78),并发现有4个基因(N、ORF20、C、Cox)和其他分离自大肠埃希菌的P2样噬菌体基因预测产物相同,这些基因在P2却没有。L-413C最显著特点是其尾丝蛋白H的马赛克结构,该结构可使L-413C区分鼠疫菌和假结核耶尔森菌。N端207个氨基酸和C端137个氨基酸与P2噬菌体的H蛋白同源,中间一段465个氨基酸的序列可能来自T4噬菌体T6和RB32的H蛋白。
(4)PY100:负责编码衣壳蛋白和参与DNA代谢的基因与大肠埃希菌噬菌体T1具有同源性[7],PY100尾丝蛋白与放线杆菌噬菌体AaΦ23的尾丝蛋白基因有同源性。ORF2编码的蛋白质与沙门菌噬菌体P22的基因3产物(小亚基末端酶)有显著相似性,位于负链的ORF18预测基因产物(大亚基末端酶),ORF2和ORF18与其他噬菌体不同,因为大多数末端酶基因都位于同一DNA链上,而它们却在两条链上。转录末端酶的基因伴随着转录终止子。位于ORF2和ORF18之间的一些小的ORF基因预测产物和已知基因产物没有相似性。ORF34和ORF35编码甲基转移酶和单链DNA结合蛋白,ORF48基因预测产物为细胞内溶素,ORF52、ORF53、ORF54基因预测产物和大肠埃希菌噬菌体T7编码的T1p16、T1p64、T1p62蛋白质相似。ORF56、ORF57、ORF59编码头部蛋白,ORF70、ORF74、ORF78、ORF79、ORF81、ORF82、ORF83编码尾部蛋白。ORF84、ORF93负责DNA代谢,包括合成、降解、重组。ORF88编码T1p22解旋酶,ORF90编码DNA重组酶,ORF91编码核酸内切酶T1p63,ORF92编码核酸外切酶可能涉及到宿主的DNA降解。
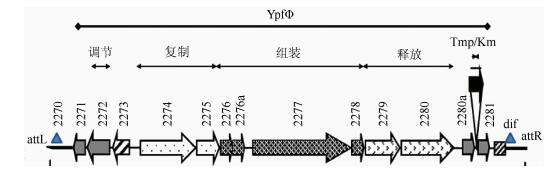
(5)YpfΦ:YpfΦ基因组长8.7 kb,在转录的反方向上有13个ORF(图 2),有一个基因间隔区涉及到转录起始[10],其机构类似于典型霍乱弧菌丝状噬菌体CTXΦ和大肠埃希菌噬菌体Ff。8个ORF涉及到噬菌体复制(YPO2274和YPO2275)、组装(YPO2276~YPO2278)、释放(YPO2279和YPO2280)3个模块。与attR临近处有2个附加的ORF(YPO2280a和YPO2281),无可预测功能。与大肠埃希菌K1强毒株丝状噬菌体CUS-1有7.1 kb以上同源,YPO2280a和YPO2281不存在于霍乱弧菌噬菌体CTXΦ,后者在该位置携带其他2个ORF,编码霍乱毒素,是霍乱弧菌的主要毒力因子。YPO2273编码转录抑制子,有调节噬菌体复制的作用。与attL相邻有2个ORF(YPO2271和YPO2272),在CTXΦ中不存在,在CUS-1中被其他的ORF取代,目前在数据库中还未找到其预测功能。
4.问题和展望:当前对鼠疫噬菌体多样性和进化途径的研究尚处于起始阶段,已知的鼠疫噬菌体基因序列的数量依然有限,且很难将一个新测序的鼠疫噬菌体某些特征与其他已被系统研究过的噬菌体特征联系起来。对鼠疫噬菌体的生物学特性、特异性与宿主菌的相互作用关系,需要更加详尽、明确的证据。迄今鼠疫噬菌体是如何识别宿主菌及其在宿主菌内如何发挥作用,以及在鼠疫菌保存机制中的作用等方面还需深入研究。如从自然界中更大数量分离鼠疫噬菌体,进行基因组功能研究,探明鼠疫噬菌体及其与宿主菌共同起源演化关系,其中鼠疫噬菌体在鼠疫流行静息规律中的作用,将是未来鼠疫噬菌体研究的重要方向。
利益冲突: 无
| [1] | Duckworth DH. "Who discovered bacteriophage?"[J]. Bacteriol Rev, 1976, 40(4): 793–802. |
| [2] |
费来玺.
鼠疫噬菌体研究进展概况[J]. 地方病译丛, 1981(4): 1–4.
Fei LX. Progress of research on Yersinia pestis bacteriophage[J]. Endem Trans, 1981(4): 1–4. |
| [3] | Zhao XN, Wu WL, Qi ZZ, et al. The complete genome sequence and proteomics of Yersinia pestis phage Yep-phi[J]. J Gen Virol, 2011, 92: 216–221. DOI:10.1099/vir.0.026328-0 |
| [4] | Filippov AA, Sergueev KV, He Y, et al. Bacteriophages capable of lysing Yersinia pestis and Yersinia pseudotuberculosis:efficiency of plating tests and identification of receptors in Escherichia coli K-12[J]. Adv Exp Med Biol, 2012, 95: 123–134. DOI:10.1007/978-1-4614-3561-7_16 |
| [5] | Garcia E, Elliott JM, Ramanculov E, et al. The genome sequence of Yersinia pestis bacteriophage phiA1122 reveals an intimate history with the coliphage T3 and T7 genomes[J]. J Bacteriol, 2003, 185(17): 5248–5262. DOI:10.1128/JB.185.17.5248-5262.2003 |
| [6] | Knapp W, Zwillenberg LO. Morphological differences between pasteurella-bacteriophages[J]. Arch Gesamt Virusfors, 1964, 14(4): 563–566. DOI:10.1007/BF01555086 |
| [7] | Schwudke D, Ergin A, Michael K, et al. Broad-host-range Yersinia phage PY100:genome sequence, proteome analysis of virions, and DNA packaging strategy[J]. J Bacteriol, 2007, 190(1): 332–342. DOI:10.1128/JB.01402-07 |
| [8] | Rashid MH, Revazishvili T, Dean T, et al. A Yersinia pestis-specific, lytic phage preparation significantly reduces viable Y. pestis on various hard surfaces experimentally contaminated with the bacterium[J]. Bacteriophage, 2012, 2(3): 168–177. DOI:10.4161/bact.22240 |
| [9] | Garcia E, Chain P, Elliott JM, et al. Molecular characterization of L-413C, a P2-related plague diagnostic bacteriophage[J]. Virology, 2008, 372(1): 85–96. DOI:10.1016/j.virol.2007.10.032 |
| [10] | Derbise A, Carniel E. YpfΦ:a filamentous phage acquired by Yersinia pestis[J]. Front Microbiol, 2014, 5: 701. DOI:10.3389/fmicb.2014.00701 |
| [11] | Filippov AA, Sergueev KV, He Y, et al. Bacteriophage-resistant mutants in Yersinia pestis:identification of phage receptors and attenuation for mice[J]. PLoS One, 2011, 6(9): e25486. DOI:10.1371/journal.pone.0025486 |
| [12] |
赵向娜. 鼠疫耶尔森氏菌噬菌体Yep-phi基因组研究及其受体鉴定[D]. 北京: 中国人民解放军军事科学院, 2013.
Zhao XN. Study on the genome of Yersinia pestis phage Yep-phi and identification of its receptors[D]. Beijing:Academy of Military Sciences PLA China, 2013. |
| [13] | Chouikha I, Charrier L, Filali S, et al. Insights into the infective properties of YpfΦ, the Yersinia pestis filamentous phage[J]. Virology, 2010, 407(1): 43–52. DOI:10.1016/j.virol.2010.07.048 |
 2017, Vol. 38
2017, Vol. 38