文章信息
- 王莉娜, Zhang Zuofeng.
- Wang Lina, Zhang Zuofeng.
- 孟德尔随机化法在因果推断中的应用
- Mendelian randomization approach, used for causal inferences
- 中华流行病学杂志, 2017, 38(4): 547-552
- Chinese journal of Epidemiology, 2017, 38(4): 547-552
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.04.027
-
文章历史
收稿日期: 2016-12-14
2. 90095洛杉矶, 加州大学洛杉矶分校公共卫生学院流行病学系
2. Department of Epidemiology, School of Public Health, University of California, Los Angeles, CA 90095, U.S
传统观察性流行病学研究在发现疾病病因以及因果推断中遇到诸多挑战,比如,反向因果关联、潜在混杂因素、微效暴露因素以及多重检验等,当研究者诉诸于随机对照试验研究(random control trial,RCT)设计,以寻找暴露因素X与疾病结局Y直接关联证据时,又因人类医学伦理和诸多试验设计的局限而难以实践。近年来,孟德尔随机化(Mendelian Randomization,MR)设计通过引入计量经济学中的工具变量(instrumental variable,Ⅳ)的概念,将基因变异作为待研究暴露因素的工具变量,为解决上述问题提供了有效的途径[1]。
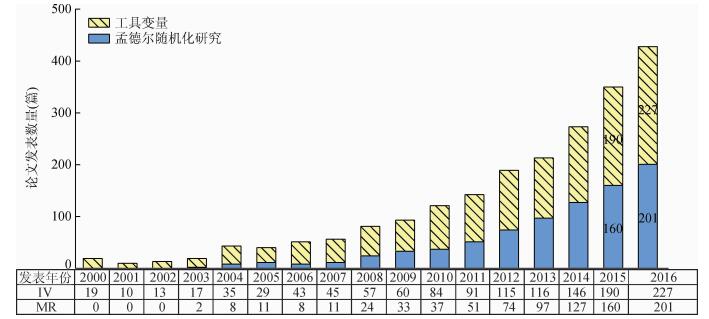
1986年,Katan[2]首次提出MR的遗传思想:由于配子形成时,遵循“亲代等位基因随机分配给子代”的孟德尔遗传规律,如果基因型决定表型,基因型通过表型而与疾病发生关联,因此可以使用基因型作为工具变量来推断表型与疾病之间的关联[3]。由于基因与疾病结局的关联不会受到出生后的环境、社会经济地位、行为因素等常见混杂因素的干扰,且因果时序合理,因此基因作为工具变量进行疾病关联研究已经成为近年来流行病学研究的热点[4-6],特别是近5年以MR法发表论文数量已经翻倍[6](图 1)。本文将就MR设计在流行病学因果关联研究中的应用进展以及常见问题做一简要介绍。
|
| 图 1 孟德尔随机化法研究以及工具变量的应用研究发表情况(Sekula P,et al. JASN,2016 Aug 2;数据更新至2016年10月24日) |
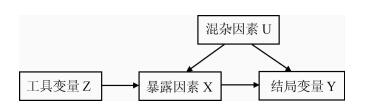
1.关联分析中工具变量的引入:观察性研究的因果推断中,无论多么完美的流行病学研究设计和精准的测量,都不能排除潜在的(potential confounders,C)、无法测量的混杂因素(unmeasured confounders,U)的影响,因此流行病学家试图寻找一种可以忽略潜在混杂因素存在,仍然能够确定因果关联的“理想”方法。工具变量的应用使得流行病学家们的“理想”变成“现实”[7]。工具变量一直被广泛应用于计量经济学中,如回归方程估计、结构方程模型以及两阶段最小二乘法模型中[8],最早被应用于医学领域的是Permutt和Hebel在1989年研究母亲吸烟与新生儿低出生体重的关联时使用的[9],随后Greenland[10]对工具变量在流行病学混杂因素控制方面的应用做了详细阐述:① 工具变量Z与混杂因素U无关联;② 工具变量Z与暴露因素X有关联;③ 工具变量Z与结局变量Y无关联,Z只能通过变量X与Y发生关联。
则:关联ZY=关联ZX×关联XY,见图 2。
|
| 图 2 工具变量在因果关联中的DAG(Directed Acyclic Graph)模型 |
上述方程的使用必须满足条件:① 变量X与Y之间的关联一定会受到潜在混杂因素U的影响,但工具变量Z与变量X以及Z与变量Y之间无潜在混杂因素影响;② 变量X与结局Y之间的关联无法直接观察获得,因为无法直接测量变量X,但是Z是可测量的,并且Z与X直接的关联是已知的或者可测量的,并独立于其他因素而存在[10]。这些对于工具变量的限制条件也使得正确选择合适的工具变量成为关联研究的难点。
2. MR设计:MR是新近出现的流行病学研究设计方法,并且应用日渐广泛。MR遵循“亲代等位基因随机分配给子代”的孟德尔遗传定律,选择合适的“基因变异”作为工具变量,指代无法测量的待研究暴露因素,通过测量遗传变异与暴露因素、遗传变异与疾病结局之间的关联,进而推断暴露因素与疾病结局之间的关联[11]。MR最重要的优点在于遗传变异是可以直接准确测量的,并且不受外界环境、社会行为等因素的影响,属于长期而稳定的暴露因素。MR设计被称为是“最自然”的RCT研究,它不用象传统RCT研究设计那样无法严格控制样本的代表性,它不用设定排除标准即可选择有代表性的样本,并且可以随机分配到各个观察组,最大程度的降低偏倚的作用[12]。
(1)遗传工具变量的选择:MR设计的最关键步骤是寻找合适的遗传变异作为工具变量。研究最多的遗传变异类型是单核苷酸多态性(single nucleotide polymorphism,SNP),事实上任何一种变异类型都可以作为工具变量,包括拷贝数变异(copy number variants,CNV)[13]。建立遗传工具变量的方法一般包括两种:① 选择与目标暴露因素有直接强关联(robust)的遗传变异,如与血清CRP水平直接相关的CRP基因变异(SNPs)[14],与酒精代谢直接相关的乙醇脱氢酶1b(ADH1B)基因变异[15],与白介素6受体水平相关的IL6R基因变异等[16];② 从全基因组关联研究(genomic wide association study,GWAS)数据库获得遗传工具变量,目前全球GWAS研究目录显示(http://www.ebi.ac.uk/gwas/)超过1万条有潜在功能学意义的SNP,其中4 000个以上的SNPs与相应表型有唯一关联[17],可以从中筛选合适的工具变量。
(2)MR设计的策略:MR的研究策略随着统计学方法的深入而被不断地推陈出新,从最早的一阶段MR到单一样本MR、两样本MR、两阶段MR、双向MR以及基因-环境交互作用MR和网络MR,设计方法不断深入,有的方法仅提供因果关联的推断或因果效应的大小估计。各种方法的原理如下:
① 一阶段MR(One stage MR):最早的MR研究设计是由G-X和G-Y的关联来推断X-Y的关联(图 3),也是最为简单的关联推断,因为没有X-Y因果效应大小的估计,只是通过推断来估计X与Y的可能关联,比如,脂蛋白[Lp(a)]相关的KIV-2基因拷贝数变异与血浆Lp(a)水平有关,同时又与心肌梗死(myocardial infarction,MI)发生有关,因此推断Lp(a)水平与MI的发生有关[13],尽管此结论是建立在推断基础上的,但比直接测量研究人群Lp(a)水平和MI发生之间的关联时无法忽略的混杂因素相比,有着更可靠的把握度。但随后,该方法又因无效应大小估计而逐步被取代。
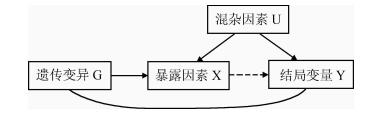
|
| 图 3 孟德尔随机化的一阶段设计DAG(Directed Acyclic Graph)模型 |
② 独立样本MR(One-sample MR):该方法利用单一研究样本,通过使用2阶段最小二乘法回归模型(2-stage least-squares regression,2SLS),定量估计暴露因素X与Y之间的关联效应大小。
如图 4,第一步:建立G-X回归模型,获得暴露因素预测值(predicted value,P),可以使用单个SNP、多个SNPs、等位基因个数或者遗传风险评分(genetic risk scores,GRSs)纳入方程(随着GRS评分增加,暴露因素风险增加多少)。第二步:构建P-Y的回归模型,即获得暴露因素预测值P和结局变量Y之间的回归方程。例如:欲获得X-Y直接的关联,可能受到混杂因素C和U的影响,通过2SLS来实现。见图 4。

|
| 图 4 孟德尔随机化的单一样本设计 |
为获得X-Y关联强度大小:E(Y|X,C,U)=β0+βXX+βCC+βUU
第一步:E(X|C,G)=α0+αCC+αZG=PXCG(PXCG为G-X回归模型暴露因素预测值);
第二步:E(Y|PXG,C)=θ0 +θX PXCG +θCC,
其中第二步中需根据结局变量的类型选择合适的回归模型,如果结局为连续型变量(如标志物水平),使用线性回归模型,结局为二分类变量(如疾病与否),则使用logistic回归模型(θX=log OR)[17]。θX的流行病学意义在于:因为遗传变异所导致的暴露因素水平每增加一个单位,疾病或者结局发生风险增加了多少。由于该方法局限于单个样本,把握度较小,工具变量的选择也比较局限,容易受到潜在混杂因素的影响。2SLS的分析方法在Stata软件中可以使用“ivregress”(StataCorp)、在R软件中使用“ivpack”(R Foundation)来实现。
上述方法针对单一研究人群的资料,而实际上很多研究者无法获得多数研究的原始资料,只能根据汇总后的数据(发表后的论文或者Meta分析合成数据)提供关联证据。如果是单一位点(SNP)作为工具变量,可以使用Wald比值和其sx来估计X-Y关联大小。如图 5,通常Wald比值法有两个步骤:第一步:通过G-X和G-Y的回归模型获得回归系数αg和δg,可以来自现有关联研究数据;第二步:获得G-Y的回归模型系数δg,计算获得X-Y回归模型系数βx=δg/αg,并估计其sx。

|
| 注:αg:G-X关联系数;βx:X-Y关联系数;δg:G-Y关联系数 图 5 孟德尔随机化的单一样本设计DAG(Directed Acyclic Graph)模型 |
目前很多MR的研究使用多个SNPs位点作为工具变量的设计,此时可以使用加权线性回归模型(weighted linear regression),或者使用Wald比值法先进行单个SNP的关联,然后再选择固定效应模型(fixed effect model)或者随机效应模型(random effect model)对多个位点效应进行Meta汇总。但是,两种方法的前提条件必须满足各SNPs之间是完全独立的,或者通过连锁不平衡运算排除SNPs之间的关联[18]。
③ 两样本MR(Two-sample MR):两样本MR的设计策略是建立在G-X和G-Y的关联研究人群来自相同人群的两个独立样本(如GWAS与暴露,GWAS与结局的关联数据[19]),要求两样本具有相似的年龄、性别和种族分布特征,因为样本量较大,该方法可以获得更大的把握度。目前,两样本MR因为全球大量GWAS合作组的公共数据而被广泛使用[19],比如国际血压研究合作组(the International Consortium for Blood Pressure),冠心病全基因组重复验证和Meta分析合作组(Coronary Artery Disease Genome wide Replication and Meta-analysis,CARDIoGRAM,http://www.cardiogramplusc4d.org/),全球血脂遗传合作组(Global Lipids Genetics Consortium),全球吸烟与遗传合作组(Tobacco and Genetics Consortium)等,可以通过合作组的网站直接下载G-X与G-Y关联结果数据。此设计是基于现有数据,具有把握度大、经济、高效的特点[18]。
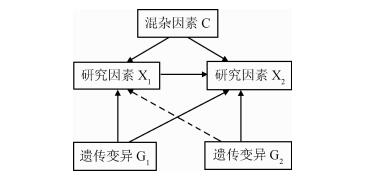
④ 双向MR(Bidirectional MR):又称为互为MR(Reciprocal MR),如图 6,如果待研究因素X1与研究因素X2有关联,遗传变异G1与X1和X2将都有关联,但是遗传变异G2与X2有关却与X1不存在关联(虚线)[20]。这种方法有助于进一步理清危险因素与疾病结局之间的关联。如Timpson等[21]在双向MR设计中使用肥胖基因FTO(rs9939609)(G1)和CRP基因(rs3091244)(G2)作为工具变量,分别指代BMI(X1)和循环CRP水平(X2),观察性研究结果提示BMI与循环CRP之间有关联(P<0.000 1),但无法推断因果方向。通过FTO(rs9939609)指代BMI与CRP之间有显著性关联(P=0.006),而CRP(rs3091244)指代CRP与BMI之间无显著性关联(P=0.2),可以推断BMI升高可引起肥胖症进而引起CRP水平改变,但CRP水平不会引起肥胖症[21]。此方法在解决因果网络方向的问题上将会有很大用途[22],但是在分析未知生物学效应的两个变量时,要防止被双向MR的结果误导[20]。
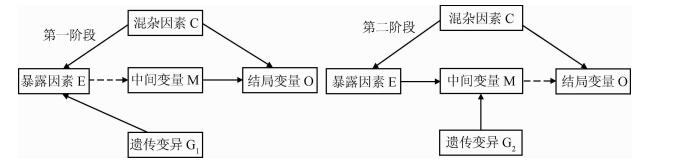
⑤ 两阶段MR(Two-step MR):与两样本MR不同的是,两阶段MR需要使用遗传工具变量来评价因果关联的可能中间变量M(Mediation),来探讨环境暴露因素(E)是否通过表观遗传指标(M)而导致疾病(O)改变,见图 7。第一阶段,遗传工具变量G1独立于混杂因素,指代暴露因素E与结局O之间的关联,并且必须经过中间变量M才能实现;第二阶段,另一独立遗传工具变量G2作为中间变量M的指代工具,分析中间变量M与结局O之间的关联。比如BMI通过血压来间接影响冠心病的发生[23]。目前此方法已被应用于表观遗传流行病学(Epigenetic Epidemiology)研究[24],Binder和Michels[25]使用母亲MTHFR C677T,A1298C两位点作为工具变量,发现7个CpG位置参与了红细胞叶酸与甲基化改变之间的关联。Dekkers等[26]使用全基因组甲基化数据发现,免疫细胞差异甲基化结果是由个体内部血脂水平(TG,LDL-C,HDL-C)变化所导致,反之则不亦然。此方法必须满足E-M和E-O之间的关联呈线性以及同质性的假设前提,并且已被延伸成为分析复杂因果网络关系的基础,如网络MR设计(Network MR)[27]。
⑥ 基因-暴露交互作用MR(Gene-exposure interactions):MR研究设计还可以用于探讨基因-暴露因素在疾病发生中的交互作用现象,同时要求基因与结局的关联必须取决于暴露因素的状态。这种方法可以区分基因直接作用于结局,还是基因通过暴露因素而作用于结局。如Holmes等[28]发现携带酒精代谢酶基因ADH1B rs1229984突变等位基因A的个体不饮酒或者少量饮酒的比例更高,进而发生冠心病和中风的风险亦显著降低。假设同时满足ADH1B rs1229984 A在饮酒者中与冠心病和中风的发生无关,这说明ADH1B rs1229984与冠心病和中风的关联不通过饮酒而发生(直接效应),可能会有另外的通路存在。因此需要有无暴露组或亚人群的基因-疾病无关联的证据支持。Brunner等[15]同样使用酒精代谢酶基因(ADH,ALDH)的68个SNPs作为工具变量,发现饮酒不能增加前列腺癌的风险,但是可影响疾病预后。Taylor等[29]使用大样本欧洲人群MR研究证实饮用咖啡不能降低前列腺癌的风险。
二、 MR研究可靠性评价1.敏感度分析(sensitivity analysis):MR分析中如果出现非特异性的SNPs作为工具变量(即SNPs与目标暴露因素有关,同时与其他暴露因素也有关联),此时可以使用敏感度分析来确定其存在可以造成多大的影响。如果剔除非特异性SNPs以后,其他遗传工具变量和结局之间的关联结果依然有统计学意义,则说明暴露因素与结局的因果关联证据更强。
2. MR-Egger回归分析:如果采用多个SNPs作为工具变量进行因果推断,很难避免基因多效性(pleiotropy)带来的偏倚,一方面可以使用直观的漏斗图的对称性来判断小样本研究是否存在偏倚,同时也可以使用MR-Egger回归分析的方法来评价基因多效性带来的偏倚,MR-Egger回归直线的斜率可以估计定向多效性(directional pleiotropy)的大小[30]。Bowden等[31]提出在MR-Egger回归中使用I2检验来估计多效性带来的偏倚,特别是在两样本设计的MR研究中,该方法基于“无测量误差”的无效假设前提(NO Measurement Error,NOME),并且基因-暴露之间的关联已被确认,使用I2GX(0~1)的大小反映MR-Egger可能存在的偏倚,越接近1,说明因果关联越不可能被潜在的偏倚所影响。但是Hartwig和Davies[32]对此研究提出部分质疑,认为其忽略弱工具变量偏倚(weak instrument bias)。
三、 MR研究的局限性近年来各种统计新方法、大样本GWAS数据、分子表观遗传学以及各种“组学”技术的应用,为MR研究复杂暴露因素与疾病结局因果关联提供了广阔的基础,也使得流行病学与多种学科交叉上升到新的台阶。随着应用的广泛深入,人们对MR研究的应用在“理想”和“现实”之间也日趋理智。MR仍然有些问题比较棘手:① 难以发现合适的遗传工具变量:并非所有SNPs都适宜作为工具变量,基于GWAS的GRS也并非完美,很难控制弱工具变量偏倚[32]。② 把握度较低:只有通过扩大样本量获得足够的把握度,比如使用仅占1%效应的遗传工具探讨暴露和疾病之间的关联,至少需要9 500对以上的病例和对照样本才能有80%的把握度获得增加50%(OR=1.5)的生物学效应(每个标准差水平)[33]。③ Beavis效应:基于GWAS数据的MR研究可能会高估了遗传和暴露之间的关联,亦被称之为“胜利者的诅咒(the winner ’s curse)”,因为SNPs与混杂因素之间可能有潜在的关联[34]。④ 合理的生物学解释:MR研究发现高水平IL 6R可降低心血管疾病(CAD)的风险[16],而观察性研究结果提示IL 6R与CAD风险增加有关[35],因此需要进一步研究验证。尽管如此,MR仍然在因果推断中发挥了重要作用,并不断完善。
利益冲突: 无
| [1] | Lawlor DA, Harbord RM, Sterne JAC, et al. Mendelian randomization:Using genes as instruments for making causal inferences in epidemiology[J]. Stat Med, 2008, 27(8): 1133–1163. DOI:10.1002/sim.3034 |
| [2] | Katan MB. Apolipoprotein E isoforms, serum cholesterol, and cancer[J]. Lancet, 1986, 1(8479): 507–508. |
| [3] | Thomas DC, Conti DV. Commentary:the concept of 'Mendelian Randomization'[J]. Int J Epidemiol, 2004, 33(1): 21–25. DOI:10.1093/ije/dyh048 |
| [4] | Didelez V, Sheehan N. Mendelian randomization as an instrumental variable approach to causal inference[J]. Stat Methods Med Res, 2007, 16(4): 309–330. DOI:10.1177/0962280206077743 |
| [5] | Thanassoulis G, O'Donnell CJ. Mendelian randomization:nature's randomized trial in the post-genome era[J]. JAMA, 2009, 301(22): 2386–2388. DOI:10.1001/jama.2009.812 |
| [6] | Sekula P, del Greco MF, Pattaro C, et al. Mendelian randomization as an approach to assess causality using observational data[J]. J Am Soc Nephrol, 2016, 27(11): 3253–3265. DOI:10.1681/ASN.2016010098 |
| [7] | Hernán MA, Robins JM. Instruments for causal inference:an epidemiologist's dream[J]. Epidemiology, 2006, 17(4): 360–372. DOI:10.1097/01.ede.0000222409.00878.37 |
| [8] | Martens EP, Pestman WR, de Boer A, et al. Instrumental variables:application and limitations[J]. Epidemiology, 2006, 17(3): 260–267. DOI:10.1097/01.ede.0000215160.88317.cb |
| [9] | Permutt T, Hebel JR. Simultaneous-equation estimation in a clinical trial of the effect of smoking on birth weight[J]. Biometrics, 1989, 45(2): 619–622. DOI:10.2307/2531503 |
| [10] | Greenland S. An introduction to instrumental variables for epidemiologists[J]. Int J Epidemiol, 2000, 29(4): 722–729. DOI:10.1093/ije/29.4.722 |
| [11] | Smith GD, Ebrahim S. 'Mendelian randomization':can genetic epidemiology contribute to understanding environmental determinants of disease[J]. Int J Epidemiol, 2003, 32(1): 1–22. DOI:10.1093/ije/dyg070 |
| [12] | Hingorani A, Humphries S. Nature's randomised trials[J]. Lancet, 2005, 366(9501): 1906–1908. DOI:10.1016/S0140-6736(05)67767-7 |
| [13] | Kamstrup PR, Tybjaerg-Hansen A, Steffensen R, et al. Genetically elevated lipoprotein (a) and increased risk of myocardial infarction[J]. JAMA, 2009, 301(22): 2331–2339. DOI:10.1001/jama.2009.801 |
| [14] | Elliott P, Chambers JC, Zhang W, et al. Genetic Loci associated with C-reactive protein levels and risk of coronary heart disease[J]. JAMA, 2009, 302(1): 37–48. DOI:10.1001/jama.2009.954 |
| [15] | Brunner C, Davies NM, Martin RM, et al. Alcohol consumption and prostate cancer incidence and progression:a Mendelian randomisation study[J]. Int J Cancer, 2017, 140(1): 75–85. DOI:10.1002/ijc.30436.Epub2016Oct8 |
| [16] | Interleukin-6 Receptor Mendelian Randomisation Analysis (IL 6R MR) Consortium. The interleukin-6 receptor as a target for prevention of coronary heart disease:a mendelian randomisation analysis[J]. Lancet, 2012, 379(9822): 1214–1224. DOI:10.1016/S0140-6736(12)60110-X.Epub2012Mar14 |
| [17] | Haycock PC, Burgess S, Wade KH, et al. Best (but oft-forgotten) practices:the design, analysis, and interpretation of Mendelian randomization studies[J]. Am J Clin Nutrit, 2016, 103(4): 965–978. DOI:10.3945/ajcn.115.118216 |
| [18] | Pierce BL, Burgess S. Efficient design for Mendelian randomization studies:subsample and 2-sample instrumental variable estimators[J]. Am J Epidemiol, 2013, 178(7): 1177–1184. DOI:10.1093/aje/kwt084 |
| [19] | Burgess S, Scott RA, Timpson NJ, et al. Using published data in Mendelian randomization:a blueprint for efficient identification of causal risk factors[J]. Eur J Epidemiol, 2015, 30(7): 543–552. DOI:10.1007/s10654-015-0011-z |
| [20] | Smith GD, Hemani G. Mendelian randomization:genetic anchors for causal inference in epidemiological studies[J]. Hum Mol Genet, 2014, 23(R1): R89–98. DOI:10.1093/hmg/ddu328 |
| [21] | Timpson NJ, Nordestgaard BG, Harbord RM, et al. C-reactive protein levels and body mass index:Elucidating direction of causation through reciprocal Mendelian randomization[J]. Int J Obes (Lond), 2011, 35(2): 300–308. DOI:10.1038/ijo.2010.137 |
| [22] | Neto EC, Keller MP, Attie AD, et al. Causal graphical models in systems genetics:a unified framework for joint inference of causal network and genetic architecture for correlated phenotypes[J]. Ann Appl Stat, 2010, 4(1): 320–339. DOI:10.1214/09-AOAS288 |
| [23] | International Consortium for Blood Pressure Genome-Wide Association Studies. Genetic variants in novel pathways influence blood pressure and cardiovascular disease risk[J]. Nature, 2011, 478(7367): 103–109. DOI:10.1038/nature10405 |
| [24] | Latvala A, Ollikainen M. Mendelian randomization in (epi) genetic epidemiology:an effective tool to be handled with care[J]. Genome Biol, 2016, 17(1): 156. DOI:10.1186/s13059-016-1018-9 |
| [25] | Binder AM, Michels KB. The causal effect of red blood cell folate on genome-wide methylation in cord blood:a Mendelian randomization approach[J]. BMC Bioinformatics, 2013, 14: 353. DOI:10.1186/1471-2105-14-353 |
| [26] | Dekkers KF, van Iterson M, Slieker RC, et al. Blood lipids influence DNA methylation in circulating cells[J]. Genome Biol, 2016, 17(1): 138. DOI:10.1186/s13059-016-1000-6 |
| [27] | Burgess S, Daniel RM, Butterworth AS, et al. Network Mendelian randomization:using genetic variants as instrumental variables to investigate mediation in causal pathways[J]. Int J Epidemiol, 2015, 44(2): 484–495. DOI:10.1093/ije/dyu176 |
| [28] | Holmes MV, Dale CE, Zuccolo L, et al. Association between alcohol and cardiovascular disease:Mendelian randomisation analysis based on individual participant data[J]. BMJ, 2014, 349: G4164. DOI:10.1136/bmj.g4164 |
| [29] | Taylor AE, Martin RM, Geybels MS, et al. Investigating the possible causal role of coffee consumption with prostate cancer risk and progression using Mendelian randomization analysis[J]. Int J Cancer, 2017, 140(2): 322–328. DOI:10.1002/ijc.30462 |
| [30] | Bowden J, Smith GD, Burgess S. Mendelian randomization with invalid instruments:effect estimation and bias detection through Egger regression[J]. Int J Epidemiol, 2015, 44(2): 512–525. DOI:10.1093/ije/dyv080 |
| [31] | Bowden J, del Greco MF, Minelli C, et al. Assessing the suitability of summary data for two-sample Mendelian randomization analyses using MR-Egger regression:the role of the I2 statistic[J]. Int J Epidemiol, 2016. DOI:10.1093/ije/dyw220 |
| [32] | Hartwig FP, Davies NM. Why internal weights should be avoided (not only) in MR-Egger regression[J]. Int J Epidemiol, 2016, 45(5): 1676–1678. DOI:10.1093/ije/dyw240 |
| [33] | Burgess S. Sample size and power calculations in Mendelian randomization with a single instrumental variable and a binary outcome[J]. Int J Epidemiol, 2014, 43(3): 922–929. DOI:10.1093/ije/dyu005 |
| [34] | Goring HH, Terwilliger JD, Blangero J. Large upward bias in estimation of locus-specific effects from genomewide scans[J]. Am J Hum Genet, 2001, 69(6): 1357–1369. DOI:10.1086/324471 |
| [35] | IL 6R Genetics Consortium Emerging Risk Factors Collaboration. Interleukin-6 receptor pathways in coronary heart disease:a collaborative meta-analysis of 82 studies[J]. Lancet, 2012, 379(9822): 1205–1213. DOI:10.1016/S0140-6736(11)61931-4 |
 2017, Vol. 38
2017, Vol. 38