文章信息
- 罗伟平, 杜雨峰, 黄靖, 黄武卿, 徐铭, 严波, 莫雄飞, 张彩霞.
- Luo Weiping, Du Yufeng, Huang Jing, Huang Wuqing, Xu Ming, Yan Bo, Mo Xiongfei, Zhang Caixia.
- 外周血全基因组DNA甲基化在甲基供体与乳腺癌关系中作用的研究
- Effect of peripheral bloodgenomic DNA methylation on the relationship between methyl donor status and risk of breast cancer
- 中华流行病学杂志, 2017, 38(4): 537-541
- Chinese journal of Epidemiology, 2017, 38(4): 537-541
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.04.025
-
文章历史
收稿日期: 2016-09-05
2. 510180 广州医科大学附属广州市第一人民医院预防保健科;
3. 510080 广州, 中山大学附属第一医院血管甲状腺外科
2. Department of Prevention and Health Care, Guangzhou First People's Hospital, Guangzhou Medical University, Guangzhou 510180, China;
3. Department of Thyroid Breast Surgery, The First Affiliated Hospital, Sun Yat-sen University, Guangzhou 510080, China
DNA甲基化异常与乳腺癌的发生密切相关。Szyf等[1]发现乳腺癌形成的过程中有两个相互矛盾的DNA甲基化模式:全基因组的低甲基化以及特定基因的区域性高甲基化。全基因组DNA甲基化水平受外源性和内源性两种因素的调节,外源性因素中一个重要方面就是膳食甲基供体及相关营养素,包括叶酸、蛋氨酸、总胆碱和甜菜碱的供给。基于此,本研究重点探讨外周血全基因组DNA甲基化程度在甲基供体摄入与乳腺癌发病关系中的作用。本研究的研究假设是甲基供体可能通过影响全基因组DNA甲基化水平而影响乳腺癌。
对象与方法1.研究对象:采用频数匹配的病例对照研究设计。共纳入300例乳腺癌病例和300例对照。病例组来自中山大学附属第一医院和广东省妇幼保健院,选取自2011年9月以后新入院的乳腺癌病例。纳入标准:年龄25~70岁的女性;广东户籍或在广东省居住达≥5年;病理组织学确诊的乳腺癌病例,确诊到调查时间不超过3个月;未同时或曾经患其他恶性肿瘤。对照组来自与病例同时期同医院来源的合格的完成膳食问卷的非肿瘤病例。对照组与病例组按照年龄(±5岁)进行频数匹配。本研究由中山大学公共卫生学院伦理委员会批准。应用PASS 11.0软件估计所需样本量。检验水准α=0.05,检出功效1-β不低于95%,对照组甲基供体及相关营养素暴露率P0为25%,预期效应强度OR值估计为0.40[2],为了保证检验效率在估计样本量基础上增加10%,计算得到病例组和对照组样本量至少各为242例。
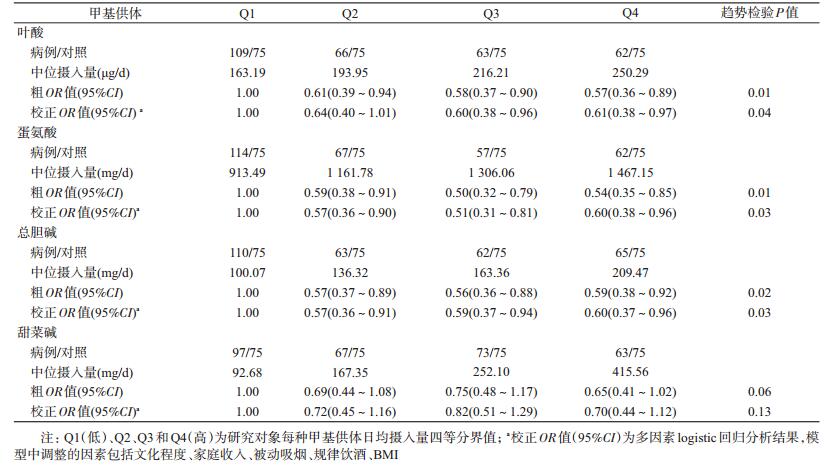
2.资料收集与膳食调查:采用结构问卷,由经过专业培训的调查员以面对面访谈的方式进行调查。采用经过信度和效度评估的食物频数问卷(food frequency questionnaire,FFQ)收集膳食数据[3],FFQ包括总共7大类81小类。通过研究对象回忆过去1年内各类食物的食用频率(未吃、每日、每周、每月或每年次数)和每次食用份量(中碗、片、中个、杯、两、中粒、个、小匙、中盒、中片、条),并参照中国食物成分表(2004版)换算出甲基供体叶酸(μg/d)、蛋氨酸(mg/d)、胆碱(mg/d)和甜菜碱(mg/d)的日均摄入量。日均甲基供体等营养素的摄入量公式:I=F×R×T×S×E×C,其中,I为日均营养素摄入量;F为食物摄入频率;R为年/月/周/日的时间系数;T为每次摄入的份量;S为该食物份量的平均质量(g);E为该食物在食物成分表中的可食部(%);C为食物成分表提供的食物营养素含量。将对照组每种甲基供体的日均摄入量进行四等分,然后根据对照组四等分的界值,将所有研究对象每种甲基供体日均摄入量划分成4组,即Q1(低)、Q2、Q3和Q4(高)。
各种膳食营养成分的日均摄入量采用食物图谱协助被调查者估计食物的食用量。调查的潜在混杂因素包括社会人口学特征(年龄、婚姻状况、职业、文化程度、家庭收入)、目前的体重和身高、月经和生育史、避孕药使用史、良性乳腺疾病史、一级亲属乳腺癌家族史、体力活动、主动和被动吸烟以及饮酒等。相关临床诊断和病理结果从医院病历中抄录。
3. DNA提取及外周血全基因组DNA甲基化分析:病例组和对照组研究对象被要求采血前一天20:00后禁食,保证空腹12 h。次日早上使用含乙二胺四乙酸(EDTA)的抗凝管收集研究对象刚入院尚未接受任何治疗前的外周血液标本,在室温下静置30 min,随后在4 ℃、800×g离心15 min,分为血浆、白细胞层和红细胞,并进行分装,其中白细胞层用于后续的DNA提取。采用天根生化科技(北京)有限公司生产的全基因组DNA提取试剂盒提取DNA,使用美国Epigentek公司生产的MethylFlashTM Methylated DNA Quantification Kit(Colorimetric)进行外周血全基因组DNA甲基化分析,采用ELISA法。检测的样品均设置复孔,并在每8个样品中设置1个重复样品,计算得到重复样品与原样品的批内变异系数为4%,批间变异系数为8%。
4.统计学分析:采用SPSS 20.0统计软件进行资料分析。对于连续型变量采用均值±标准差或M(P25~P75)进行描述,并使用t检验或Wilcoxon秩和检验进行病例和对照组之间的差异比较,外周血全基因组DNA甲基化程度经倒数转换后呈正态分布,使用t检验;对于分类变量,采用人数和百分比进行描述,通过χ2检验对病例和对照组之间的差异进行检验。采用非条件logistic回归分析方法进行多因素分析,使用Enter法强制纳入可疑的混杂因素,计算其OR值和95%CI。把各分类变量作为连续型变量,检验剂量反应趋势,计算Wald统计量和P值。
运用AMOS 17.0软件进行通径分析,探讨叶酸、蛋氨酸、总胆碱和甜菜碱摄入与乳腺癌发病的关系。根据甲基供体摄入、外周血全基因组DNA甲基化程度和乳腺癌发病的关系,绘制相应的模型。其中甲基供体摄入以经残差法调整后的连续型变量形式进入模型;全基因组DNA甲基化程度以连续型变量形式进入模型;乳腺癌发病以二分类变量(是/否)进入模型。因存在二分类变量,处理过程采用贝叶斯分析法[4]。执行计算命令后得到路径图和通径系数,路径图直观表现甲基供体摄入、全基因组DNA甲基化程度和乳腺癌发病之间的相互关系。每条路径上相应的通径系数,即标准化回归系数β,定量地反映各个变量之间的相互关系,β的符号反映了作用的方向,绝对值大小反映影响程度。以P<0.05为差异有统计学意义。
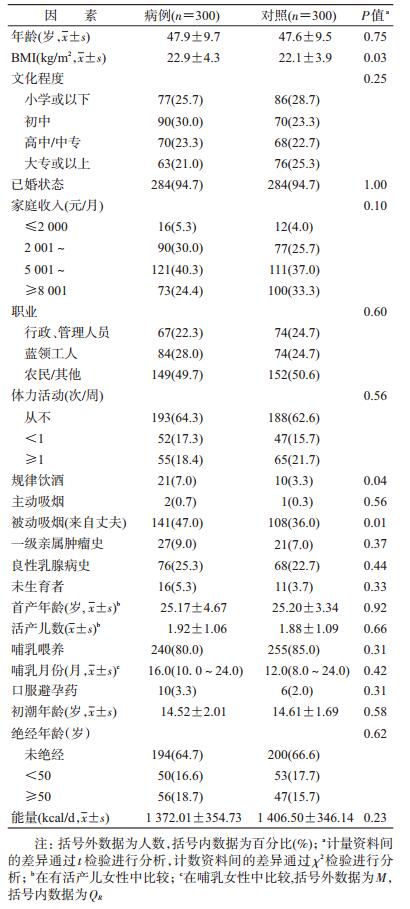
结果1.一般人口学特征:病例组和对照组的年龄为(47.9±9.7)岁和(47.6±9.5)岁,差异无统计学意义。与对照组相比,乳腺癌病例BMI较高,规律饮酒人数较多,被动吸烟(来自丈夫)人数较多。两组其他基线特征和生育因素(包括是否生育、首产年龄、活产数、是否哺乳、哺乳月份、初潮年龄、口服避孕药和绝经年龄)的差异无统计学意义(P>0.05),见表 1。
2.甲基供体与乳腺癌发病的关系:膳食中叶酸、蛋氨酸、总胆碱的摄入是乳腺癌发病的保护因素,并且存在剂量反应关系。校正潜在的混杂因素后,与摄入最低组(Q1)相比,叶酸、蛋氨酸、总胆碱的摄入最高组(Q4)的OR值(95%CI)分别为0.61(0.38~0.97)、0.60(0.38~0.96)、0.60(0.37~0.96),趋势性检验均有统计学意义(均P<0.05)。而甜菜碱摄入与乳腺癌的发病无关(表 2)。
3.外周血全基因组DNA甲基化与乳腺癌发病的关系:病例组和对照组外周血全基因组DNA甲基化率分别为0.46%±0.25%和0.53%±0.34%,差异有统计学意义(P<0.01)。校正混杂因素(文化程度、家庭收入、被动吸烟、规律饮酒、BMI)后,外周血全基因组DNA甲基化的OR值(95%CI)为0.44(0.25~0.79)(P<0.01)。
4.外周血全基因组DNA甲基化在甲基供体与乳腺癌发病中的关系:路径分析结果未发现叶酸、总胆碱和甜菜碱与外周血全基因组DNA甲基化程度间存在联系(β=0.010,β=0.001,β=0.001,P>0.05)。蛋氨酸摄入与外周血全基因组DNA甲基化程度呈正相关(β=0.065,P<0.05),而外周血全基因组DNA甲基化程度与乳腺癌发病风险呈负相关(β=-0.027,P<0.05),提示外周血全基因组DNA甲基化在蛋氨酸摄入与乳腺癌发病风险之间起了中介作用。
讨论DNA甲基化是肿瘤发生发展过程中重要的表观遗传改变,本研究结果显示,病例组外周血全基因组DNA甲基化率低于对照组,差异有统计学意义(P<0.01),提示外周血全基因组DNA甲基化水平降低会增加乳腺癌的发病风险。既往的三项人群研究均发现全基因组DNA甲基化水平降低会增加乳腺癌发病风险[5-7],与本研究结果一致。在结直肠和膀胱肿瘤的研究中,也发现了全基因组DNA甲基化水平降低增加相关肿瘤的发病风险[8-9]。目前普遍认为外周血全基因组DNA低甲基化导致肿瘤的发生是通过导致染色体不稳定性而致病,其主要过程是外周血全基因组DNA低甲基化可以导致突变率增加,形成突变热点,增加染色体的不稳定性,使转座子异常表达,继而引起基因表达异常,导致细胞恶变,最终形成肿瘤[10]。在肿瘤细胞中,外周血全基因组低甲基化和特定基因高甲基化可同时发生[1],抑癌基因p16,已被发现在乳腺癌患者中存在高甲基化,并与抑癌基因的失活相关,从而干扰抑癌基因的正常功能导致肿瘤的发生[11]。然而,这两种表观遗传改变是各自独立的过程,并在肿瘤形成及进展过程中扮演不同的角色[1]。本研究着重探讨了外周血全基因组DNA甲基化程度与乳腺癌发生的关系,后续的研究将对相关抑癌基因的甲基化进行探索。
本研究发现外周血全基因组DNA甲基化在蛋氨酸摄入与乳腺癌发病风险之间起了中介作用,可能机制为蛋氨酸的代谢产物SAM可为一碳循环提供甲基,继而影响全基因组DNA甲基化程度,最终导致肿瘤的发生[12]。本研究在其他甲基供体摄入与乳腺癌的路径分析中未发现外周血全基因组DNA甲基化所起的作用。动物实验中发现,低叶酸膳食会降低基因组的甲基化程度,而在补充充足的叶酸后甲基化程度可以恢复至正常水平[13]。提示甲基供体的摄入可能导致全基因组DNA甲基化程度的改变,从而影响疾病的发生。本研究尽管未发现外周血全基因组DNA甲基化在叶酸摄入和乳腺癌间的中介作用,但结果的P值接近0.05,提示可能存在这种趋势。在4种甲基供体中本研究仅发现蛋氨酸摄入可影响外周血全基因组DNA甲基化程度,可能由于本研究样本量较小,未能发现其他甲基供体对外周血全基因组甲基化程度的影响。外周血全基因组DNA甲基化作为甲基供体和乳腺癌发病之间可能存在的中介因素,其水平受外源性和内源性两种因素的调节。本研究关注的甲基供体摄入属于外源性因素,然而该机制还可能受其他因素的影响[14],因此需要更大型的研究进行探讨。
本研究具有以下优点:使用的FFQ量表已在广东人群经过信度效度评价,适用于本研究人群。通过科学的方法对研究人群的基线特征和生育因素等资料进行了全面收集,保证可以获得潜在混杂因素,并在回归模型中进行了校正。能量是膳食研究中重要的混杂因素,采用能量残差法对营养素的摄入量进行校正。利用路径分析法探讨全基因组DNA甲基化在甲基供体和乳腺癌之间的中介作用。
本研究存在局限性。病例对照研究可能存在选择偏倚。为了降低选择偏倚,同时期在中山大学附属第一医院多个科室选择对照,具有较宽的疾病谱,包括各种眼部疾病、耳鼻喉疾病、静脉曲张和急性阑尾炎等,这些疾病与膳食因素不存在明显关联。且病例组和对照组的高应答率也降低了选择偏倚对本研究结果的影响。病例对照研究还存在一定的信息偏倚。为了降低来自研究对象方面的信息偏倚,采用新发病例,同时,调查者在调查过程中采用食物图谱帮助参与者准确回忆膳食摄入情况。外周血全基因组DNA甲基化检测采用盲测,全基因组DNA甲基化检测设置复孔,重复样一致率、批内和批间变异系数均满足质控要求。
利益冲突: 无
| [1] | Szyf M, Pakneshan P, Rabbani SA. DNA methylation and breast cancer[J]. Biochem Pharmacol, 2004, 68(6): 1187–1197. DOI:10.1016/j.bcp.2004.04.030 |
| [2] | Zhang CX, Pan MX, Li B, et al. Choline and betaine intake is inversely associated with breast cancer risk:A two-stage case-control study in China[J]. Cancer Sci, 2013, 104(2): 250–258. DOI:10.1111/cas.12064 |
| [3] | Zhang CX, Ho SC. Validity and reproducibility of a food frequency Questionnaire among Chinese women in Guangdong province[J]. Asia Pac J Clin Nutr, 2009, 18(2): 240–250. DOI:10.6133/apjcn.2009.18.2.13 |
| [4] | Na KS, Oh SJ, Jung HY, et al. Alexithymia and low cooperativeness are associated with suicide attempts in male military personnel with adjustment disorder:A case-control study[J]. Psychiatry Res, 2013, 205(3): 220–226. DOI:10.1016/j.psychres.2012.08.027 |
| [5] | Choi JY, James SR, Link PA, et al. Association between global DNA hypomethylation in leukocytes and risk of breast cancer[J]. Carcinogenesis, 2009, 30(11): 1889–1897. DOI:10.1093/carcin/bgp143 |
| [6] | Xu X, Gammon MD, Hernandez-Vargas H, et al. DNA methylation in peripheral blood measured by LUMA is associated with breast cancer in a population-based study[J]. FASEB J, 2012, 26(6): 2657–2666. DOI:10.1096/fj.11-197251 |
| [7] | Kuchiba A, Iwasaki M, Ono H, et al. Global methylation levels in peripheral blood leukocyte DNA by LUMA and breast cancer:a case-control study in Japanese women[J]. Br J Cancer, 2014, 110(11): 2765–2771. DOI:10.1038/bjc.2014.223 |
| [8] | Lim U, Flood A, Choi SW, et al. Genomic methylation of leukocyte DNA in relation to colorectal adenoma among asymptomatic women[J]. Gastroenterology, 2008, 134(1): 47–55. DOI:10.1053/j.gastro.2007.10.013 |
| [9] | Moore LE, Pfeiffer RM, Poscablo C, et al. Genomic DNA hypomethylation as a biomarker for bladder cancer susceptibility in the Spanish Bladder Cancer Study:a case-control study[J]. Lancet Oncol, 2008, 9(4): 359–366. DOI:10.1016/S1470-2045(08)70038-X |
| [10] | Esteller M. DNA methylation and cancer therapy:new developments and expectations[J]. Curr Opin Oncol, 2005, 17(1): 55–60. DOI:10.1097/01.cco.0000147383.04709.10 |
| [11] | Cho YH, Shen J, Gammon MD, et al. Prognostic significance of gene-specific promoter hypermethylation in breast cancer patients[J]. Breast Cancer Res Treat, 2012, 131(1): 197–205. DOI:10.1007/s10549-011-1712-y |
| [12] | Hoffman RM. Altered methionine metabolism, DNA methylation and oncogene expression in carcinogenesis:a review and synthesis[J]. Biochim Biophys Acta, 1984, 738(1/2): 49–87. DOI:10.1016/0304-419X(84)90019-2 |
| [13] | Mckay JA, Williams EA, Mathers JC. Folate and DNA methylation during in utero development and aging[J]. Biochem Soc Trans, 2004, 32: 1006–1007. DOI:10.1042/BST0321006 |
| [14] | Shames DS, Minna JD, Gazdar AF. DNA methylation in health, disease, and cancer[J]. Curr Mol Med, 2007, 7(1): 85–102. DOI:10.2174/156652407779940413 |
 2017, Vol. 38
2017, Vol. 38