文章信息
- 陈谦, 仇小强.
- Chen Qian, Qiu Xiaoqiang.
- 基于大数据的肝癌血管侵袭标志物分析
- Bioinformatics on vascular invasion markers in hepatocellular carcinoma via Big-Data analysis
- 中华流行病学杂志, 2017, 38(4): 522-527
- Chinese journal of Epidemiology, 2017, 38(4): 522-527
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.04.022
-
文章历史
收稿日期: 2016-11-02
肝细胞癌(肝癌)具有较高的死亡率[1-2]。我国是肝癌高发的国家之一,每年死于肝癌的人数占全球肝癌年死亡人数的50%[3-5]。目前肝癌的发病机制尚不清楚,治疗方式主要通过手术切除,但术后容易转移复发,5年生存率仅有20%~30%[6]。血管侵袭是影响肝癌转移复发的主要因素之一,在实际治疗过程中,只有术后通过显微镜观察才能判断是否发生血管侵袭,因此,研究其生物标志物对肝癌的预后十分重要。microRNA是一类长度为21~23 nt的内源性小分子RNA,主要通过转录后对靶基因负调控发挥作用[7]。近年来,国内外学者对microRNA在肝癌的发生、发展中进行了多项研究[8-14],但利用公共组学数据研究microRNA在肝癌血管侵袭中的作用鲜有报道。癌症基因组图谱(The Cancer Genome Atlas,TCGA)和人类肿瘤相关的基因表达汇编(Gene Expression Omnibus,GEO)数据库是两个最大的组学数据库。本研究分析GEO数据库中177例样本的microRNA芯片表达谱数据,发现在肝癌血管侵袭中高表达的microRNA,并结合TCGA数据库中362例肝癌病例预后数据,发现与预后相关的microRNA,并对其所调控的靶基因进行功能富集(GO)和通路富集(KEGG)分析,解释其可能的分子机制。
对象与方法1.研究对象:GEO数据库(http://www.ncbi.nlm.nih.gov/geo)中GSE67140数据集[9],芯片平台为Affymetrix miRNA Array,包括177例肝癌组织与细胞样本,其中81例血管侵袭阳性组织样本,91例血管侵袭阴性组织样本,2例侵袭能力强的肝癌细胞样本(SNU423、SNU449),3例侵袭能力弱的肝癌细胞样本(HepG2、Hep3B、SNU398)。TCGA数据库(http://cancergenome.nih.gov/publications/publicationguidelines)中肝癌样本有373例,测序平台为Illumina HiSeq 2000 RNA Sequencing,其中随访资料清晰的样本有362例。
2.研究方法:
(1)差异microRNA分析:运用R语言limma软件进行差异分析。数据经过标准化与log2转换。在差异分析中,对差异倍数的绝对值>2倍,P<0.05的microRNA进行下一步研究。
(2)microRNA的靶基因预测:利用microT-CDS平台对microRNA的靶基因进行预测,以P<0.05为差异临界值[15]。
(3)靶基因GO和KEGG分析:利用mirPath平台对靶基因进行GO与KEGG分析,以P<0.05,MicroT<0.8为差异临界值[16]。
3.统计学分析:运用R语言及SPSS 22.0软件进行统计分析。应用Kaplan-Meier法进行单因素生存分析,采用对数秩检验(log-rank检验);Cox比例风险回归模型进行多因素生存分析。所有统计检验均采用双侧检验,P<0.05为差异有统计学意义。
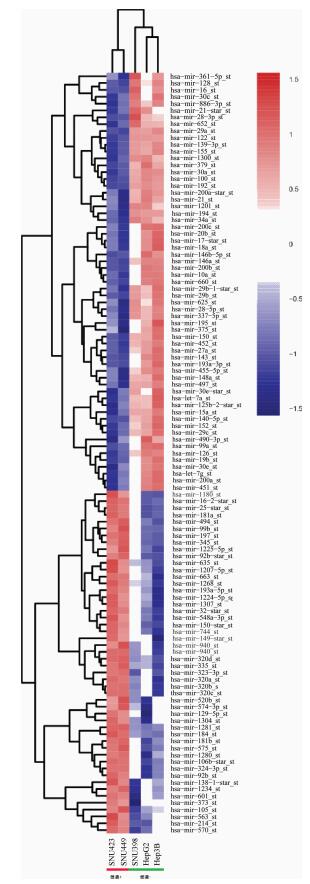
结果1.肝癌侵袭microRNA的表达:分析GEO数据库中GSE67140数据集,其中包括2例侵袭能力强的肝癌细胞样本(SNU423、SNU449),3例侵袭能力弱的肝癌细胞样本(HepG2、Hep3B、SNU398)。如图 1所示,共有110个差异表达的microRNA(认为差异倍数>2倍,P<0.05的microRNA有统计学意义),其中在SNU423、SNU449细胞系中表达上调的有50个(包括原始文献报道的microRNA-494),在HepG2、Hep3B、SNU398细胞系中表达上调的有60个。从中挑选hsa-mir-1180、hsa-mir-149、hsa-mir-744、hsa-mir-940,在172例临床组织样本表达谱中验证(81例血管侵袭阳性组织样本,91例血管侵袭阴性组织样本),发现上述4条均在血管侵袭阳性组织样本中表达上调,在血管侵袭阴性样本中表达下调,与细胞水平表达一致(P<0.05)。见表 1、图 2。
|
| 注:SNU423、SNU449为侵袭能力强的肝癌细胞系,HepG2、Hep3B、SNU398为侵袭能力弱的肝癌细胞系 图 1 microRNA在不同肝癌细胞系侵袭能力的表达 |

|
| 注:血管侵袭(+)样本81例,血管侵袭(-)样本91例 图 2 在172例组织样本中4种microRNA差异表达情况 |
2. 4种差异表达microRNA的生存分析:为分析4种microRNA的预后,选取TCGA数据库中有生存与表达谱数据的362例肝癌病例进行生存分析(表 2、图 3)。其中男性247例(68.2%),女性115例(31.8%);平均年龄(59.434±13.218)岁;白种人187例(51.7%),非白种人175例(48.3%);肝癌组织学分级1级54例(14.9%),2级175例(48.3%),3级120例(33.1%),4级13例(3.7%)。其中125例(34.5%)在随访结束时死亡。Kaplan-Meier法单因素生存分析结果显示,hsa-mir-1180、hsa-mir-149、hsa-mir-744和hsa-mir-940高表达组的生存时间均低于其低表达组生存时间(P<0.05)。为进一步研究microRNA表达对肝癌病例预后的影响,运用Cox比例风险回归模型进行多因素分析,模型中调整年龄、性别、肝癌分级3个变量,结果显示hsa-mir-1180(HR=1.623,95%CI:1.114~2.365,P=0.012),hsa-mir-149(HR=2.400,95%CI:1.639~3.514,P=0.000),hsa-mir-744(HR=1.679,95%CI:1.161~2.427,P=0.006),hsa-mir-940(HR=1.704,95%CI:1.188~2.443,P=0.004)是肝癌病例预后独立危险因素。

|
| 图 3 microRNA高表达组和低表达组的Kaplan-Meier生存分析 |
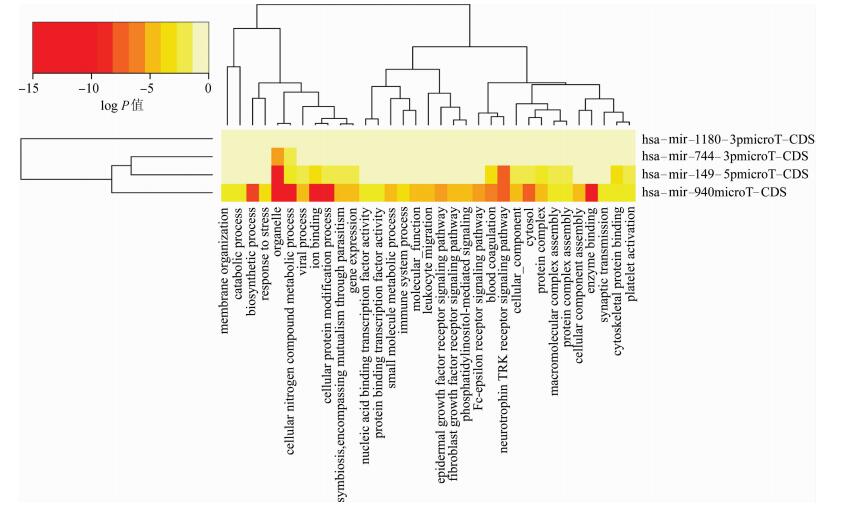
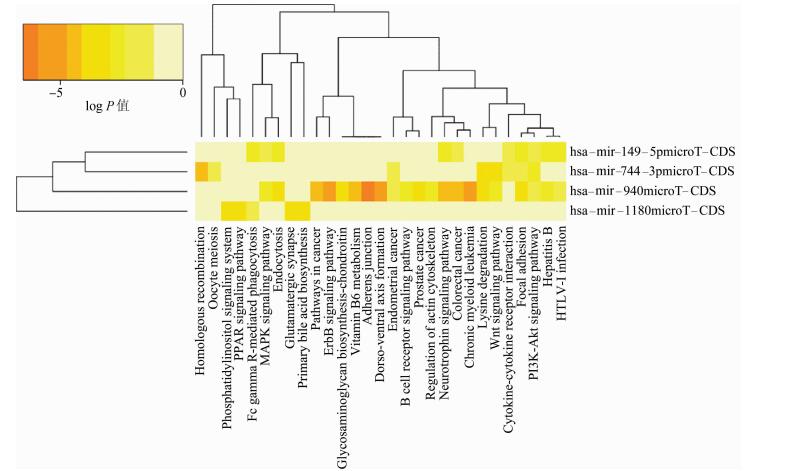
3. 4种差异表达microRNA靶基因的GO与KEGG分析:通过对microT-CDS平台预测出来的靶基因进行GO与KEGG分析(图 4、5),靶基因的功能主要与免疫反应、应激、细胞骨架构建相关,主要富集在细胞黏附、连接、细胞间因子-受体反应、免疫反应及Wnt、MAPK、ErbB、PI3K-Akt信号通路上,其中细胞黏附、连接等信号通路是与侵袭功能密切相关的通路。
|
| 图 4 microRNA靶基因的GO分析 |
|
| 图 5 microRNA靶基因的KEGG分析 |
microRNA是一类长度为21~23 nt的内源性小分子RNA,具有高度保守性、时序性和组织特异性。microRNA在肝癌的发生发展中起到十分重要的作用。已往研究认为,hsa-mir-199在肝癌组织中特异性低表达,与不良预后及复发相关[11]。Xu等[17]发现hsa-mir-101可作用于EZH2,以增强对化疗药物的敏感性,抑制肝癌细胞的增殖、迁移与侵袭。血清中的hsa-mir-16、hsa-mir-21、let-7f、hsa-mir-139可作为肝癌的预后复发的指标[10, 12]。血管侵袭是影响肝癌复发转移的主要原因之一,也是预后危险因素。目前仅有Chuang等[9]运用芯片技术发现hsa-mir-494通过TET1抑制DNA甲基化,从而抑制多种血管侵袭抑制相关的microRNA表达,进而发生肿瘤血管侵袭。进一步分析发现hsa-mir-1180、hsa-mir-149、hsa-mir-744、hsa-mir-940在侵袭阳性细胞和组织中表达上调(logFC>1,P<0.05),生存分析显示4种microRNA为肝癌预后独立危险因素,高表达与患者生存期缩短显著相关(P<0.05)。
Tan等[18]发现hsa-mir-1180在肝癌组织中表达下调,可通过NF-κB信号通路的激活抑制肝癌细胞的凋亡促进增殖;动物实验证明抑制hsa-mir-1180表达后,小鼠的移植瘤变小;临床预后分析证实高表达组hsa-mir-1180的生存率低于其低表达组的生存率,与本文结果一致。Lin等[13]发现hsa-mir-149在肝癌组织中表达下调,过表达可以抑制肝癌细胞的生长,但在裸鼠成瘤动物模型中过表达可以增强肝癌细胞对化疗、放疗的敏感性;Chen等[19]发现在前列腺癌的侧群细胞中,下调的hsa-mir-149会促进凋亡,抑制其增殖能力,本研究发现hsa-mir-149血管侵袭阳性的组织中表达上调,与文献一致,分析其原因可能因为microRNA具有组织细胞特异性,在不同细胞中表达功能不同。Li等[20]发现高表达的hsa-mir-774与喉鳞状细胞癌的淋巴结转移相关,抑制其表达后降低细胞侵袭、迁移能力,动物实验表明也减少了肺转移结节的数目;Miyamae等[21]在94例胰腺癌患者与68例健康对照研究中,发现血浆中hsa-mir-744可以作胰腺癌患者筛查的指标和预后的危险因素,本文结果显示hsa-mir-744在肝癌组织及细胞中表达升高,未在肝癌患者血浆中验证。有研究认为表达的hsa-mir-940可以促进胰腺癌细胞的增殖、迁移与侵袭能力[22]。Liu等[23]在胃癌研究中发现hsa-mir-940通过抑制ZNF24的功能,促进胃癌细胞迁移和侵袭,本研究发现hsa-mir-940在侵袭能力强的肝癌细胞系中表达上调,提示高表达的hsa-mir-940可能在肝癌中发挥促进侵袭的作用。
目前运用组学手段通过临床样本对血管侵袭标志物的研究较少,大多是细胞水平分子生物学功能的基础研究,本文的创新性在于通过2个数据库的大样本数据(539例),从临床的角度分析了4种microRNA的表达与预后情况,为今后肝癌预后分析提供了新的标志物。
本研究存在不足。如microRNA主要通过对靶基因沉默进行调控,靶基因则通过编码蛋白质发挥功能,由于原始芯片只包含microRNA表达谱的数据,因而研究中未分析靶基因和蛋白的表达情况。今后将通过PCR与免疫组化方法检测靶基因和蛋白表达情况,构建microRNA-mRNA-protein调控网络,进一步解释其分子机制。
综上所述,本研究利用GEO与TCGA数据库初步发现hsa-mir-1180、hsa-mir-149、hsa-mir-744、hsa-mir-940在肝癌血管侵袭中高表达,并影响肝癌预后,可作为肝癌预后的生物标志物。
利益冲突: 无
| [1] | El-Serag HB. Hepatocellular carcinoma[J]. N Engl J Med, 2011, 365(12): 1118–1127. DOI:10.1056/NEJMra1001683 |
| [2] | Jemal A, Bray F, Center MM, et al. Global cancer statistics[J]. CA Cancer J Clin, 2011, 61(2): 69–90. DOI:10.3322/caac.20107 |
| [3] |
陈建国, 陈万青, 张思维, 等.
中国2003-2007年肝癌发病率与死亡率分析[J]. 中华流行病学杂志, 2012, 33(6): 547–553.
Chen JG, Chen WQ, Zhang SW, et al. Incidence and mortality of Hver cancer in China:an analysis on data from the National Registration System between 2003 and 2007[J]. Chin J Epidemiol, 2012, 33(6): 547–553. DOI:10.3760/cma.j.issn.0254-6450.2012.06.001 |
| [4] |
王黎君, 殷鹏, 刘韫宁, 等.
1990年与2013年中国人群肝癌疾病负担研究[J]. 中华流行病学杂志, 2016, 37(6): 758–762.
Wang LJ, Yin P, Liu YN, et al. Disease burden of liver cancer in the Chinese population, in 1990 and 2013[J]. Chin J Epidemiol, 2016, 37(6): 758–762. DOI:10.3760/cma.j.issn.0254-6450.2016.06.003 |
| [5] | Hall AJ, Wild CP. Liver cancer in low and middle income countries[J]. BMJ, 2003, 326(7397): 994–995. DOI:10.1136/bmj.326.7397.994 |
| [6] | Pang RWC, Joh JW, Johnson PJ, et al. Biology of hepatocellular carcinoma[J]. Ann Surg Oncol, 2008, 15(4): 962–971. DOI:10.1245/s10434-007-9730-z |
| [7] | He L, Thomson JM, Hemann MT, et al. A microRNA polycistron as a potential human oncogene[J]. Nature, 2005, 435(7043): 828–833. DOI:10.1038/nature03552 |
| [8] | Bai SM, Nasser MW, Wang B, et al. MicroRNA-122 inhibits tumorigenic properties of hepatocellular carcinoma cells and sensitizes these cells to sorafenib[J]. J Biol Chem, 2009, 284(46): 32015–32027. DOI:10.1074/jbc.M109.016774 |
| [9] | Chuang KH, Whitney-Miller CL, Chu CY, et al. MicroRNA-494 is a master epigenetic regulator of multiple invasion-suppressor microRNAs by targeting ten eleven translocation 1 in invasive human hepatocellular carcinoma tumors[J]. Hepatology, 2015, 62(2): 466–480. DOI:10.1002/hep.27816 |
| [10] | Ge W, Yu DC, Li QG, et al. Expression of serum miR-16, let-7f, and miR-21 in patients with hepatocellular carcinoma and their clinical significances[J]. Clin Lab, 2014, 60(3): 427–434. |
| [11] | Hou J, Lin L, Zhou WP, et al. Identification of miRNomes in human liver and hepatocellular carcinoma reveals miR-199a/b-3p as therapeutic target for hepatocellular carcinoma[J]. Cancer Cell, 2011, 19(2): 232–243. DOI:10.1016/j.ccr.2011.01.001 |
| [12] | Li T, Yin JK, Yuan LJ, et al. Downregulation of microRNA-139 is associated with hepatocellular carcinoma risk and short-term survival[J]. Oncol Rep, 2014, 31(4): 1699–1706. DOI:10.3892/or.2014.3032 |
| [13] | Lin L, Zhang YD, Chen ZY, et al. The clinicopathological significance of miR-149 and PARP-2 in hepatocellular carcinoma and their roles in chemo/radiotherapy[J]. Tumour Biol, 2016, 37(9): 12339–12346. DOI:10.1007/s13277-016-5106-y |
| [14] | Tsai WC, Hsu PW, Lai TC, et al. MicroRNA-122, a tumor suppressor microRNA that regulates intrahepatic metastasis of hepatocellular carcinoma[J]. Hepatology, 2009, 49(5): 1571–1582. DOI:10.1002/hep.22806 |
| [15] | Paraskevopoulou MD, Georgakilas G, Kostoulas N, et al. DIANA-microT web server v5.0:service integration into miRNA functional analysis workflows[J]. Nucleic Acids Res, 2013, 41(Suppl 1): W169–173. DOI:10.1093/nar/gkt393 |
| [16] | Vlachos IS, Zagganas K, Paraskevopoulou MD, et al. DIANA-miRPath v3.0:deciphering microRNA function with experimental support[J]. Nucleic Acids Res, 2015, 43(Suppl1): W460–466. DOI:10.1093/nar/gkv403 |
| [17] | Xu L, Beckebaum S, Iacob S, et al. MicroRNA-101 inhibits human hepatocellular carcinoma progression through EZH2 downregulation and increased cytostatic drug sensitivity[J]. J Hepatol, 2014, 60(3): 590–598. DOI:10.1016/j.jhep.2013.10.028 |
| [18] | Tan GS, Wu LW, Tan JF, et al. MiR-1180 promotes apoptotic resistance to human hepatocellular carcinoma via activation of NF-kappaB signaling pathway[J]. Sci Rep, 2016, 6: 22328. DOI:10.1038/srep22328 |
| [19] | Chen YT, Zhao JH, Luo Y, et al. Downregulated expression of miRNA-149 promotes apoptosis in side population cells sorted from the TSU prostate cancer cell line[J]. Oncol Rep, 2016, 36(5): 2587–2600. DOI:10.3892/or.2016.5047 |
| [20] | Li JZ, Gao W, Lei WB, et al. MicroRNA 744-3p promotes MMP-9-mediated metastasis by simultaneously suppressing PDCD4 and PTEN in laryngeal squamous cell carcinoma[J]. Oncotarget, 2016, 7(36): 58218–58233. DOI:10.18632/oncotarget.11280 |
| [21] | Miyamae M, Komatsu S, Ichikawa D, et al. Plasma microRNA profiles:identification of miR-744 as a novel diagnostic and prognostic biomarker in pancreatic cancer[J]. Br J Cancer, 2015, 113(10): 1467–1476. DOI:10.1038/bjc.2015.366 |
| [22] | Yang HW, Liu GH, Liu YQ, et al. Over-expression of micro RNA-940 promotes cell proliferation by targeting GSK3β and sFRP1 in human pancreatic carcinoma[J]. Biomed Pharmacother, 2016, 83: 593–601. DOI:10.1016/j.biopha.2016.06.057 |
| [23] | Liu XY, Ge XX, Zhang Z, et al. MicroRNA-940 promotes tumor cell invasion and metastasis by downregulating ZNF24 in gastric cancer[J]. Oncotarget, 2015, 6(28): 25418–25428. DOI:10.18632/oncotarget.4456 |
 2017, Vol. 38
2017, Vol. 38




