文章信息
- 王郁, 吴红岩, 赵希畅, 朱渭萍, 万千, 陆一涵, 姜庆五.
- Wang Yu, Wu Hongyan, Zhao Xichang, Zhu Weiping, Wan Qian, Lu Yihan, Jiang Qingwu.
- 上海市浦东新区注射吸毒者HCV的进化分析与正选择位点研究
- Evolutionary characteristics and positive selection site of hepatitis C virus isolated in intravenous drug users in Pudong new district, Shanghai
- 中华流行病学杂志, 2017, 38(3): 378-383
- Chinese journal of Epidemiology, 2017, 38(3): 378-383
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.03.021
-
文章历史
收稿日期: 2016-09-06
2. 200120 上海市浦东新区疾病预防控制中心;
3. 200120 上海, 复旦大学浦东预防医学研究院
2. Shanghai Pudong New District Center for Disease Control and Prevention, Shanghai 200120, China;
3. Fudan University Pudong Institute of Preventive Medicine, Shanghai 200120, China
HCV感染是一个全球性公共卫生问题。据估计,全球约有1.7亿~2.1亿人(约占世界人口的3%)感染HCV,每年约有300万~400万新发病例[1],约有35万人死于HCV感染相关疾病[2]。吸毒者是HCV感染的高危人群[3-4];研究表明,我国静脉注射吸毒人群由于共用注射器具等高危行为,其HCV感染率可能达50.4%[4]。根据中国国家禁毒委员会办公室《2015中国禁毒报告》显示,全国累计登记吸毒人员达295.5万名。调查显示,我国注射吸毒人数呈上升趋势[5],随之导致的HCV感染及其向一般人群传播的风险不容忽视。
HCV是单股正链RNA病毒,其基因组包含NS5B等多个编码区域[6]。截至目前,HCV可划分为7个明确的基因型与67个亚型,各个基因型/亚型在不同地区的分布差异较大[7]。基因1、2、3型在世界范围均有分布,其他各型则局限于某些地区;4型主要分布在非洲和中东地区;5型和6型则主要流行于南非和东南亚国家[8];7型为最新在中非国家分离得到[9]。既往研究显示,HCV基因型/亚型与丙型病毒性肝炎(丙肝)急性感染期发展、疾病严重程度、转归与预后、药物治疗效果等均密切相关[10-12]。此外,HCV基因序列上某些关键位点的变异也与药物治疗效果密切相关。例如,NS5B基因编码的蛋白具有RNA依赖的RNA聚合酶活性(RNA-dependent RNA polymerase,RdRp),是目前治疗HCV感染的药物包括直接抗病毒药物(Direct-acting antiviral,DAA)等的重要结合靶位[6]。本研究通过分离上海市浦东新区注射吸毒者的HCV NS5B片段,并结合同期当地的志愿献血者、MSM、法定报告丙肝病例中分离获得的序列,分析HCV主要基因型病毒种群的增长演化过程;同时研究注射吸毒者HCV病毒株的正选择位点,为控制丙肝疫情和预测评价DAA治疗效果提供基础数据。
材料与方法1.研究对象:本研究于2013年1月至2014年12月在上海市浦东新区美沙酮门诊开展,研究设计经上海市浦东新区CDC医学伦理审查委员会审核并通过。研究对象纳入标准:年龄满18周岁;自愿接受美沙酮替代治疗的注射吸毒者;未接受过丙肝治疗;知情同意。该门诊人数维持在200~300人,加上注射吸毒者较为敏感,因此采用方便抽样,共纳入研究对象200例,其中男性166例、女性34例,平均年龄(42.9±9.1)岁,采集其血液标本并按常规方法分离血清。
2. HCV RNA检测:首先采用病毒RNA提取试剂盒(北京天根生化科技有限公司),按操作说明从140 μl血清中提取HCV RNA,并溶解于60 μl无RNase双蒸水中,保存于-80 ℃备用。然后采用反转录试剂盒(大连宝生物工程有限公司),按说明将RNA反转录成cDNA,后采用巢式PCR方法,扩增HCV RNA NS5B区域377-nt片段(参考H77株8256-8632 nt,GenBank登录号AF009606)。扩增引物和PCR程序设置条件均参考既往文献[13]。阳性PCR产物割胶、纯化后进行测序(上海生工生物工程股份有限公司)。每份PCR产物均双向测序以获得准确的核苷酸序列。
3.进化分析:为纳入更多的基因序列以满足后续分析需求,本课题组将该地区2013-2014年采集的志愿献血者、MSM,以及2008-2015年采集的法定报告丙肝病例中分离获得的HCV NS5B 377-nt片段,亦纳入分析;上述三类人群与注射吸毒者研究对象相互独立。利用Mega v6.0软件比对扩增的核苷酸序列,计算主要基因型分离序列的同源性程度。同时,根据MUSCLE密码子比对法编译为125-aa氨基酸序列[14],结合jModelTest运算结果和BEAST软件的内设模型来选择核苷酸替代模型,分子钟模型则依据Bayesian因子来选择,然后采用BEAST v1.8.3软件包进行分析。该软件包是基于Bayesian统计推断与MCMC算法开发的基因序列进化分析软件,其利用序列的分离时间及其包含的基因信息来探索病毒的进化起源和种群演变过程[15]。在BEAST软件中设定各分离序列的采样时间,并设定MCMC链反应为1×109次,每1 000次取样。BEAST运算结果使用Tracer v1.5软件分析,计算当地流行的主要基因型的平均进化速率并构建其病毒种群的增长演变过程。核苷酸进化速率是以核苷酸的替代速率来表示,即每年每个位置的核苷酸替换数[16]。
4.选择压力分析:计算非同义-同义替代速率比值(ω=dN/dS)是评价选择压力的一个常用方法。当ω>1时,表明该位点受到正选择压力作用,同时该密码子被称为正选择位点;ω=1、ω<1则分别说明该位点受到中性选择和纯化选择(即负选择)作用[17]。
采用Datamonkey在线软件包(http://www.datamokey.org/)筛选正选择位点[18]。为排除基因片段间重组可能对正选择位点的影响,分别使用Datamonkey内置的GARD算法[19]和RDP v4.72软件[20]验证是否存在重组现象。Datamonkey筛选正选择位点的算法主要包括SLAC(Single likelihood ancestor counting)、FEL(Fixed effects likelihood model)和REL(Random effects likelihood model)3种。其中,SLAC方法运行速度较快,算法较保守,存在错漏选择位点的可能,但亦能防止将中性位点误判为正选择位点,适用于大型数据集(50~150条序列)的分析;REL方法则相反,其依赖的假设较多,易出现假阳性结果,适用于分析小型数据集(5~15条序列);FEL方法则介于两者之间[21],适用于中型或者大型数据集的分析。本研究共有39条吸毒者序列,从各方法的适应条件以及出现假阳性和假阴性结果等方面综合考虑,采用FEL方法,筛选出正选择位点(P<0.1)后参照H77株进行统一定位。
结果1.进化分析:本研究纳入200例注射吸毒者,其中男性166例、女性34例,性别比为4.9 : 1,男性平均年龄(40.7岁±8.9岁)高于女性(37.0岁±9.7岁)(t=2.15,P=0.032)。HCV RNA阳性检出率19.5%(39/200),男性30例,女性9例,平均年龄差异无统计学意义[男性:(43.5±9.5)岁,女性:(38.7±6.2)岁;t=1.80,P>0.05]。此外,男性与女性吸毒者中HCV RNA阳性与阴性者的年龄差异均无统计学意义(P>0.05)。分离获得的HCV NS5B 377-nt片段基因分型分为3a亚型(14份)、3b亚型(13份)、1b亚型(7份)、6a亚型(4份)和6n亚型(1份)(表 1),其中3亚型为主导基因型,占69.2%(27/39)。
结合本课题组分离的上海市浦东新区志愿献血者、MSM、法定报告丙肝病例的HCV NS5B 377-nt片段,4类人群序列共133条(包括1b亚型66条、3a亚型35条、3b亚型32条;其中志愿献血者、MSM序列分离年份为2013-2014年,法定报告丙肝病例序列分离年份为2008-2015年),分别计算注射吸毒者3个主要基因亚型序列(1b、3a、3b)的内部同源性以及与同一亚型的其他3类人群序列的同源性。结果显示,注射吸毒者分离序列与同一亚型的其他人群序列的同源性均较高(表 2)。
对1b、3a、3b亚型的基因序列进行BEAST分析,结果发现各型HCV NS5B 377-nt片段的进化过程均符合“松散的”(relaxed)分子钟模型的对数正态分布(Lognormal distribution),HKY模型是1b亚型的最优碱基替代模型、3a和3b亚型则最符合TN93模型。其进化速率、95%最大后验概率密度区间(the Highest Posterior Density Interval,HPD)、有效抽样数(Effective Sample Size,ESS)见表 3。其中,1b亚型的进化速率最快,3a和3b亚型则较慢。
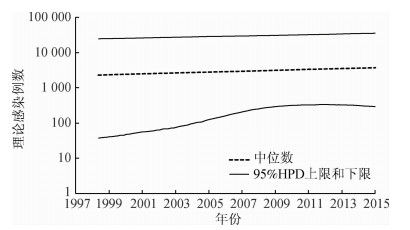
通过BEAST分析,构建Bayesian Skyline Plot分析病毒种群的演变过程(即理论感染人数)的变化趋势,发现:1b亚型的理论感染人数于20世纪90年代开始持续上升,在21世纪初进入平台期(图 1);3a亚型感染人数从20世纪90年代中期也呈上升趋势,但是幅度缓慢(图 2),而3b亚型感染人数的变化幅度较小(图 3)。

|
| 注:纵坐标为底数为10的对数刻度坐标轴 图 1 上海市浦东新区HCV 1b亚型的理论感染人数随时间变化趋势及95% HPD |

|
| 注:纵坐标为底数为10的对数刻度坐标轴 图 2 上海市浦东新区HCV 3a亚型的理论感染人数随时间变化趋势及95% HPD |
|
| 注:纵坐标为底数为10的对数刻度坐标轴 图 3 上海市浦东新区HCV 3b亚型的理论感染人数随时间变化趋势及95%HPD |
2.选择压力分析:GARD算法与RDP软件检测结果均表明本研究纳入的HCV NS5B 377-nt片段不存在重组。编译为125-aa氨基酸片段(对应H77株NS5B区域219-343 aa)后进行选择位点分析,结果显示仅2个(1.6%)氨基酸位点(第324、341位)受到正选择压力的作用,同时另有78个(62.4%)氨基酸位点受到负选择压力的作用(表 3)。该125-aa氨基酸片段中,共包含7个DAA结合位点,分别为第220、225、282、291、316、319、321氨基酸位点[12]。本研究分离获得的注射吸毒者39条片段中,在上述7个位点的突变率仅为2.2%(6/273);其中,4个DAA结合位点(282、291、319、321)均为负选择位点。
既往研究表明,全球范围内注射吸毒人群HCV感染率可能达到60%~80%,在吸毒人数较多的美国、俄罗斯和中国,HCV感染率估计为67%~73%[3]。中国广东省吸毒者中,HCV RNA检出率可达64.7%[22],西北地区则高达78.4%[23],而华东地区强制戒毒人群的检出率甚至达到84.8%[24]。本研究结果发现,上海市浦东新区注射吸毒者的HCV RNA检出率为19.5%,较前述研究偏低。这可能与本研究对象来源于相对较为固定的美沙酮门诊人群有关。我国美沙酮门诊自建立以来,已证实可明显降低HIV、HCV感染的风险[25]。同时,本研究也发现该检出率高于本课题组在当地检测的MSM人群(5.5%)和志愿献血者(0.11%)(结果尚未发表),表明注射吸毒者是该地区HCV感染的高危人群。
近年来,分子流行病学研究越来越多地采用生物信息学方法分析病原体的核苷酸/氨基酸序列。其中,BEAST软件包是被广泛使用的一款生物信息学软件,可用于估算病原体的进化速率,分析其流行与传播过程,探讨其种群随时间的演变过程等[26-27]。本研究结果表明,注射吸毒者中分离的HCV 3a、3b亚型多于1b亚型,且检出一定比例的6a和6n亚型,与既往研究结果较为一致[8]。HCV理论感染人数的变化趋势也表明,HCV 1b和3a亚型的感染人数从20世纪90年代开始呈上升趋势,这可能与90年代初期中国出现大规模不规范血液制品输注导致的艾滋病、肝炎的流行有关;既往研究也表明1993-2000年间,我国HCV的感染数量经历了一段快速增长期[26-27]。然而,本研究同时也发现,HCV 1b和3a亚型在2000年之后的理论感染人数依然呈逐渐上升的趋势,这可能是因为HCV慢性感染者基数较大、可作为传染源持续传播病毒,因此短期内感染者(尤其是吸毒人群)数量可能仍将持续上升[26]。通过BEAST软件包估算的NS5B 377-nt片段的进化速率显示,HCV 1b亚型高于3a、3b亚型,表明1b亚型变异速率较快,因此在未来的DAA治疗中针对1b亚型的变异分析可能较为重要。我国HCV感染以1b与2a亚型为主,近年来由于人口流动、注射吸毒人群数量增加等原因,HCV基因型呈现出lb与2a亚型逐渐减少、3型与6型逐年增加的趋势[9];本课题组分离的注射吸毒者HCV序列与其他人群(包括志愿献血者,该人群通常被认为是健康状况良好)分离序列的同源性均较高,也证实某些HCV基因型在高危人群和一般人群中同时存在;考虑到传播来源,提示其可能从高危人群向一般人群传播,这也与既往研究结果一致[28]。本研究结果表明,上海市浦东新区HCV 3a亚型的理论感染人数逐渐增加,但1b亚型的感染数仍保持较高的水平,结合其较高的进化速率,仍须在未来的预防与治疗工作中加强关注。
相对于病毒基因序列的进化分析,正选择位点筛选是研究病毒进化模式的另一个重要方法。Datamonkey作为HyPhy在线软件包,集成了SLAC、FEL、REL等多个算法,已被广泛应用于生物进化分析研究[5, 18, 21, 29]。本研究选择既往研究较多采用的HCV NS5B区域进行分析。针对HCV NS5B 377-nt片段,仅确认2个正选择位点,而负选择位点则较多,这可能是因为HCV NS5B基因相对保守。既往研究也表明,NS5B基因处于负选择压力下,且NS5B区域相对于E2、E1、NS5A等区域,其正选择压力最小[29]。近年来,针对HCV复制周期的DAA药物飞速的发展,但各类DAA在其应用过程中均已产生耐药突变,因此研究可能的耐药位点变异对HCV及其相关疾病的个体化治疗具有重要意义[30]。本研究发现,注射吸毒者在NS5B区域的DAA结合位点的突变率极低,且均不是正选择位点(其中4个为负选择位点),因此可以估计DAA针对NS5B RdRp结合的效果较好。
本研究存在局限性。由于目前DAA药物尚未在国内上市,相关研究主要依据基因序列分析来初步判断治疗效果[12]。由于本研究仅针对NS5B 377-nt片段进行分析,并非NS5B完整基因序列;且样本量相对较小,不同基因型耐药突变位点也存在一定的差异,因此对DAA的结合效果评价有限,未来将扩大样本量并结合不同片段、对HCV各基因型/亚型进行综合分析。此外,HCV存在少数重复感染现象[31-32],本研究直接采用RT-PCR方法扩增的序列可能仅是其感染的优势毒株亚型,而无法考虑混合感染的情况。
本研究结果表明,上海市浦东新区注射吸毒者HCV感染以3a和3b亚型为主导基因型。HCV 1b亚型在各人群中普遍分布,进化速率较快,其导致的感染数可能将持续上升,仍是未来HCV防治的重点。HCV NS5B 377-nt片段虽然存在少数的正选择位点,但针对HCV NS5B RdRp结合的DAA对当地注射吸毒者感染HCV的治疗效果预计较好,同时仍须加强监测。
利益冲突: 无
| [1] | El-Shabrawi MH, Kamal NM. Burden of pediatric hepatitis C[J]. World J Gastroenterol, 2013, 19(44): 7880–7888. DOI:10.3748/wjg.v19.i44.7880 |
| [2] | Perz JF, Armstrong GL, Farrington LA, et al. The contributions of hepatitis B virus and hepatitis C virus infections to cirrhosis and primary liver cancer worldwide[J]. J Hepatol, 2006, 45(4): 529–538. DOI:10.1016/j.jhep.2006.05.013 |
| [3] | Nelson PK, Mathers BM, Cowie B, et al. Global epidemiology of hepatitis B and hepatitis C in people who inject drugs:results of systematic reviews[J]. Lancet, 2011, 378(9791): 571–583. DOI:10.1016/S0140-6736(11)61097-0 |
| [4] |
王昊鹏, 杨静静, 王小昭, 等.
中国大陆吸毒人群HIV、HBV、HCV感染状况及其相关因素的Meta分析[J]. 中华疾病控制杂志, 2010, 14(4): 300–304.
Wang HP, Yang JJ, Deng XZ, et al. HIV/HBV/HCV infection among drug users:a Meta analysis of data collected in Chinese mainland[J]. Chin J Dis Control Prev, 2010, 14(4): 300–304. |
| [5] |
苏木金, 许望纯, 张伟杰.
不同年代9818例海洛因依赖者吸毒方式与并发传染病分析[J]. 中国药物滥用防治杂志, 2006, 12(5): 277–278.
Su MJ, Xu WC, Zhang WJ. Investigation of drug-taking and incidence of infectious disease among 9818 cases of heroin addicts[J]. Chin J Drug Abuse Prevent Treat, 2006, 12(5): 277–278. DOI:10.3969/j.issn.1006-902X.2006.05.011 |
| [6] |
熊瑜琳, 张长江, 王小红.
丙型肝炎病毒基因组结构及功能[J]. 中国生物化学与分子生物学报, 2008, 24(7): 587–592.
Xiong YL, Zhang CJ, Wang XH. Hepatitis C virus genomic structure and function[J]. Chin J Biochem Mol Biol, 2008, 24(7): 587–592. DOI:10.3969/j.issn.1007-7626.2008.07.001 |
| [7] | Smith DB, Bukh J, Kuiken C, et al. Expanded classification of hepatitis C virus into 7 genotypes and 67 subtypes:updated criteria and genotype assignment web resource[J]. Hepatology, 2014, 59(1): 318–327. DOI:10.1002/hep.26744 |
| [8] |
苏迎盈, 刘慧鑫, 汪宁.
中国丙型肝炎病毒基因型分布[J]. 中华流行病学杂志, 2013, 34(1): 80–84.
Su YY, Liu HX, Wang N. Hepatitis C virus genotypes in China:a systematic review[J]. Chin J Epidemiol, 2013, 34(1): 80–84. DOI:10.3760/cma.j.issn.0254-6450.2013.01.019 |
| [9] | Murphy DG, Sablon E, Chamberland J, et al. Hepatitis C virus genotype 7, a new genotype originating from central Africa[J]. J Clin Microbiol, 2015, 53(3): 967–972. DOI:10.1128/JCM.02831-14 |
| [10] | Kobayashi M, Tanaka E, Sodeyama T, et al. The natural course of chronic hepatitis C:a comparison between patients with genotypes 1 and 2 hepatitis C viruses[J]. Hepatology, 1996, 23(4): 695–699. DOI:10.1053/jhep.1996.v23.pm0008666319 |
| [11] |
乔斌, 汪明, 袁乐永, 等.
丙型肝炎病毒基因型与慢性丙型肝炎患者病情严重程度之间关系的研究[J]. 检验医学与临床, 2015(1): 4–5.
Qiao B, Wang M, Yuan LY, et al. Relationship between HCV genotypes and severity degree of patients with hepatitis C[J]. Lab Med Clin, 2015(1): 4–5. DOI:10.3969/j.issn.1672-9455.2015.01.002 |
| [12] | Cuypers L, Li GD, Libin P, et al. Genetic diversity and selective pressure in hepatitis C virus genotypes 1-6:Significance for direct-acting antiviral treatment and drug resistance[J]. Viruses, 2015, 7(9): 5018–5039. DOI:10.3390/v7092857 |
| [13] | Pybus OG, Barnes E, Taggart R, et al. Genetic history of hepatitis C virus in East Asia[J]. J Virol, 2009, 83(2): 1071–1082. DOI:10.1128/JVI.01501-08 |
| [14] | Tamura K, Stecher G, Peterson D, et al. MEGA6:Molecular Evolutionary Genetics Analysis version 6.0[J]. Mol Biol Evol, 2013, 30(12): 2725–2729. DOI:10.1093/molbev/mst197 |
| [15] | Drummond AJ, Rambaut A. BEAST:Bayesian evolutionary analysis by sampling trees[J]. BMC Evol Biol, 2007, 7: 214. DOI:10.1186/1471-2148-7-214 |
| [16] |
陈建华.
分子进化的速率问题[J]. 自然杂志, 1988, 11(4): 243–247.
Chen JH. The rate of molecular evolution[J]. Chin J Nat, 1988, 11(4): 243–247. |
| [17] | Choisy M, Woelk CH, Guégan JF, et al. Comparative study of adaptive molecular evolution in different human immunodeficiency virus groups and subtypes[J]. J Virol, 2004, 78(4): 1962–1970. DOI:10.1128/JVI.78.4.1962-1970.2004 |
| [18] | Pond SL, Frost SD. Datamonkey:rapid detection of selective pressure on individual sites of codon alignments[J]. Bioinformatics, 2005, 21(10): 2531–2533. DOI:10.1093/bioinformatics/bti320 |
| [19] | Kosakovsky PSL, Posada D, Gravenor MB, et al. GARD:a genetic algorithm for recombination detection[J]. Bioinformatics, 2006, 22(24): 3096–3098. DOI:10.1093/bioinformatics/btl474 |
| [20] | Martin DP, Lemey P, Lott M, et al. RDP3:a flexible and fast computer program for analyzing recombination[J]. Bioinformatics, 2010, 26(19): 2462–2463. DOI:10.1093/bioinformatics/btq467 |
| [21] |
陈晓霞, 苏应娟, 王艇.
细鳞苔科psbA基因的适应性进化分析[J]. 西北植物学报, 2010, 30(8): 1534–1544.
Chen XX, Su YJ, Wang T. Adaptive evolution analysis of the psbA gene in lejeuneaceae[J]. Acta Bot Boreali Occident Sin, 2010, 30(8): 1534–1544. |
| [22] | Fu YS, Qin WB, Cao H, et al. HCV 6a prevalence in Guangdong province had the origin from Vietnam and recent dissemination to other regions of China:phylogeographic analyses[J]. PLoS One, 2012, 7(1): e28006. DOI:10.1371/journal.pone.0028006 |
| [23] |
张春涛, 李秀华, 宋爱京, 等.
吸毒人群中HIV/HCV核酸和抗体关系的分析[J]. 中国病毒学, 2005, 20(6): 600–602.
Zhang CT, Li XH, Song AJ, et al. The relationship of the nucleic acid and antibody of HIV/HCV among drug users[J]. Virol Sin, 2005, 20(6): 600–602. |
| [24] |
龚建明, 杨静静, 王昊鹏, 等.
南京地区吸毒人群丙型肝炎的常见基因型分型研究[J]. 东南国防医药, 2009, 11(3): 199–201.
Gong JM, Yang JJ, Wang HP, et al. Genotype study on hepatitis C virus among drug users in Nanjing, China[J]. Mil Med J Southeast Chin, 2009, 11(3): 199–201. |
| [25] |
曹晓斌, 吴尊友, 庞琳, 等.
中国首批8个社区美沙酮维持治疗门诊5年治疗效果分析[J]. 中华流行病学杂志, 2012, 33(9): 879–882.
Cao XB, Wu ZY, Pang L, et al. Evaluation on the long-term effectiveness among the first set eight methadone maintenance treatment clinics in China[J]. Chin J Epidemiol, 2012, 33(9): 879–882. DOI:10.3760/cma.j.issn.0254-6450.2012.09.001 |
| [26] |
童汪霞, 朱建芸, 雷姿颖, 等.
HCV在中国流行多样性和独特生长史的分析[J]. 中华实验和临床病毒学杂志, 2014, 28(5): 336–338.
Tong WX, Zhu JY, Lei ZY, et al. Great variety and unique growth history of HCV in China[J]. Chin J Exp Clin Virol, 2014, 28(5): 336–338. DOI:10.3760/cma.j.issn.1003-9279.2014.05.006 |
| [27] | Lu L, Tong WX, Gu L, et al. The current Hepatitis C virus prevalence in China may have resulted mainly from an officially encouraged plasma campaign in the 1990s:a coalescence inference with genetic sequences[J]. J Virol, 2013, 87(22): 12041–12050. DOI:10.1128/JVI.01773-13 |
| [28] | Lu L, Wang M, Xia WJ, et al. Migration patterns of hepatitis C virus in China characterized for five major subtypes based on samples from 411 volunteer blood donors from 17 provinces and municipalities[J]. J Virol, 2014, 88(13): 7120–7129. DOI:10.1128/JVI.00414-14 |
| [29] |
王真真, 丁娜, 刘俊, 等.
丙型肝炎病毒5个基因的正选择位点研究[J]. 生物学杂志, 2012, 29(6): 23–28.
Wang ZZ, Ding N, Liu J, et al. Assay on the positively selected sites of five genes of hepatitis C virus[J]. J Biol, 2012, 29(6): 23–28. DOI:10.3969/j.issn.2095-1736.2012.06.023 |
| [30] |
聂爱华.
丙型肝炎病毒NS5B RNA聚合酶抑制剂研究进展[J]. 国际药学研究杂志, 2012, 39(2): 89–103.
Nie AH. Recent advances in HCV NS5B RNA-dependent RNA polymerase inhibitors[J]. J Int Pharm Res, 2012, 39(2): 89–103. DOI:10.3969/j.issn.1674-0440.2012.02.001 |
| [31] |
裴德恩, 彭劲松, 刘满清, 等.
武汉市美沙酮门诊治疗者丙型肝炎病毒混合基因型感染的研究[J]. 中华流行病学杂志, 2009, 30(2): 207–208.
Pei DE, Peng JS, Liu MQ, et al. A study of mix-infections with differerent genotypes of hepatitis C virus in patients from a methadone maintenance clinic in Wuhan[J]. Chin J Epidemiol, 2009, 30(2): 207–208. DOI:10.3760/cma.j.issn.0254-6450.2009.02.030 |
| [32] |
常静霞, 孟运运, 吴亚光, 等.
南京地区218例丙型肝炎患者HCV基因分型[J]. 中华实验和临床感染病杂志:电子版, 2012, 6(5): 5–7.
Chang JX, Meng YY, Wu YG, et al. Analysis on the genotyping of 218 patients with hepatitis C virus infection in Nanjing[J]. Chin J Experiment Clin Infect Dis:Electron Ed, 2012, 6(5): 5–7. DOI:10.3877/cma.j.issn.1674-1358.2012.05.002 |
 2017, Vol. 38
2017, Vol. 38






