文章信息
- 张冰, 洪丽娟, 段广才, 梁文娟, 杨海燕, 郗园林 .
- Zhang Bing, Hong Lijuan, Duan Guangcai, Liang Wenjuan, Yang Haiyan, Xi Yuanlin .
- 4株志贺菌无抗生素压力下连续传代90次的耐药表型及CRISPR/Cas系统变化
- Changes of resistant phenotype and CRISPR/Cas system of four Shigella strains passaged for 90 times without antibiotics
- 中华流行病学杂志, 2017, 38(2): 235-239
- CHINESE JOURNAL OF EPIDEMIOLOGY, 2017, 38(2): 235-239
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.02.020
-
文章历史
收稿日期: 2016-08-01
2. 453003 新乡医学院分子诊断与医学检验技术河南省协同创新中心
2. Henan Innovition Center of Molecular Diagnosis and Laboratory Medicine, Xinxiang Medical University, Xinxiang 453003, China
由志贺菌引起的细菌性痢疾是一类具有高度传染性且危害较严重的肠道传染病,抗生素滥用导致志贺菌不断产生多重耐药,为临床有效治疗细菌性痢疾带来挑战。成簇规律间隔短回文重复序列(CRISPR)和CRISPR相关蛋白(Cas)组成的CRISPR/Cas系统广泛存在于原核生物中,可有效处理外源遗传物质、限制耐药基因等水平转移[1-2]。CRISPR作为该系统重要的结构基础,主要由重复序列(repeat)和间隔序列(spacer)构成的R-S单元排列组成。其中重复序列长28~47 bp,在同一个CRISPR位点高度保守;间隔序列长17~84 bp,高度可变[3-4],是外源遗传物质入侵的“记录器”,使得CRISPR基因座存在不同噬菌体或质粒的信息,是细菌发挥防护作用的基础[5]。CRISPR/Cas系统的进化速度十分缓慢[6],CRISPR序列可在103~105年间保持不变[7],但也有研究发现,CRISPR位点3′端的重复和间隔序列构成的R-S单元经常被删除[8-9]。本研究在无抗生素压力下连续传代90次,检测志贺菌耐药表型及CRISPR/Cas系统的变化,以期为志贺菌耐药及CRISPR/Cas系统的研究提供资料。
材料与方法1. 材料:
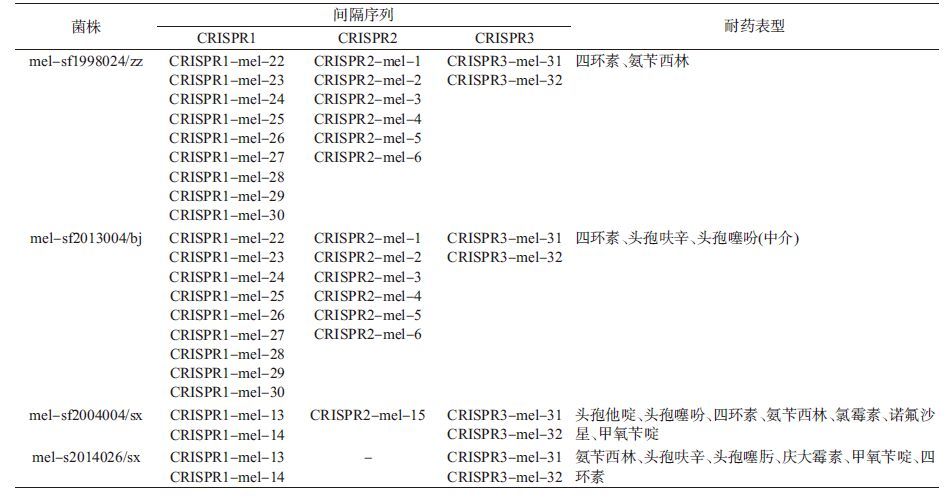
(1)菌株:临床分离的4株志贺菌mel-sf1998024/zz、mel-sf2013004/bj、mel-sf2004004/sx、mel-s2014 026/sx间隔序列信息及耐药表型信息见表 1。大肠埃希菌ATCC25922购自中国普通微生物菌种管理保藏中心。
(2)培养基及主要试剂:胰蛋白胨、酵母浸粉购自英国 Oxoid 公司,M-H(A)培养基购自北京索莱宝科技有限公司。细菌基因组DNA 提取试剂盒购于上海莱枫生物科技有限公司。PCR相关试剂:2×Taq PCR Master Mix master mix、ddH2O、MgCl2购自上海生工生物工程有限公司。氨苄西林、头孢氨苄、头孢噻肟、头孢呋辛、四环素、氯霉素、链霉素、诺氟沙星、甲氧苄啶等抗菌药物标准品购自中国药品生物制品检定所。
2. 研究方法:
(1)多耐药志贺菌的无抗生素压力连续传代:分别挑取4株耐药谱不同的志贺菌单克隆菌落,经LB肉汤过夜增菌,以LB肉汤1 ∶ 500倍稀释,37 ℃、200 r/min振荡培养6~7 h,至A600值为0.08~0.12后,继续以LB肉汤1 ∶ 500倍稀释后再次按照上述条件进行培养,3次/d,连续培养30 d。
(2)志贺菌药物敏感性测定:对无抗生素压力连续传代前后的志贺菌使用琼脂稀释法测定不同抗生素的最低抑菌浓度(MIC),以大肠埃希菌标准株ATCC25922作为质控菌株。根据美国临床实验室标准化协会(CLSI)2012 年出版的《抗微生物药物敏感性试验执行标准》第22版信息增刊M100-S22 进行结果判断。
(3)志贺菌CRISPR/Cas系统CRISPR位点的扩增:引物序列见表 2。PCR 扩增体系为25 μl:2×Taq PCR Master Mix 12.5 μl,ddH2O 8.5 μl,上、下游引物各1 μl(10 mmol/L),模板DNA 2 μl,反应条件:94 ℃预变性5 min;94 ℃变性 60 s,退火温度见表 2;45 s,72 ℃延伸1 kb/min,共32 个循环;最后72 ℃延伸10 min。产物送上海生工生物工程有限公司进行测序。
结 果1. 4株志贺菌无抗生素压力传代90次后的耐药表型:传代后,mel-sf1998024/zz对氨苄西林、头孢氨苄、头孢噻肟、氯霉素的敏感性均增加;mel-s2014026/sx对诺氟沙星和甲氧苄啶的敏感性增加;mel-sf2004004/sx对氨苄西林、头孢呋辛、头孢噻肟、氯霉素、甲氧苄啶的敏感性均增加;mel-sf2013004/bj对氯霉素的敏感性增加。且氯霉素对mel-sf1998024/zz、mel-sf2004004/sx、mel- sf2013004/bj的MIC变化为4倍。4株细菌对链霉素、四环素的敏感性未见改变。见表 3。
2. CRISPR/Cas系统的变化:经无抗生素压力传代90次后,2株志贺菌mel-sf1998024/zz和mel- sf2013004/bj相同的CRISPR3位点的第2条间隔序列与第2条重复序列(即3′的R-S单元)均丢失,见图 1、2。根据前期CRISPRTarget的分析发现,丢失的间隔序列的匹配基因编码产物是Cas1蛋白[11]。其余菌株的CRISPR位点均未见改变。

|
| 注: 方框中为丢失的重复和间隔序列; *为相同序列 图 1 志贺菌mel-sf1998024/zz无抗生素压力传代90次前、后CRISPR3位点比对 |

|
| 注: 方框中为丢失的重复和间隔序列; *为相同序列 图 2 志贺菌mel-sf2013004/bj无抗生素压力传代90次前、后CRISPR3位点比对 |
本研究中,4株志贺菌在无抗生素压力连续传代90次后,对不同抗生素的敏感性有不同程度的增加,但对链霉素和四环素的耐药情况均未见改变。朱静媛等[12]对10株耐药谱不同、携带不同整合子的志贺菌进行无抗生素压力传代30次后,其中1株志贺菌由整合子介导的耐药性经过多次传代培养后丢失。1株多耐药单核细胞增生李斯特菌的研究显示,在细菌由质粒介导的氯霉素、红霉素、四环素抗性在连续传代400次后仍然有很高的稳定性,并没有在频繁传代过程中丢失[13]。在1株粪肠球菌的无抗生素选择压力传代600次后,由质粒介导的四环素抗性也十分稳定[14]。本研究中,不同志贺菌对某些抗生素的耐药性降低,可能是因为无抗生素压力的存在情况下,细菌生存不会受到威胁,会降低甚至丢失对某些抗生素的耐药性,但是对链霉素和四环素的耐药性比较稳定。
经多次传代后,志贺菌mel-s2014026/sx和mel-sf2004004/sx的CRISPR/Cas相关位点均未见变化。生物信息学的分析结果表明,相对于其他快速进化的CRISPR/Cas系统,I-E型的CRISPR/Cas系统进化速度十分缓慢[6],CRISPR序列可在103~105年间保持不变,且在漫长的进化过程中,CRISPR序列不是逐渐改变,而是罕见地会有间隔序列的变化[7],这与快速连续进化的免疫防御系统功能不符,推测在大肠埃希菌中CRISPR/Cas系统可能无活性。有研究表明,在大肠埃希菌中,相差<25万年的细菌具有相同的CRISPR位点[15],同样地,与细菌CRISPR/Cas系统中间隔序列的多样性和动态性相悖。但是有研究发现,细菌CRISPR位点的重复和间隔序列构成的R-S单元经常被删除[8-9],被删除的序列一般是3′比较保守的序列[9],与之相似,本研究结果显示,传代后的志贺菌mel-sf1998024/C和mel-sf2013004/C的CRISPR3位点发生退化,丢失了3′的重复-间隔序列单元。有人推测这样的删除可能是在CRISPR位点的重复序列间发生了同源重组所致[16]。本课题组前期发现,被丢失的间隔序列匹配的基因是cas1,可能说明CRISPR位点与cas基因存在共进化。间隔序列保留外源遗传物质的信息以应对相同外源物质的再次入侵,重复序列在crRNA识别自身基因与外源基因的过程中发挥重要作用。在细菌的进化过程中,CRISPR/Cas系统可以通过间隔序列的不断增加和删除限制CRISPR位点的大小,限制转录长度,同时有助于细菌更新,以应对外环境的变化。
总之,本研究中获得了耐药性降低且CRISPR3位点发生退化的志贺菌株,为志贺菌的耐药及CRISPR/Cas系统的研究提供基础资料。
利益冲突: 无
| [1] | Bikard D, Hatoum-Aslan A, Mucida D, et al. CRISPR interference can prevent natural transformation and virulence acquisition during in vivo bacterial infection[J]. Cell Host Microbe, 2012, 12(2): 177–186. DOI:10.1016/j.chom.2012.06.003 |
| [2] | Marraffini LA, Sontheimer EJ. CRISPR interference limits horizontal gene transfer in staphylococci by targeting DNA[J]. Science, 2008, 322(5909): 1843–1845. DOI:10.1126/science.1165771 |
| [3] | Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J]. Genome Biol, 2007, 8(4): R61. DOI:10.1186/gb-2007-8-4-r61 |
| [4] | Shah SA, Hansen NR, Garrett RA. Distribution of CRISPR spacer matches in viruses and plasmids of crenarchaeal acidothermophiles and implications for their inhibitory mechanism[J]. Biochem Soc Trans, 2009, 37(Pt 1): 23–28. DOI:10.1042/BST0370023 |
| [5] | Barrangou R, Fremaux C, Deveau H, et al. CRISPR provides acquired resistance against viruses in prokaryotes[J]. Science, 2007, 315(5819): 1709–1712. DOI:10.1126/science.1138140 |
| [6] | Andersson AF, Banfield JF. Virus population dynamics and acquired virus resistance in natural microbial communities[J]. Science, 2008, 320(5879): 1047–1050. DOI:10.1126/science.1157358 |
| [7] | Touchon M, Charpentier S, Clermont O, et al. CRISPR distribution within the Escherichia coli species is not suggestive of immunity-associated diversifying selection[J]. J Bacteriol, 2011, 193(10): 2460–2467. DOI:10.1128/JB.01307-10 |
| [8] | Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinformatics, 2007, 8(1): 172. DOI:10.1186/1471-2105-8-172 |
| [9] | Deveau H, Barrangou R, Garneau JE, et al. Phage response to CRISPR-encoded resistance in Streptococcus thermophilus[J]. J Bacteriol, 2008, 190(4): 1390–1400. DOI:10.1128/JB.01412-07 |
| [10] |
王琳琳. CRISPR/Cas系统与志贺菌耐药的关系[D]. 郑州:郑州大学,2015.
Wang LL. The relationship between CRISPR/Cas system and drug resistance in Shigella[D]. Zhengzhou:Zhengzhou University, 2015. http://cdmd.cnki.com.cn/Article/CDMD-10459-1015302743.htm |
| [11] |
王鹏飞. 志贺菌、大肠杆菌和沙门菌CRISPR结构特征生物信息学分析[D]. 郑州:郑州大学,2015.
Wang PF. Bioinformatics analysis of CRISPR structures and feature in Shigella,Escherichia coli and Salmonella[D]. Zhengzhou:Zhengzhou University,2015. |
| [12] |
朱静媛, 段广才, 范清堂, 等.
志贺菌属整合子的耐药贡献与稳定性研究[J]. 第三军医大学学报, 2011, 33(3): 278–281.
Zhu JY, Duan GC, Fan QT, et al. Drug resistance of Shigella integrons and their stability[J]. Acta Acad Med Milit Tert, 2011, 33(3): 278–281. |
| [13] |
李丽丽, 石磊, 何建华, 等.
一株食源性多重耐药单核细胞增生李斯特菌的耐药性研究[J]. 现代食品科技, 2015, 31(7): 105–110.
Li LL, Shi L, He JH, et al. Antibiotic resistance of foodborne multidrug-resistant Listeria monocytogenes[J]. Mod Food Sci Technol, 2015, 31(7): 105–110. DOI:10.13982/j.mfst.1673-9078.2015.07.018 |
| [14] | Li XH, Alvarez V, Harper WJ, et al. Persistent,toxin-antitoxin system-independent,tetracycline resistance-encoding plasmid from a dairy Enterococcus faecium isolate[J]. Appl Environ Microbiol, 2011, 77(20): 7096–7103. DOI:10.1128/AEM.05168-11 |
| [15] | Touchon M, Rocha EPC. The small,slow and specialized CRISPR and anti-CRISPR of Escherichia and Salmonella[J]. PLoS One, 2010, 5(6): e11126. DOI:10.1371/journal.pone.0011126 |
| [16] | Wilmes P, Simmons SL, Denef VJ, et al. The dynamic genetic repertoire of microbial communities[J]. FEMS Microbiol Rev, 2009, 33(1): 109–132. DOI:10.1111/j.1574-6976.2008.00144.x |
 2017, Vol. 38
2017, Vol. 38