文章信息
- 刘远, 蒋力云, 罗雷, 曹毅敏, 景钦隆, 杨智聪 .
- Liu Yuan, Jiang Liyun, Luo Lei, Cao Yimin, Jing Qinlong, Yang Zhicong .
- 广州市2001-2015年登革病毒2型E基因进化分析
- Phylogenetic analysis of envelope gene of dengue virus serotype 2 in Guangzhou, 2001-2015
- 中华流行病学杂志, 2017, 38(1): 90-95
- Chinese journal of Epidemiology, 2017, 38(1): 90-95
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2017.01.018
-
文章历史
收稿日期: 2016-08-08
2. 510440 广州市疾病预防控制中心病毒免疫部;
3. 510440 广州市疾病预防控制中心传染病预防控制部
2. Virus Department, Guangzhou Center for Disease Control and Prevention, Guangzhou 510440, China;
3. Epidemiology Department, Guangzhou Center for Disease Control and Prevention, Guangzhou 510440, China
登革病毒(dengue virus,DENV)是引起登革热和登革出血热(dengue haemorrhagic fever,DHF)的病原。DENV有3种结构蛋白,分别是衣壳蛋白(C蛋白)、膜蛋白(PrM蛋白和M蛋白)和包膜蛋白(E蛋白)。E蛋白构成病毒颗粒的表面凸起,具有血凝活性,在与宿主细胞融合和诱导产生抗体的过程中发挥重要作用。20世纪以来,登革热在世界各地广泛流行,东南亚和西太平洋地区的登革热病例占全球病例的很大比例[1]。中国登革热流行主要发生在广东、广西、福建、浙江、海南等地区[2-4],其中广东省最为严重。广州市是登革热的重灾区,在2002、2006和2014年出现大规模登革热流行。2001-2011年广东省报告的3 577例登革热病例中,70.90%(2 536例)出现在广州市;2014年广东省登革热流行是广东省有史以来规模最大的,广州市病例占82.63%(37 340例)[5-6]。广州市登革热的流行过程中,4种血清型登革病毒均曾从病例血清中分离到,以1型为主[7]。由于DENV存在抗体依赖的病毒感染增强作用(antibody-dependent enhancement,ADE),不同血清型登革病毒的二次感染更容易引发重症登革热,导致DHF或者登革休克综合征(dengue shock syndrome,DSS)。有研究资料表明,登革2型病毒(DENV2)导致的DHF病例最多,变异率最高[8]。本研究通过对2001-2015年广州市确诊登革热病例的血清标本进行分离培养,获取DENV2毒株,测定E基因全序列,构建系统进化树,并进行分子钟模型分析,从分子水平追踪其可能的传染来源,分析广州市登革热流行特点。
材料与方法1.资料及标本来源:疫情数据来自中国疾病预防控制系统疾病监测报告信息;患者资料来自广州市CDC疫情处理档案。血清标本取自广州市各医院、区(县)CDC送检的登革热确诊病例血清标本。
2.诊断标准:参照《登革热诊断标准》(WS 216-2008)[9]。确诊病例为有登革热临床症状,伴白细胞减少、血小板减少或者束臂实验阳性者,且同时具备以下一项:血清DENV IgM和(或)IgG阳性,或在过去14 d内到过登革热疫区,或在登革热暴发点范围内,并具备以下一项:RT-PCR检测核酸片段阳性,或病毒分离阳性,或恢复期血清IgG抗体水平较急性期有4倍以上升高。
3.核酸检测:使用江苏硕世生物科技有限公司生产的登革热1~4型核酸检测试剂(荧光PCR法),对确诊病例标本进行荧光检测及分型,并将阳性标本留待下一步病毒分离。
4.病毒分离:将荧光PCR阳性标本的血清按照1 : 20、1 : 30、1 : 40、1 : 50的梯度稀释,取100 μl稀释血清接种到C6/36细胞,吸附1 h后,弃去上清,加入含2%小牛血清的1640培养基,置28 ℃孵箱中培养7 d后传代,出现细胞病变则判断为阳性,盲传3代无细胞病变则判断为阴性。
5. E基因RT-PCR扩增及序列测定:收集病变细胞上清液,采用QIAamp Viral RNA Mini Kit(德国Qiagen公司)提取DENV RNA,针对E基因设计引物,用TaKaRa One Step RNA PCR Kit[宝生物工程(大连)有限公司]进行RT-PCR扩增,扩增产物进行凝胶电泳,电泳阳性样品用QIAquick Gel Extraction Kit(德国Qiagen公司)进行回收,送英潍捷基(上海)贸易有限公司测序。
RT-PCR反应条件:50 ℃反转录30 min,94 ℃预变性5 min;94 ℃ 30 s、55 ℃ 30 s、72 ℃ 3 min,30个循环;72 ℃延伸7 min。引物序列:DEN750(5′-CAA GAA CCG AAA CGT GGA TG-3′),DEN2639(5′-TGT GGA AGC AAA TAT CAC CTG-3′)。
6. E基因进化树和分子钟模型分析:使用Mega 4.0软件Kimura 2 parameter模型绘制基因系统进化树,步长设为1 000。核苷酸变异率和分子钟使用BEASTv1.8.2.
结果1.疫情概况:2001-2015年广州市共发现登革热确诊病例41 440例,其中本地病例41 114例,占总病例数的99.22%;输入病例326例,占总病例数的0.78%。输入国家中,病例数前3位的分别为泰国(54例)、马来西亚(42例)和印度尼西亚(37例),分别占输入病例总数的16.57%、12.89%和11.35%。
2.核酸检测结果:2001-2015年广州市用荧光PCR共检测确诊病例标本3 403份,DENV总阳性数为517份,阳性率为15.20%。分型结果,1型阳性356份,2型阳性69份,3型阳性58份,4型阳性34份。
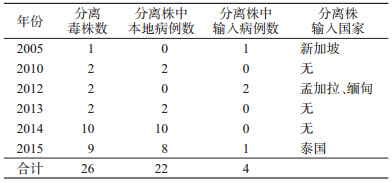
3.病毒分离结果:将69份荧光PCR检测DENV2阳性标本进行病毒培养,共分离到DENV2毒株26株(37.69%),其中22株为从本地病例标本中分离得到,占总毒株数的84.62%,4株为从输入标本中分离得到,占总毒株数的15.38%。输入国家为新加坡、孟加拉、缅甸和泰国,见表 1。
4.病毒E基因特征:
(1)核苷酸及氨基酸同源性比较:对分离的26株DENV2,针对E基因扩增,并进行序列分析,核苷酸相似率为91.1%~100.0%,氨基酸相似率为97.7%~100.0%。中国广州市分离株与其他国家分离到的DENV2如JN568253(澳大利亚2010年)、KT232050(尼泊尔2013年)、JF967976(印度尼西亚2009年)、JN030329(新加坡2010年)、KJ175134(泰国2014年)、KT175129(越南2014年)以及KJ470751(缅甸2013年)有很高的相似性,核苷酸同源性为91.1%~100.0%,氨基酸同源性为97.2%~100.0%。
(2)基因组序列系统进化树分析:中国广州市分离到的26株DENV2毒株中,按照同一年份序列相同的毒株只保留1条序列的原则,最终获得16条不同序列。将16条分离株序列上传至National Center for Biotechnology Information(NCBI),与39条NCBI上的标准株序列及其他国家历年来获得的代表株序列,进行分析比对,经Mega 4.0软件,构建进化树。分析发现,从基因型分类上看,大部分广州市分离到的DENV2都属于全球型,仅2012年分离到的1株病毒属于亚洲Ⅰ型,见图 1。

|
| 图 1 2001-2015年广州市DENV E基因系统进化树 |
(3)贝叶斯法分子进化钟分析:将16条分离株序列和39条在NCBI上下载的序列用BEASTv1.8.2绘制分子进化钟,选择Lognormal relaxed clock(uncorrelated)进行计算,使用General Time-Reversible(GTR)模型,运行10 000 000步,每1 000步取值一次,并舍去每次运行中的前1 000步[10]。计算出核苷酸变异率为每年每位点7.1×10-4,广州市分离到的全球型DENV2毒株的最近相同祖先(the time to most resent common ancestor,tMRCA)出现在大概63年前(即1952年),全球型在46年前(1969年)和35年前(1980年)分化出2个亚型。亚洲Ⅰ型DENV2毒株的tMRCA出现在大概64年前(即1951年)。而DENV2所有基因型的tMRCA出现在353年前(即1662年),见图 2。

|
| 图 2 2001-2015年广州市登革热病毒E基因贝叶斯法分子进化钟 |
2001-2015年,广州市每年都有登革热病例报告,以DENV1流行为主[7]。2010年前,仅在2005年分离到DENV2。2012年开始每年都能分离到DENV2。在2014年广州市出现的登革热历史性大流行中,广州市南沙区出现了大范围的DENV2流行[11],本研究分离到的10株DENV2毒株均分离自南沙区确诊病例血清标本,这是广州市有纪录以来出现的首次由DENV2引起的登革热流行。
近年来,由于与周边国家的贸易往来、旅游交流增多,人口流动逐年增加,中国广州市发生输入性登革热疫情的风险不容忽视[12]。基因进化分析结果显示(图 1),2001-2015年我国广州市分离到的毒株与东南亚地区有密切的关系,其中4株DENV2输入病例来自新加坡、孟加拉、缅甸和泰国(表 1)。相关流行病学调查结果也证实了中国广州市登革热病例输入地以东南亚地区为主。随着经济发展,东南亚地区旅游成为出国旅游热点,这为登革热的输入和传播提供了有利条件。同时,登革热在周边国家的流行和传播历史已久,血清型和基因型多样[13-15],对我国广州市登革热的输入造成了极大压力。一旦发生多种血清型登革热的输入,很可能造成重症登革热病例的暴发。增强出国人员的传染病防控意识,加强国境检验检疫,及时发现和诊断登革热病例,对登革热的防控有重要意义。
广州市分离到的DENV2毒株可进一步分为全球型和亚洲Ⅰ型2种基因型,并以全球型为主。由图 2可以看出,全球型和亚洲Ⅰ型的tMRCA出现在63年前(1952年)和64年前(1951年),提示大概在这段时期,全球型和亚洲Ⅰ型在亚洲地区乃至世界范围的传播和扩散。这段时期经历了第二次世界大战,有可能是由于战争和人口的迁移造成登革热的传播和扩散[16]。DENV2所有基因型的tMRCA出现在353年前(1662年),说明大约在这个时期,DENV2出现了基因型的分化。
2001-2015年广州市流行的DENV2主要是全球型。全球型在46年前和35年前进一步出现亚型的分化,广州市分离到的DENV2全球型毒株也明显地归入这2个亚型,反映了流行于广州市的全球型DENV2可能存在2个不同输入来源。
2012年广州市分离出亚洲Ⅰ型毒株,与FJ196851(中国,1998年)、KF955399(柬埔寨,2008年)、JF968045(泰国,2010年)和KJ470751(缅甸,2013年)有很近的亲缘关系,并以99.0%的步长支持率处于进化树的同一分支上(图 1)。这可能展示了该毒株在相邻国家的传播和进化过程。同一血清型不同基因型的DENV重复感染导致的登革热重症迄今未见报道。然而,基因型的增多会使得本地DENV变异率增大,并导致自然选择出更有感染力的毒株[17],这将使得DENV2在广州市的流行传播更为复杂。
DENV与流感病毒、HIV和冠状病毒等RNA病毒类似,其聚合酶都是RNA依赖的RNA聚合酶,病毒的变异率和进化速度都较含其他聚合酶的病毒高[18]。BEASTv1.8.2计算出我国广州市DENV2的平均变异率为7.1×10-4每年每位点,与1956-2005年印度DENV2的平均变异率6.5×10-4每年每位点和2011-2014年的平均变异率7.7×10-4每年每位点相比[16, 19],基本持平。
利益冲突: 无
| [1] | WHO. Dengue and severe dengue[EB/OL].[2016-08-06] 2016. http://www.who.int/mediacentre/factsheets/fs117/en/ |
| [2] |
熊益权, 陈清.
1978-2014年我国登革热的流行病学分析[J]. 南方医科大学学报 , 2014(12) : 1822–1825.
Xiong YQ, Chen Q. Epidemiology of dengue fever in China since 1978[J]. J South Med Univ , 2014(12) : 1822–1825. DOI:10.3969/j.issn.1673-4254.2014.12.24 |
| [3] |
魏墩宏, 韩雪玲, 胡淑芳.
登革热和登革出血热的流行病学研究现状[J]. 医学动物防制 , 2014, 30(4) : 405–408.
Wei DH, Han XL, Hu SF. Epidemiology research progress of dengue fever and dengue hemorrhagic fever[J]. J Med Pest Control , 2014, 30(4) : 405–408. DOI:10.7629/yxdwfz201404017 |
| [4] |
杜建伟, 潘先海.
中国登革热流行概况与流行特征[J]. 中华流行病学杂志 , 2010, 31(12) : 1429–1433.
Du JW, Pan XH. Prevalent status and features of dengue fever in China[J]. Chin J Epidemiol , 2010, 31(12) : 1429–1433. DOI:10.3760/cma.j.issn.0254-6450.2010.12.025 |
| [5] |
樊景春, 林华亮, 吴海霞, 等.
广东省2006-2011年登革热时空分布特征[J]. 中国媒介生物学及控制杂志 , 2013, 24(5) : 389–391.
Fan JC, Lin HL, Wu HX, et al. Spatial and temporal distribution characteristics of dengue fever in Guangdong province, China during 2006-2011[J]. Chin J Vector Biol Control , 2013, 24(5) : 389–391. DOI:10.11853/j.issn.1003.4692.2013.05.003 |
| [6] |
郭汝宁, 彭志强, 宋铁, 等.
广东省1990-2012年登革热和基孔肯雅热感染状况及流行风险分析[J]. 中华流行病学杂志 , 2014, 35(2) : 167–169.
Guo RN, Peng ZQ, Song T, et al. Current infection status and epidemic risk analysis of Dengue fever and Chikungunya in Guangdong province, from 1990 to 2012[J]. Chin J Epidemiol , 2014, 35(2) : 167–169. DOI:10.3760/cma.j.issn.0254-6450.2014.02.014 |
| [7] | Jiang LY, Wu XW, Wu YJ, et al. Molecular epidemiological and virological study of dengue virus infections in Guangzhou, China, during 2001-2010[J]. Virol J , 2013, 10 : 4. DOI:10.1186/1743-422X-10-4 |
| [8] | Rico-Hesse R, Harrison LM, Salas RA, et al. Origins of dengue type 2 viruses associated with increased pathogenicity in the Americas[J]. Virology , 1997, 230(2) : 244–251. DOI:10.1006/viro.1997.8504 |
| [9] | 中华人民共和国国家卫生和计划生育委员会.登革热诊断标准[EB/OL]. (2008-02-28)[2016-08-01]. http://www.nhfpc.gov.cn/zwgkzt/s9491/200802/38819.shtml. |
| [10] | Drummond AJ, Rambaut A. BEAST:Bayesian evolutionary analysis by sampling trees[J]. BMC Evol Biol , 2007, 7 : 214. DOI:10.1186/1471-2148-7-214 |
| [11] |
张志忠, 张海洋, 陆少艳, 等.
2014年广州市南沙区登革热疫情分析[J]. 热带医学杂志 , 2016, 160(1) : 108–110.
Zhang ZZ, Zhang HY, Lu SY, et al. Analysis on epidemic situation of dengue fever in Nansha district of Guangzhou in 2014[J]. J Trop Med , 2016, 160(1) : 108–110. |
| [12] |
段雪飞, 王晓杰, 郭晓笛, 等.
14例输入性登革热病例的流行病学及临床特点分析[J]. 中华实验和临床感染病杂志:电子版 , 2011, 5(4) : 443–448.
Duan XF, Wang XJ, Guo XD, et al. Clinical and epidemiological features of 14 imported cases with dengue fever[J]. Chin J Exp Clin Infect Dis:Elect Ver , 2011, 5(4) : 443–448. DOI:10.3877/cma.j.issn.1674-1358.2011.04.008 |
| [13] | Wilder-Smith A, Yoksan S, Earnest A, et al. Serological evidence for the co-circulation of multiple dengue virus serotypes in Singapore[J]. Epidemiol Infect , 2005, 133(4) : 667–671. DOI:10.1017/S0950268805003821 |
| [14] | Mohd-Zaki AH, Brett J, Ismail E, et al. Epidemiology of dengue disease in Malaysia (2000-2012):a systematic literature review[J]. PLoS Negl Trop Dis , 2014, 8(11) : e3159. DOI:10.1371/journal.pntd.0003159 |
| [15] | Limkittikul K, Brett J, L'Azou M. Epidemiological trends of dengue disease in Thailand (2000-2011):a systematic literature review[J]. PLoS Negl Trop Dis , 2014, 8(11) : e3241. DOI:10.1371/journal.pntd.0003241 |
| [16] | Kumar SR, Patil JA, Cecilia D, et al. Evolution, dispersal and replacement of American genotype dengue type 2 viruses in India (1956-2005):selection pressure and molecular clock analyses[J]. J Gen Virol , 2010, 91(Pt 3) : 707–720. DOI:10.1099/vir.0.017954-0 |
| [17] | Holmes EC, Burch SS. The causes and consequences of genetic variation in dengue virus[J]. Trends Microbiol , 2000, 8(2) : 74–77. DOI:10.1016/s0966-842X(99)01669-8 |
| [18] | Mendez JA, Usme-Ciro JA, Domingo C, et al. Phylogenetic history demonstrates two different lineages of dengue type 1 virus in Colombia[J]. Virol J , 2010, 7 : 226. DOI:10.1186/1743-422X-7-226 |
| [19] | Afreen N, Naqvi IH, Broor S, et al. Evolutionary analysis of dengue serotype 2 viruses using phylogenetic and bayesian methods from New Delhi, India[J]. PLoS Negl Trop Dis , 2016, 10(3) : e4511. DOI:10.1371/journal.pntd.0004511 |
 2017, Vol. 38
2017, Vol. 38