文章信息
- 高莹, 高文静, 曹卫华 .
- Gao Ying, Gao Wenjing, Cao Weihua .
- 儿童青少年肥胖与DNA甲基化关联研究进展
- Current status and progress in studies on the association between obesity and DNA methylation among children and adolescents
- 中华流行病学杂志, 2016, 37(8): 1169-1174
- CHINESE JOURNAL OF EPIDEMIOLOGY, 2016, 37(8): 1169-1174
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.08.023
-
文章历史
收稿日期: 2016-01-21
肥胖影响因素研究一般分为遗传和环境两方面。遗传研究显示,肥胖存在家族聚集性,遗传对于肥胖有一定的影响[1-4]。环境因素多样且在生命不同时期所起作用可能不同[5-7]。基因-环境之间存在交互作用[8],单纯遗传或环境因素研究不能完全解释肥胖发生机制。而表观遗传学研究有助于从生物学机制上解释遗传因素、环境因素、基因-环境因素交互作用对于肥胖发生的影响,为肥胖病因学探究开拓思路[9]。
一、 表观遗传学研究表观遗传是指DNA序列未发生变化但基因功能发生了可遗传的改变。目前DNA甲基化(DNA methylation)即甲基化是最常见、研究最多的表观遗传现象[10],是指在DNA甲基化转移酶(DNA methyltransferase,DNMT)的作用下,在基因组CpG二核苷酸的胞嘧啶5’碳位以共价键结合一个甲基基团。正常情况下,人类基因组中非编码区序列的CpG二核苷酸相对稀少,并且总是处于甲基化的稳定状态;而位于基因的启动子区或第一个外显子区的大小约100~1 000 bp且富含CpG二核苷酸的CpG岛则总是处于未甲基化的不稳定状态[11]。
甲基化研究设计主要有两种:一是横断面研究;二是纵向研究即生命早期甲基化情况与后期表型关联分析。其研究类型又可分为三种:总体基因甲基化水平关联研究、候选基因位点甲基化关联研究以及全基因组甲基化关联研究。
甲基化测定技术大致分为两种,候选基因/特定位点和全基因组的甲基化分析。前者常用技术有甲基化特异性PCR(methylation-specific PCR,MSP)、亚硫酸氢盐处理后测序(bisulfite sequencing PCR,BSP)、荧光定量法(Methylight)、甲基化敏感性高分辨率溶解曲线(Methylation Specific High Resolution Melting curve analysis,MS-HRM)、联合亚硫酸氢钠的限制性内切酶分析法(COmBined Restriction enzyme Analyses,COBRA)、焦磷酸测序(Pyrosequencing)等。全基因组甲基化测定目前常用芯片法,Illumina公司Infinium Human Methylation27和450芯片分别覆盖27 578和450 000个甲基化位点,后者因为高覆盖度、高通量以及低价格成为目前分析全基因组甲基化最常用的芯片技术[12]。
二、 儿童肥胖甲基化研究动物研究和人体研究均显示,肥胖发生与生命早期环境因素(如宫内营养不良等)相关[13-16],而生命早期环境因素可能通过甲基化增加个体肥胖发生风险[17-18],研究生命早期甲基化有助于发现肥胖高危人群,对于预防和控制成年肥胖具有重要作用。儿童青少年相比成年人群,受生命后期复杂环境影响较少,且较少受到肥胖相关慢性疾病的干扰,对于研究肥胖发生的表观遗传机制具有重要作用。
(一) 儿童肥胖甲基化测定生物组织现有甲基化研究测定所采用的生物组织类型多样。甲基化测定与基因测序不同,甲基化存在组织特异性,不同组织细胞中基因序列相同,但其基因表达不同,甲基化情况也存在差异。某一组织细胞中甲基化情况可能仅反映该种组织的甲基化情况,不能反映其他组织及整个机体甲基化情况[19]。
无论是成年人还是儿童青少年研究,外周血细胞是目前甲基化研究最常用的材料,因其提取相对简单,被广泛应用于大型流行病学调查中。但外周血细胞包含一系列细胞,不同类型血细胞其甲基化信息可能存在差异[20-21],采用血细胞为研究材料时,个体间甲基化差异可能源于血细胞组成成分差异。虽然有研究显示,总体甲基化水平以及大部分位点甲基化情况在不同类型血细胞间高度一致[22-23],但为了得到可靠结果,应先对血细胞中不同类型细胞组成进行校正,以消除细胞组成不同可能带来的影响[24]。
唾液因易于取得,且无侵害性,也是提取生物DNA的常见重要材料之一,常见于儿童青少年研究。有研究显示,唾液提取的DNA与全血提取的DNA甲基化程度高度相关[25],现有部分研究采取唾液代替血液进行甲基化研究。脐带组织(如脐带血细胞)因可反映生命早期(胚胎期)基因情况,可用于疾病筛查,早期基因甲基化情况对于预示某些疾病如肥胖具有重要意义,并且通过比较脐带组织与生命后期血液等其他组织甲基化情况可了解甲基化变化情况,有利于揭示环境因素对于甲基化的影响。
虽然不同类型细胞其甲基化信息可能存在差异,但某些位点(如重复序列ALU)甲基化情况可能在个体不同组织间存在高度一致,有研究认为可以使用这些位点反映机体总体甲基化情况[23]。但甲基化主要影响基因表达,为了明确甲基化对于某种表型(如肥胖)基因表达的影响,应该重点关注与该表型密切相关的组织细胞。肥胖相关甲基化情况应首选与肥胖关系最为密切的脂肪组织。脂肪细胞在能量储存及内分泌中起到重要作用,并且不同类型的脂肪细胞存在于不同位置,可能有不同的作用。测定不同脂肪细胞的甲基化情况可能更有利于揭示肥胖的表观遗传机制。但由于脂肪组织较难取得,现尚未见到采用脂肪组织的儿童青少年肥胖甲基化研究。
(二) 儿童肥胖甲基化研究结果1. 横断面肥胖甲基化研究:
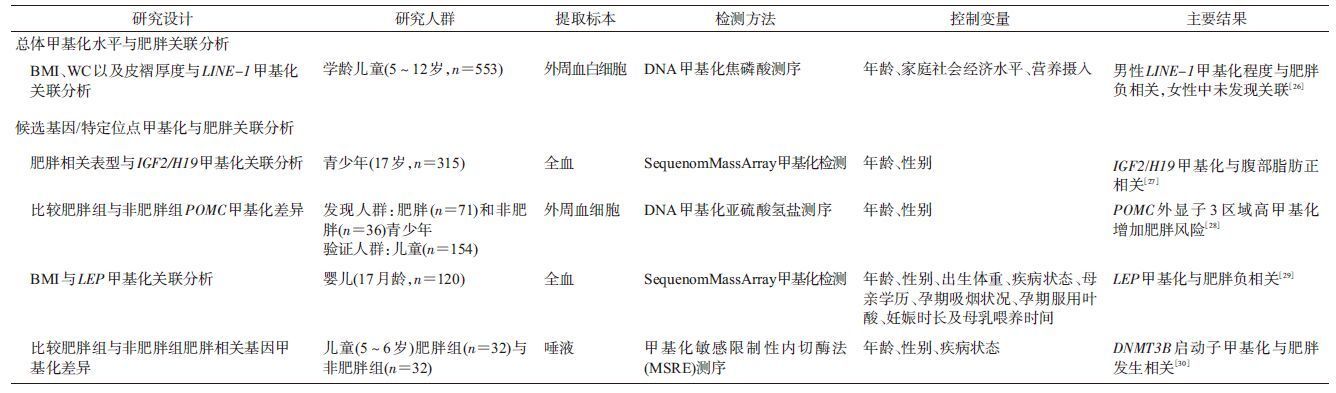
(1)总体甲基化水平关联研究:总体甲基化水平指基因组中所有甲基化的胞嘧啶占总体胞嘧啶的比值,常用5-甲基化胞嘧啶百分比(percentage 5-methylcytosine,%5meC)来表示。低水平的%5meC个体容易发生染色体突变等变异。人体中存在大量的重复序列散在分布于基因组中,比如长散在重复序列(long interspered repeated sequences,LINEs),其甲基化程度常用于表示基因组的%5meC。有国外研究发现,儿童青少年男性总体甲基化水平与肥胖呈负相关,但是在女性中未发现此种关联[26](表 1)。
肥胖总体甲基化研究在成年人中开展相对较儿童青少年多,但研究结果并不统一。有研究显示,总体甲基化水平与血清中HDL-C呈正相关[31],但存在性别差异[32];有育龄妇女研究显示总体甲基化水平与肥胖呈负相关[33];也有部分研究显示校正其他因素后肥胖与总体甲基化水平不存在关联[34-36]。总体甲基化水平易受很多混杂因素的影响,如种群、年龄、环境暴露等,并且现有的研究样本量均有限,可能导致研究结果存在差异。总体甲基化水平对于解释肥胖机制作用有限。
(2)候选基因/特定位点甲基化关联研究:候选基因一般选择以往研究发现的与肥胖发生相关的特定基因位点,现有候选基因研究主要集中在与食欲控制、胰岛素信号通路、能量代谢、免疫和生物节律等相关的基因位点,以及一些印记基因位点,评价这些基因位点甲基化与不同肥胖表型之间的关联(表 1)。特定位点甲基化对基因表达的影响取决于该位点所在的基因序列位置,通常认为基因非编码区的甲基化与基因低表达有关,而编码区甲基化与基因高表达有关[37]。国外儿童青少年研究显示,POMC外显子、DNMT3B启动子高甲基化、LEP低甲基化与肥胖相关,但并未在其他研究中得到验证[34-36]。IGF2/H19印记区域甲基化与肥胖相关[33],儿童IGF/H19甲基化差异可能源自于亲代影响,有研究显示,父母肥胖影响子代IGF2的甲基化表达[38]。生命早期环境因素(如父母成年期BMI、母亲孕期BMI、孕期宫内营养状况等)可能通过影响个体基因甲基化,从而影响肥胖发生。
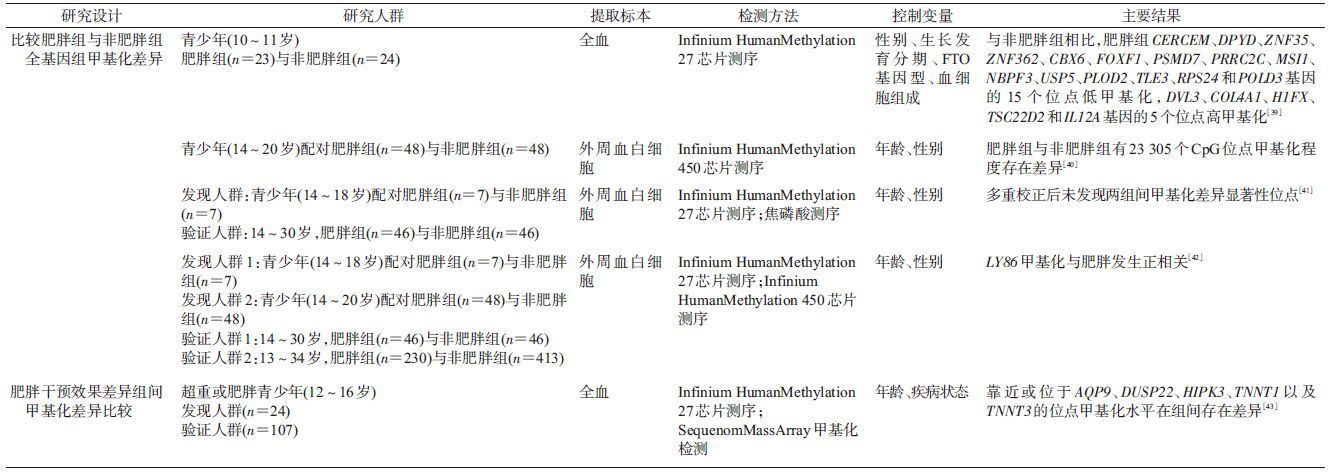
(3)全基因组甲基化关联研究:该技术的发展使甲基化研究不再着重关注于特异位点,而通过对全基因组或大量位点扫描发现疾病相关甲基化位点,针对儿童青少年肥胖国外现有全基因组甲基化研究(表 2)。肥胖与非肥胖组儿童全基因组甲基化比较研究,发现大量甲基化差异位点[39-41],但经多重校正或不同人群验证后,只有少量位点甲基化与肥胖发生相关。不同人群验证后显示LY86甲基化与肥胖发生相关[42]。青少年肥胖干预研究发现,干预效果与两组间某些基因位点(AQP9、DUSP22、HIPK3、TNNT1以及TNNT3)甲基化存在差异,提示某些人群对于肥胖干预措施可能更加敏感,从而降低其肥胖进一步发生或肥胖相关疾病发生风险[43]。
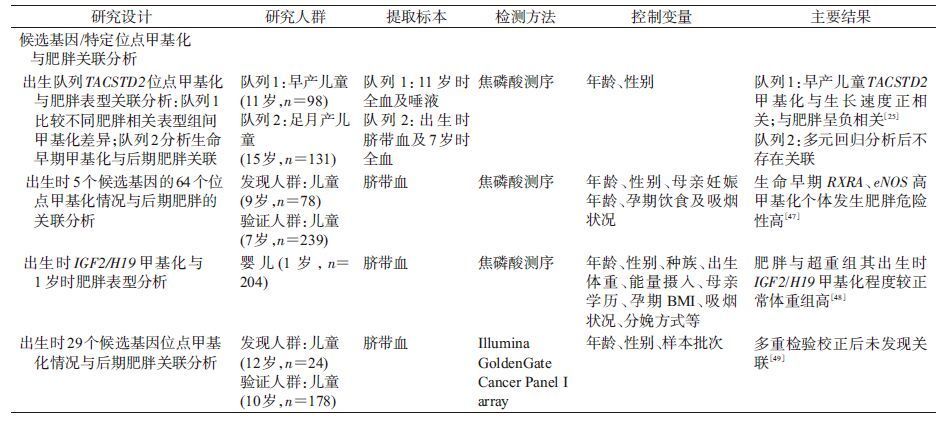
2. 纵向肥胖甲基化研究:胚胎发育期通常被认为是基因发生甲基化的关键时期亲代营养状况、母亲孕期情况及宫内营养状况等影响后代基因组甲基化情况,进而影响后代肥胖发生风险[44-46]。生命早期(胚胎时期)的基因组甲基化情况可能预示后期肥胖发生风险,个体出生时的脐带血细胞因能反映胚胎期基因甲基化情况,现成为研究生命早期基因甲基化情况的重要生物组织。研究发现(表 3),出生时IGF2/H19、RXRα、eNOS及TACSTD2高甲基化与生命后期肥胖发生相关[25, 47-48]。这提示某些位点甲基化建立于生命早期,并且与后期不同时期的肥胖发生相关,这些位点可能将来用于早期预测肥胖发生风险。
现有儿童青少年肥胖甲基化研究发现较多肥胖相关甲基化位点,但只有IGF2/H19及LY86甲基化与肥胖关联在不同研究或人群中得到了验证。IGF2/H19印记区域甲基化与肥胖相关,这一结果在不同儿童青少年研究中得到了验证[33, 48],并且与成年人研究结果一致[50];IGF2/H19与生长发育有关,参与糖、脂肪和蛋白质代谢,其不仅与肥胖发生相关,还与糖尿病等肥胖相关疾病发生存在关联。全基因组甲基化关联分析,经不同人群验证后发现LY86甲基化与肥胖发生相关,LY86基因编码淋巴细胞表面抗原,除参与炎症反应外,还与胰岛素抵抗发生相关,提示其可能参与肥胖相关代谢疾病发生[42]。
遗传因素、环境因素等多种因素均可影响甲基化[51]:首先,不同种族甲基化可能存在差异[52],甲基化可能存在种族效应修饰;其次,甲基化情况可能与时间相关,动物试验与人群流行病学调查均显示甲基化情况随年龄发生改变[53-54],不同年龄儿童甲基化情况存在差异,这种差异可能与遗传或时间积累的环境因素暴露有关;再者,某些位点甲基化情况与其所在位置相关,其甲基化可能受到相邻基因调控,使其甲基化更不稳定,更易发生变异[55]。现有相关研究控制因素存在差异,除常见一般人口学信息(年龄、性别、种族)、社会经济学信息(家庭经济水平、母亲受教育水平)、生活方式(饮食摄入)以及生命早期环境因素(母亲孕期情况)外,个体基因型、生物组织材料(如血细胞构成)差异、甲基化测量方法(如检验批次)等也可能影响研究甲基化水平,造成研究结果存在差异。另外,甲基化差异划分缺乏统一标准,有按照研究人群甲基化中位数或者四分位数作为判定高低标准,或者比较肥胖组与非肥胖组间甲基化程度差异是否具有统计学意义,划分标准的不同也可能造成结果差异。
目前肥胖甲基化研究设计常见为横断面设计,肥胖表型与DNA甲基化情况同时测定,因此无法推断两者因果关联,但早期甲基化情况对于后期肥胖的预测仍有重要作用。现有些研究通过统计学方法推断其因果关联,如孟德尔随机化方法,利用SNP(single nucleotide polymorphism,SNP)数据作为工具变量进行分析[56],但一般较难获得相关SNP数据。纵向研究对于研究肥胖具有重要意义,亲代基因型以及相关表型特征以及个体生命早期生物组织资料的收集不仅有利于确立因果关联,并且有助于观察个体基因对于DNA甲基化的影响及DNA甲基化随时间的变化情况。另外,基因甲基化主要影响基因表达,目前研究普遍仅关注基因甲基化情况,未测定基因表达情况;测定基因甲基化的同时测定基因表达有助于解释甲基化对于基因表达的影响,探索肥胖发生的生物学通路。
儿童肥胖甲基化研究对于揭示肥胖发生机制具有重要意义,是近年来的研究热点。中国人群此方面研究尚缺乏。未来研究可关注中国儿童青少年,在中国人群中寻找肥胖相关甲基化位点,为中国儿童肥胖机制和防治策略提供证据。并且为确定甲基化与肥胖因果关联,应进一步关注于纵向研究,同时进行基因表达测定,探索甲基化对于基因表达的影响,进一步提示肥胖发生机制。
| [1] | Manco M, Dallapiccola B. Genetics of pediatric obesity[J]. Pediatrics , 2012, 130 (1) : 123–133 DOI:10.1542/peds.2011-2717 |
| [2] | Reilly JJ, Armstrong J, Dorosty AR, et al. Early life risk factors for obesity in childhood:cohort study[J]. BMJ , 2005, 330 (7504) : 1357 DOI:10.1136/bmj.38470.670903.E0 |
| [3] | Rankinen T, Zuberi A, Chagnon YC, et al. The human obesity gene map:the 2005 update[J]. Obesity (Silver Spring) , 2006, 14 (4) : 529–644 DOI:10.1038/oby.2006.71 |
| [4] | Elks CE, den Hoed M, Zhao JH, et al. Variability in the heritability of body mass index:a systematic review and meta-regression[J]. Front Endocrinol (Lausanne) , 2012, 3 : 29 DOI:10.3389/fendo.2012.00029 |
| [5] | Tabacchi G, Giammanco S, La Guardia M, et al. A review of the literature and a new classification of the early determinants of childhood obesity:from pregnancy to the first years of life[J]. Nutr Res , 2007, 27 (10) : 587–604 DOI:10.1016/j.nutres.2007.06.001 |
| [6] | Harrison K, Bost KK, McBride BA, et al. Toward a developmental conceptualization of contributors to overweight and obesity in childhood:the six-Cs model[J]. Child Dev Perspect , 2011, 5 (1) : 50–58 DOI:10.1111/j.1750-8606.2010.00150.x |
| [7] | Dhurandhar EJ, Keith SW. The aetiology of obesity beyond eating more and exercising less[J]. Best Pract Res Clin Gastroenterol , 2014, 28 (4) : 533–544 DOI:10.1016/j.bpg.2014.07.001 |
| [8] | Lv D, Zhang DD, Wang H, et al. Genetic variations in SEC16B,MC4R,MAP2K5 and KCTD15 were associated with childhood obesity and interacted with dietary behaviors in Chinese school-age population[J]. Gene , 2015, 560 (2) : 149–155 DOI:10.1016/j.gene.2015.01.054 |
| [9] | Adams J. Obesity,epigenetics,and gene regulation[J]. Nat Educ , 2008, 1 (1) : 128 |
| [10] | Bird A. Perceptions of epigenetics[J]. Nature , 2007, 447 (7143) : 396–398 DOI:10.1038/nature05913 |
| [11] | Jones PA, Takai D. The role of DNA methylation in Mammalian epigenetics[J]. Science , 2001, 293 (5532) : 1068–1070 DOI:10.1126/science.1063852 |
| [12] | Carless MA.Determination of DNA methylation levels using illumina human methylation 450 bead chips//Chellappan SP[M].New York: Springer, 2015: 143–192 |
| [13] | Dubois L, Girard M. Early determinants of overweight at 4.5 years in a population-based longitudinal study[J]. Int J Obes (London) , 2006, 30 (4) : 610–617 DOI:10.1038/sj.ijo.0803141 |
| [14] | Yang Z, Zhao W, Zhang XB, et al. Impact of famine during pregnancy and infancy on health in adulthood[J]. Obes Rev , 2008, 9 : S95–99 DOI:10.1111/j.1467-789X.2007.00447.x |
| [15] | Wang YH, Wang XL, Kong YH, et al. The great Chinese famine leads to shorter and overweight females in Chongqing Chinese population after 50 years[J]. Obesity (Silver Spring) , 2010, 18 (3) : 588–592 DOI:10.1038/oby.2009.296 |
| [16] | Stein AD, Kahn HS, Rundle A, et al. Anthropometric measures in middle age after exposure to famine during gestation:evidence from the Dutch famine[J]. Am J Clin Nutr , 2007, 85 (3) : 869–876 |
| [17] | Seki Y, Williams L, Vuguin PM, et al. Minireview:epigenetic programming of diabetes and obesity:animal models[J]. Endocrinology , 2012, 153 (3) : 1031–1038 DOI:10.1210/en.2011-1805 |
| [18] | Curley JP, Mashoodh R, Champagne FA. Epigenetics and the origins of paternal effects[J]. Horm Behav , 2011, 59 (3) : 306–314 DOI:10.1016/j.yhbeh.2010.06.018 |
| [19] | Marsit CJ, Koestler DC, Christensen BC, et al. DNA methylation array analysis identifies profiles of blood-derived DNA methylation associated with bladder cancer[J]. J Clin Oncol , 2011, 29 (9) : 1133–1139 DOI:10.1200/JCO.2010.31.3577 |
| [20] | Reinius LE, Acevedo N, Joerink M, et al. Differential DNA methylation in purified human blood cells:implications for cell lineage and studies on disease susceptibility[J]. PLoS One , 2012, 7 (7) : e41361 DOI:10.1371/journal.pone.0041361 |
| [21] | Wu HC, Delgado-Cruzata L, Flom JD, et al. Global methylation profiles in DNA from different blood cell types[J]. Epigenetics , 2011, 6 (1) : 76–85 DOI:10.4161/epi.6.1.13391 |
| [22] | Adalsteinsson BT, Gudnason H, Aspelund T, et al. Heterogeneity in white blood cells has potential to confound DNA methylation measurements[J]. PLoS One , 2012, 7 (10) : e46705 DOI:10.1371/journal.pone.0046705 |
| [23] | Talens RP, Boomsma DI, Tobi EW, et al. Variation,patterns,and temporal stability of DNA methylation:considerations for epigenetic epidemiology[J]. FASEB J , 2010, 24 (9) : 3135–3144 DOI:10.1096/fj.09-150490 |
| [24] | Koestler DC, Christensen BC, Karagas MR, et al. Blood-based profiles of DNA methylation predict the underlying distribution of cell types:a validation analysis[J]. Epigenetics , 2013, 8 (8) : 816–826 DOI:10.4161/epi.25430 |
| [25] | Groom A, Potter C, Swan DC, et al. Postnatal growth and DNA methylation are associated with differential gene expression of the TACSTD2 gene and childhood fat mass[J]. Diabetes , 2012, 61 (2) : 391–400 DOI:10.2337/db11-1039 |
| [26] | Perng W, Mora-Plazas M, Marín C, et al. A prospective study of LINE-1 DNA methylation and development of adiposity in school-age children[J]. PLoS One , 2013, 8 (4) : e62587 DOI:10.1371/journal.pone.0062587 |
| [27] | Huang RC, Galati JC, Burrows S, et al. DNA methylation of the IGF2/H19 imprinting control region and adiposity distribution in young adults[J]. Clin Epigenetics , 2012, 4 (1) : 21 DOI:10.1186/1868-7083-4-21 |
| [28] | Kuehnen P, Mischke M, Wiegand S, et al. An Alu element-associated hypermethylation variant of the POMC gene is associated with childhood obesity[J]. PLoS Genet , 2012, 8 (3) : e1002543 DOI:10.1371/journal.pgen.1002543 |
| [29] | Obermann-Borst SA, Eilers PHC, Tobi EW, et al. Duration of breastfeeding and gender are associated with methylation of the LEPTIN gene in very young children[J]. Pediatr Res , 2013, 74 (3) : 344–349 DOI:10.1038/pr.2013.95 |
| [30] | Gardner KR, Sapienza C, Fisher JO. Genetic and epigenetic associations to obesity-related appetite phenotypes among African-American children[J]. Pediatr Obes , 2015, 10 (6) : 476–482 DOI:10.1111/ijpo.12010 |
| [31] | Pearce MS, Mcconnell JC, Potter C, et al. Global LINE-1 DNA methylation is associated with blood glycaemic and lipid profiles[J]. Int J Epidemiol , 2012, 41 (1) : 210–217 DOI:10.1093/ije/dys020 |
| [32] | Cash HL, Mcgarvey ST, Houseman EA, et al. Cardiovascular disease risk factors and DNA methylation at the LINE-1 repeat region in peripheral blood from Samoan Islanders[J]. Epigenetics , 2011, 6 (10) : 1257–1264 DOI:10.4161/epi.6.10.17728 |
| [33] | Piyathilake CJ, Badiga S, Alvarez RD, et al. A lower degree of PBMC L1 methylation is associated with excess body weight and higher HOMA-IR in the presence of lower concentrations of plasma folate[J]. PLoS One , 2013, 8 (1) : e54544 DOI:10.1371/journal.pone.0054544 |
| [34] | Zhang FF, Cardarelli R, Carroll J, et al. Significant differences in global genomic DNA methylation by gender and race/ethnicity in peripheral blood[J]. Epigenetics , 2011, 6 (5) : 623–629 DOI:10.4161/epi.6.5.15335 |
| [35] | Zhu ZZ, Hou LF, Bollati V, et al. Predictors of global methylation levels in blood DNA of healthy subjects:a combined analysis[J]. Int J Epidemiol , 2012, 41 (1) : 126–139 DOI:10.1093/ije/dyq154 |
| [36] | Zhang FF, Santella RM, Wolff M, et al. White blood cell global methylation and IL-6 promoter methylation in association with diet and lifestyle risk factors in a cancer-free population[J]. Epigenetics , 2012, 7 (6) : 606–614 DOI:10.4161/epi.20236 |
| [37] | Jones PA. Functions of DNA methylation:islands,start sites,gene bodies and beyond[J]. Nat Rev Genet , 2012, 13 (7) : 484–492 DOI:10.1038/nrg3230 |
| [38] | Soubry A, Schildkraut JM, Murtha A, et al. Paternal obesity is associated with IGF2 hypomethylation in newborns:results from a Newborn Epigenetics Study (NEST) cohort[J]. BMC Med , 2013, 11 (1) : 29 DOI:10.1186/1741-7015-11-29 |
| [39] | Almén MS, Jacobsson JA, Moschonis G, et al. Genome wide analysis reveals association of a FTO gene variant with epigenetic changes[J]. Genomics , 2012, 99 (3) : 132–137 DOI:10.1016/j.ygeno.2011.12.007 |
| [40] | Xu XJ, Su SY, Barnes VA, et al. A genome-wide methylation study on obesity:differential variability and differential methylation[J]. Epigenetics , 2013, 8 (5) : 522–533 DOI:10.4161/epi.24506 |
| [41] | Wang XL, Zhu HD, Snieder H, et al. Obesity related methylation changes in DNA of peripheral blood leukocytes[J]. BMC Med , 2010, 8 (1) : 87 DOI:10.1186/1741-7015-8-87 |
| [42] | Su SY, Zhu HD, Xu XJ, et al. DNA methylation of the LY86 gene is associated with obesity,insulin resistance,and inflammation[J]. Twin Res Hum Genet , 2014, 17 (3) : 183–191 DOI:10.1017/thg.2014.22 |
| [43] | Moleres A, Campión J, Milagro FI, et al. Differential DNA methylation patterns between high and low responders to a weight loss intervention in overweight or obese adolescents:the EVASYON study[J]. FASEB J , 2013, 27 (6) : 2504–2512 DOI:10.1096/fj.12-215566 |
| [44] | Michels KB, Harris HR, Barault L. Birthweight,maternal weight trajectories and global DNA methylation of LINE-1 repetitive elements[J]. PLoS One , 2011, 6 (9) : e25254 DOI:10.1371/journal.pone.0025254 |
| [45] | Guénard F, Deshaies Y, Cianflone K, et al. Differential methylation in glucoregulatory genes of offspring born before vs. after maternal gastrointestinal bypass surgery[J]. Proc Natl Acad Sci USA , 2013, 110 (28) : 11439–11444 DOI:10.1073/pnas.1216959110 |
| [46] | Guénard F, Tchernof A, Deshaies Y, et al. Methylation and expression of immune and inflammatory genes in the offspring of bariatric bypass surgery patients[J]. J Obes , 2013, 2013 : 492170 DOI:10.1155/2013/492170 |
| [47] | Godfrey KM, Sheppard A, Gluckman PD, et al. Epigenetic gene promoter methylation at birth is associated with child's later adiposity[J]. Diabetes , 2011, 60 (5) : 1528–1534 DOI:10.2337/db10-0979 |
| [48] | Perkins E, Murphy SK, Murtha AP, et al. Insulin-like growth factor 2/H19 methylation at birth and risk of overweight and obesity in children[J]. J Pediatr , 2012, 161 (1) : 31–39 DOI:10.1016/j.jpeds.2012.01.015 |
| [49] | Relton CL, Groom A, S t. DNA methylation patterns in cord blood DNA and body size in childhood[J]. PLoS One , 2012, 7 (3) : e31821 DOI:10.1371/journal.pone.0031821 |
| [50] | Drake AJ, McPherson RC, Godfrey KM, et al. An unbalanced maternal diet in pregnancy associates with offspring epigenetic changes in genes controlling glucocorticoid action and foetal growth[J]. Clin Endocrinol (Oxf) , 2012, 77 (6) : 808–815 DOI:10.1111/j.1365-2265.2012.04453.x |
| [51] | Ollikainen M, Smith KR, Joo EJH, et al. DNA methylation analysis of multiple tissues from newborn twins reveals both genetic and intrauterine components to variation in the human neonatal epigenome[J]. Hum Mol Genet , 2010, 19 (21) : 4176–4188 DOI:10.1093/hmg/ddq336 |
| [52] | Heyn H, Moran S, Hernando-Herraez I, et al. DNA methylation contributes to natural human variation[J]. Genome Res , 2013, 23 (9) : 1363–1372 DOI:10.1101/gr.154187.112 |
| [53] | Maegawa S, Hinkal G, Kim HS, et al. Widespread and tissue specific age-related DNA methylation changes in mice[J]. Genome Res , 2010, 20 (3) : 332–340 DOI:10.1101/gr.096826.109 |
| [54] | Wong CCY, Caspi A, Williams B, et al. A longitudinal study of epigenetic variation in twins[J]. Epigenetics , 2010, 5 (6) : 516–526 DOI:10.4161/epi.5.6.12226 |
| [55] | Dayeh TA, Olsson AH, Volkov P, et al. Identification of CpG-SNPs associated with type 2 diabetes and differential DNA methylation in human pancreatic islets[J]. Diabetologia , 2013, 56 (5) : 1036–1046 DOI:10.1007/s00125-012-2815-7 |
| [56] | Relton CL, Smith GD. Two-step epigenetic Mendelian randomization:a strategy for establishing the causal role of epigenetic processes in pathways to disease[J]. Int J Epidemiol , 2012, 41 (1) : 161–176 DOI:10.1093/ije/dyr233 |
 2016, Vol. 37
2016, Vol. 37