文章信息
- 宋菁, 胡永华 .
- Song Jing, Hu Yonghua .
- 流行病学展望:医学大数据与精准医疗
- Medical big data and precision medicine: prospects of epidemiology
- 中华流行病学杂志, 2016, 37(8): 1164-1168
- CHINESE JOURNAL OF EPIDEMIOLOGY, 2016, 37(8): 1164-1168
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.08.022
-
文章历史
收稿日期: 2016-03-18
大数据(big data)是由于容量大和结构复杂,无法用常规软件对其进行获取、管理、存储、检索、共享、传输和分析的数据集,具有数据容量(volume)大、数据类型(variety)多、处理速度(velocity)快、数据价值(value)高的特点[1]。自2007年数据密集型科学发现被确立为科学研究的“第四范式”以来[2],2008-2011年Nature、Science等顶尖杂志出版大数据专刊,2014年,Science杂志提出“干试验(dry lab biology)”的概念,主张通过挖掘现有数据资源来进行知识发现[3]。另外,大数据开始被越来越多国家纳入国家计划,例如美国的“大数据研究和发展倡议”、法国的“数字化路线图”、日本的“创建最尖端IT国家宣言”和我国的“促进大数据发展行动纲要”等。至此,大数据不仅是科学研究的结果,而逐渐成为重要的科学研究方法和国家战略资源。
精准医疗是指考虑到个体间基因、环境和生活方式等差异而进行疾病预防和治疗的新型医疗模式[4]。与传统临床医学以症状主导(symptom-oriented)的诊疗方式相比,精准医疗模式综合考虑遗传、环境、生活方式等多个因素,充分考虑到个体间差异和疾病异质性,具有典型的可预测性、可预防性、个性化和参与式的“P4医学”特点[5-6]。作为一门与数据息息相关的学科,流行病学无疑将受到精准医疗和大数据热潮的影响。如何整合、挖掘和利用现有的大数据资源,为未来精准医疗的实践提供理论研究支持,将成为今后流行病学领域的一个新热点。
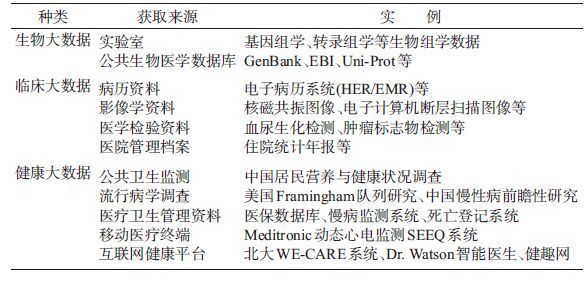
一、 医学大数据与精准医疗医学大数据(medical big data)泛指所有与医疗和生命健康相关的大数据[7]。根据来源,医学大数据又可以粗略分为生物大数据、临床大数据和健康大数据。生物大数据主要是关于生物标本和基因测序信息的数据,组学大数据是其中的重要内容[7]。与过去的分子生物学研究相比,组学研究(omics study)使基础研究由碎片连接为整体,数据容量大、动态性强、复杂性高、异质性明显[2]。临床大数据是产生于医院常规临床诊治、科研和管理过程的数据,包括门/急诊记录、住院记录、影像记录、实验室记录、用药记录、手术记录、随访记录和医疗保险数据等,具有数据量庞大、产生速度快、数据结构复杂和价值密度低等典型大数据的特征[7]。据估计,目前一家大型综合医院平均每年可产生665 TB数据量,远超过美国国会图书馆的网络数据总和。因此,临床医学大数据将成为未来大数据战略的重要资源[8]。健康大数据是来自于专门设计的基于大量人群的医学研究或疾病监测,如全国营养学和健康调查、出生缺陷监测研究、传染病及肿瘤登记报告等数据[7]。
与过去基于症状的直觉医学和现在基于模仿的循证医学不同,精准医疗依据亚人群研究,通过流行病学设计和统计学方法建立起涵盖家族史、症状、体征、实验室检查结果、遗传检测结果和分子标志物检测结果等多维变量的诊断或预测模型,是基于算法的循证医学,旨在实现对风险的精准预测、对疾病的精准诊断和分类、对药物的精准应用、对疗效的精准评估和对预后的精准预测。精准医疗研究需要对各种大数据资源整合、重组、集成和转化(表 1)。例如电子病历(EHR)的标准化互用、信息资源库数据挖掘技术、健康管理信息系统的研究和远程医疗技术和区域医疗信息平台的等信息学和大数据技术[9]。通过整合不同来源和内容的医学大数据并合理挖掘,可以开展组学研究及不同组学间的关联研究[10-11],快速识别生物标志物和研发药物[12-13],快速筛检未知病原和发现可疑致病微生物[14-15],开展传染性疾病和慢性非传染性疾病的实时监测[16-17]与健康管理[18],为精准医疗提供了数据资源和技术支持。
大数据的优势在于大范围寻找流行病学研究中潜在的关联,利用机器学习算法对大数据挖掘的结果进行合成、转化和管理,最终促进流行病学研究的效率,帮助我们深入了解疾病的病因和结局,为药物研发人员寻找更好的药物靶点,为临床研究者研发诊断性检测方法提供依据,同时提高公共卫生工作人员对疾病早期预警信号的发现能力和对传染病疫情的追踪和响应能力[19]。
大数据最大的局限性在于包含了大量噪声信号,因此通过大数据得出的关联信号无法验证是否具有意义,可能导致高估疾病的影响程度、触发错误警报、造成虚假关联和生态学谬误。最典型的案例是2013年美国的流感大暴发,科学家通过分析互联网数据估计流感的影响程度,然而与传统公共卫生监测方法相比,这种方法却大大高估了流感的高峰期水平。因此在利用大数据进行关联研究时,需要合理的流行病学方法尽可能控制选择偏倚和混杂偏倚,以避免虚假关联。
大数据的流行病学设计和方法主要分为测量准确的生物大数据关联研究和测量不够准确的非生物大数据关联研究两类[19]。对于生物大数据而言,通常采用重复试验的方法,在统计显著性方面产生更强的信号,从而有效解决真实信号和噪音信号相混的问题;对于非生物大数据,如临床大数据和健康大数据,首先要建立合理的关联假设,然后通过大队列研究、大型随机对照试验等流行病学设计验证关联和证实关联的临床实用性。最终在获得研究结果的基础上,利用专业知识对结果进行解读,形成新知识,并利用机器学习算法等大数据技术对知识内容进行合成、转化和管理。
三、 精准医疗与流行病学精准医疗体系的核心内容为将基础研究成果快速有效地转化为临床实践、预防措施和卫生政策,使得科研成果尽快用于患者乃至全民的健康促进,即转化研究。转化研究(translational research,TR)是指将实验室和临床前研究发现用于发展人群研究和实验的过程,流行病学作为一门工具学科,在精准医疗转化研究的T1~T4各阶段均发挥着重要作用,其对于实验室研究成果转化为临床实践的推动作用至关重要,为临床指南和规范的制定提供了可靠理论支持[20-21](图 1)。
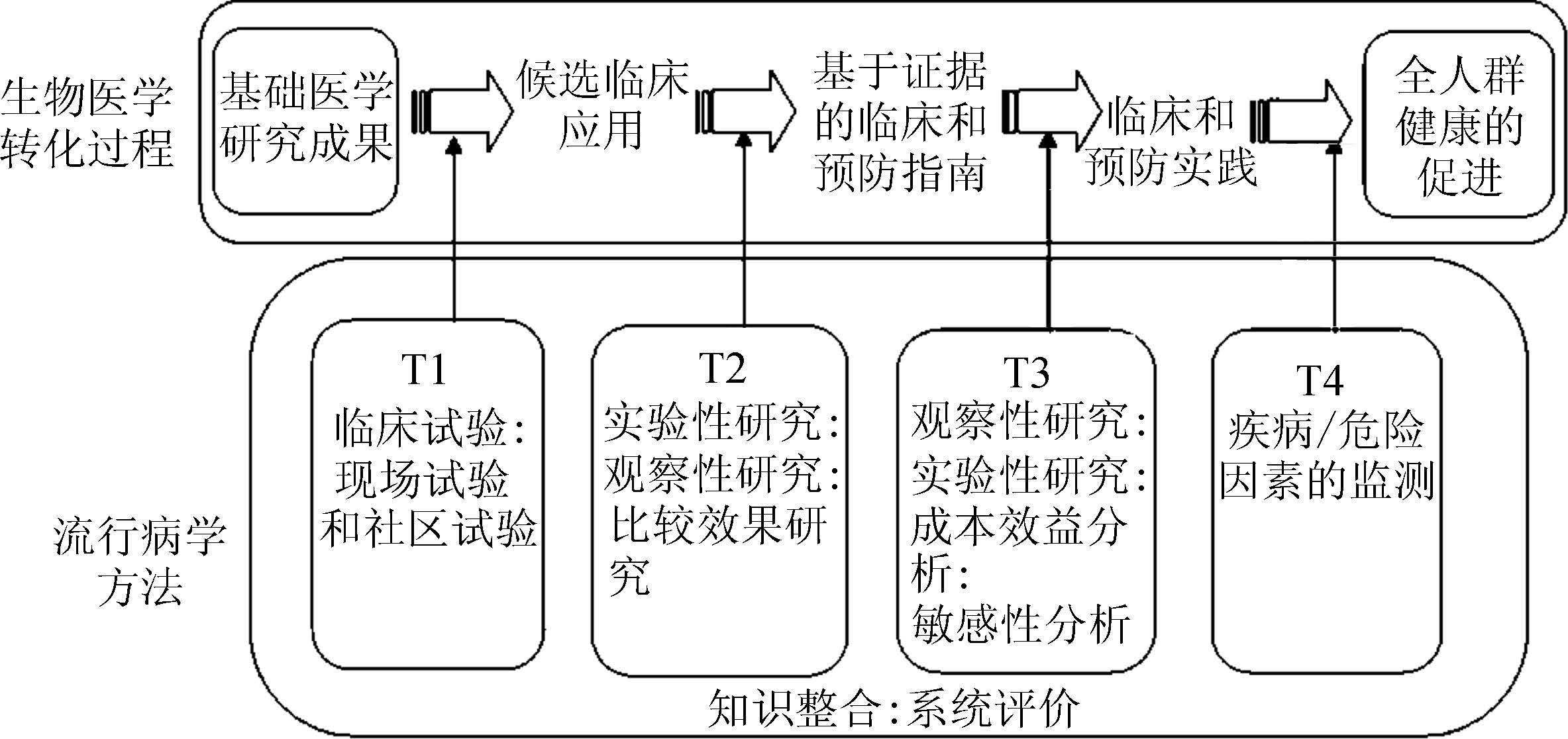
|
| 图 1 流行病学在生物医学转化中的应用 |
然而,仅依赖生物医学研究的转化,对于实现精准医疗的目标是不够的。以基因诊断、分子靶向治疗和疾病风险预测为代表的新型医疗手段价格昂贵,技术要求高,难以在医疗资源匮乏、经济水平较差的国家和人群中实施;另外,精准医疗倡导通过养成良好的生活行为方式促进全民健康,而健康宣教运动以及相关健康政策的顺利实施与政治、经济和文化等社会因素密不可分[22]。因此,在进行生物医学转化的同时,还需要利用社会流行病学的方法分析社会因素对人群疾病/健康的影响,解决疾病/健康的社会不公平问题。通过生物医学、社会科学、流行病学等多学科相互融合、优势互补,构建未来精准医疗的新框架[22](图 2)。
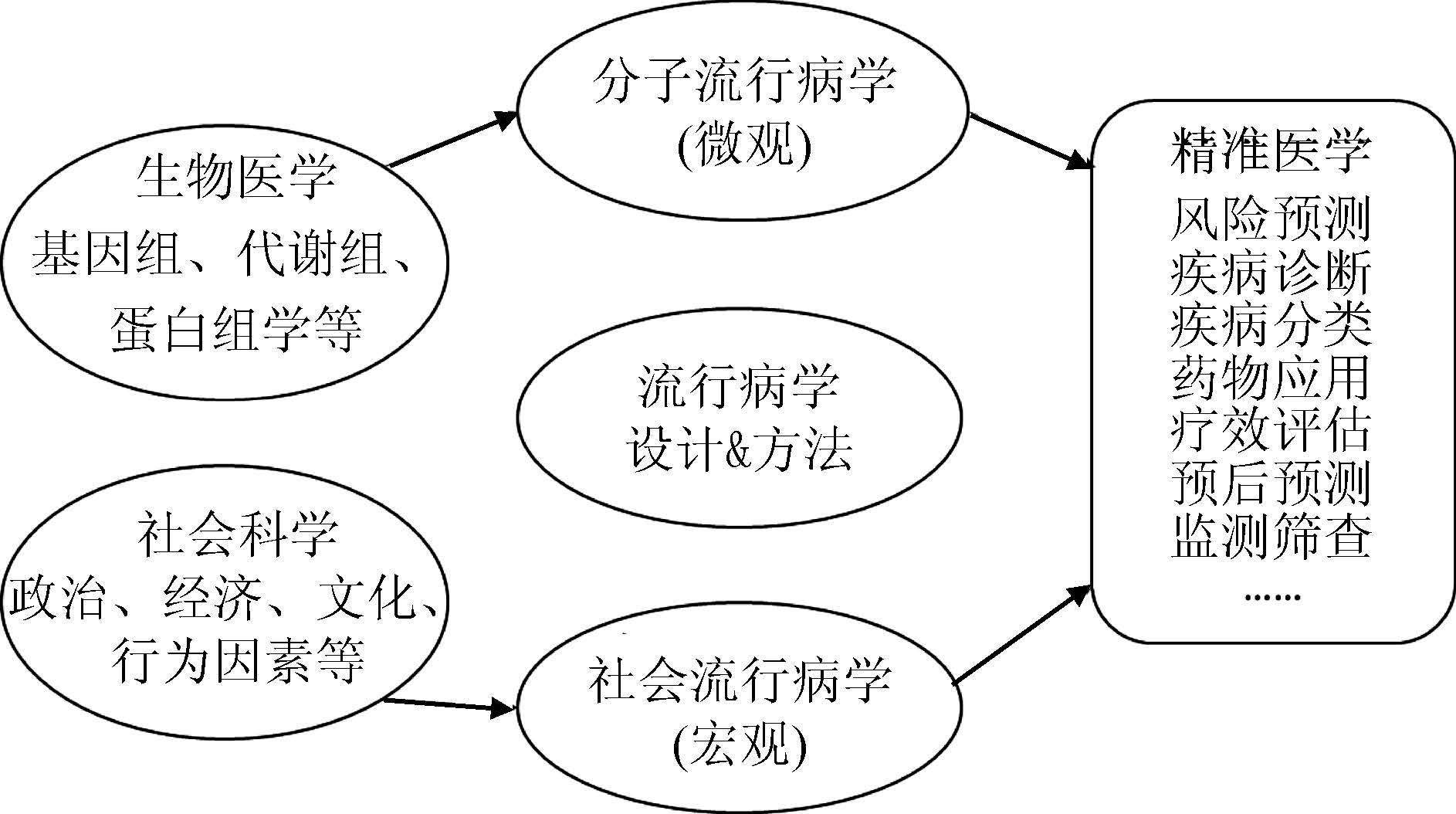
|
| 图 2 精准医疗框架:“生物医学+社会科学+流行病学” 模型 |
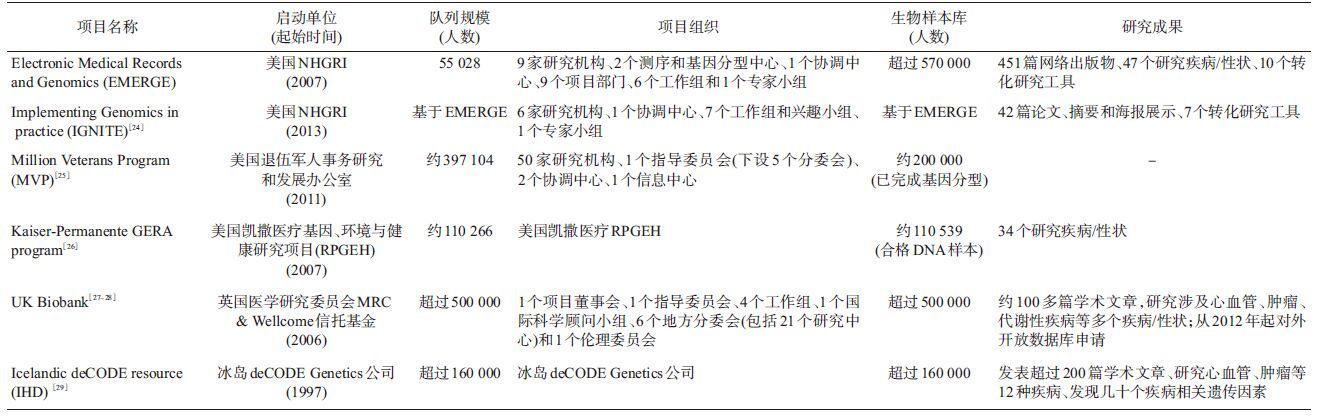
近年来,随着电子病历系统(EMR)的普及和完善,基于EHR数据建立的大型动态队列逐渐成为流行病学新的热点(表 2),通过将生物样本库中的遗传信息(基因分型、高密度测序、家族史等)及各类组学信息与EMR进行整合匹配,提供大量表型信息和一些难以测量的暴露信息,实时动态跟踪队列中个体,使得随访更加容易,克服了传统队列的多种局限性;同时,EMR可监测各种基因诊疗手段在不同人群中的实际效果,为临床决策的制定提供最真实的数据。2015年奥巴马在“精准医疗”计划中提出在已有大队列的基础上建立一个“百万美国人健康队列”,在短期内可验证现有诊疗和预防措施的有效性,从长期看可用于探索和验证疾病/健康的基因和环境因素[23]。由此可见,基于EHR的动态大队列不仅有利于我们对疾病机制及其防治的深入了解,同时也能够在短期内使高危人群获益,通过改变不良的行为因素和环境因素,实现疾病的一级和二级预防。
随着全自动高通量技术的灵敏度和可重复性不断提升、检测成本不断降低、EMR的普及应用和各类公共卫生监测数据平台的开放,为今后流行病学研究提供了丰富的数据资源。同时,基因组学、蛋白组学、转录组学、代谢组学、表观遗传组学、微生物组学、暴露组学等组学分析方法的建立和成熟,为流行病学更细致地定义疾病分类、更深入地阐释发病原因和更准确地预测疾病风险或治疗效果提供了可能,也催生出“系统流行病学”(systems epidemiology)这一流行病学新分支的产生(图 3)[30]。系统流行病学是以系统生物学(systems biology)为基础,以数学和计算机技术为手段整合各生物组学数据,并将通路分析和观察性研究设计相融合,从而加深对人类疾病的生物学机制的认知[31]。传统遗传流行病学研究中单一地将单核苷酸多态性等生物标志与疾病结局进行关联研究,忽略了基因-环境的交互作用,并且难以体现该生物标记在暴露-疾病链中发挥的作用。系统流行病学研究采用全组学设计(globolomic design),整合基因组、转录组、蛋白组、代谢组、表观遗传组和MicroRNA等多层次信息,并利用微阵列技术动态观察与分析队列中个体从基线到结局整个过程中基因表达谱的变化情况,层次更加丰富,信息更加复杂,有助于更加精准地解释暴露-疾病链中的分子机制[30]。未来流行病学研究将以现有的大规模高质量队列为基础,在系统流行病学的设计指导下,对数据、样本的获取和统计分析过程进行严格的质量控制,进一步论证已发现的生物标志的病因学作用[32]。
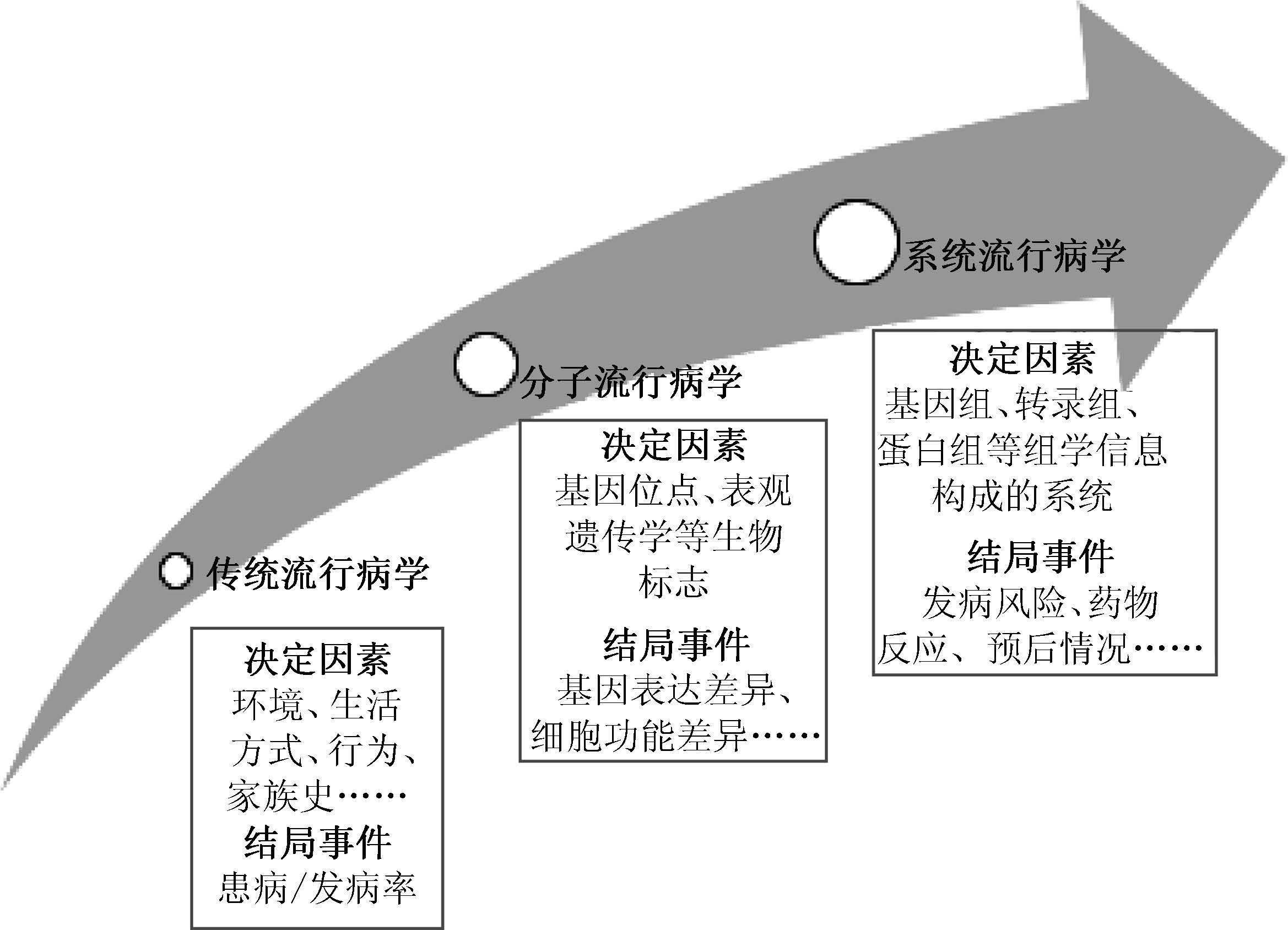
|
| 图 3 流行病学研究的发展 |
医学大数据作为新兴领域,无论是数据的挖掘、存储、共享还是安全和伦理问题都将成为其今后发展将要面临的挑战。
1. 挑战:
(1)数据挖掘:临床大数据包含大量医学影像学资料,目前的统计学方法尚无法分析如脑电图或脑部核磁共振图像等数据[8]。另外,一个高分辨率的图像大小为几十GB,如果需要对成千上万个这样的医学图像进行比较,普通计算机难以完成如此庞大的计算量,而并行计算方案尚存在分割效率低和数据转移困难等问题,因此未来的发展方向将是进一步完善分割算法并对数据进行无损压缩[33]。
(2)数据存储:组学技术的发展产生了海量数据积累,例如欧洲生物信息研究所(EBI)作为世界最大生物数据库之一,2012年存储约2 PB基因数据,且该数字以每年翻倍的速度继续增长[34];另一方面,医学大数据的非结构性使得医学数据的存储和处理较其他领域的研究数据更为复杂。目前还没有综合、经济且安全的大数据存储解决方案,因此海量数据的快速处理和存储就成为重要的课题[35]。
(3)数据共享:未来的大数据研究研究依赖多个实验室或数据平台的资源共享,而目前数据共享主要面临的困境:①数据间的整合与共享能够提高数据利用价值,以便于进一步深入挖掘数据中包含的信息,而这种整合与共享需要科研机构和公司间积极寻求合作,建立互助互利的数据联盟模式;②缺乏统一的标准,每个数据库存储所使用的软件及数据格式各不相同,特别是后者可能会给数据间的比较和分析带来困难[3];③TB甚至PB级海量数据已超出现有个人计算机和网络文件共享程序的能力范围,需要建立新型共享方式[34]。
(4)数据隐私:未来系统流行病学研究将涉及大量人群的暴露组学和生物组学信息,因此如何确保这些数据的隐私安全至关重要。已经有遗传学家证明即使是匿名数据库,个人的遗传隐私仍然可能被泄露,个人的遗传隐私无法得到保障可能引发伦理学纠纷。目前的解决方法主要是对数据库使用者进行实名登记,并公开使用者的信息这样的监督机制[3]。
2. 机遇:
(1)云计算系统:是将海量数据分配到成千上万个远程服务器“云”上[36]。目前,除EBI等学术云项目,一些商业云如Rackspace、VMware或亚马逊、IBM和微软公司的云系统也可以提供研究人员使用[34]。首先云计算系统最显著的优点是完全基于虚拟空间,有利于降低研究成本。第二,云计算系统能够提供免费、开源的基础软件,实现服务器和网络间的基础通信、服务器间分派工作以及执行复杂的计算任务。第三,一些公司提供云计算数据分析的用户友好界面,以及提供通用软件帮助找到云的便捷入口,使云的应用更加用户友好。另外,为适应急速增长的数据量,一些机构将基因组分析的各步骤组装成工作流程,如华大公司的Gaea,通过多个云计算机的并行运算使得基于云的分析更加快捷。
(2)医学大数据平台:目前全世界已建立起一些医学大数据开发平台,例如eMERGE项目,将生物样本库与EMR进行整合匹配,从而进行大规模、高通量的遗传学研究。整个系统共包含了患者纳入、生物样本采集、基因组研究、队列建立、数据隐私管理、数据整合、精准医疗、结果反馈和患者教育9个部门,研究内容涵盖基于EMR进行表型分型算法研发(PheKB)、基于eMERGE-PGx 队列研究遗传变异与药物反应间关系(SPHINX)、全表型组关联研究(PheWAS)等项目。目前已涵盖55 028个队列、进行了47个疾病表型的研究,共发表文章近400篇。
(3)转化医学研究所:如麻省理工学院-哈佛大学博德研究所(Broad Institute)、阿拉巴马大学伯明翰分校临床与转化科学中心(CCTS)、杜克大学转化医学中心、梅奥转化医学中心、耶鲁临床研究中心等一批以高校内部或高校间联盟合作模式建立起的转化医学中心,尝试通过不同的转化模式加快实验室研究和大数据挖掘成果转化为临床实践的效率,为今后医学大数据开发和精准医疗的研究提供了宝贵经验[37-40]。
综上所述,医学大数据和精准医疗为未来流行病学学科的创新发展起到了重要的推动作用,今后有望在流行病学、信息学、临床医学、生命科学等学科的共同参与下,提高疾病防控技术水平,支撑医疗卫生体系建设,提升全生命周期的生活质量,促进健康发展和健康行为,实现医疗公平和减少疾病和残疾负担,实现随时、随地、随需、个体化、人性化和高质量的全谱健康的愿景。
| [1] | 王波, 吕筠, 李立明. 生物医学大数据:现状与展望[J]. 中华流行病学杂志 , 2014, 35 (6) : 617–620 DOI:10.3760/cma.j.issn.0254-6450.2014.06.001 Wang B, Lyu J, Li LM. Big data in biomedicine:status quo and perspective[J]. Chin J Epidemiol , 2014, 35 (6) : 617–620 DOI:10.3760/cma.j.issn.0254-6450.2014.06.001 |
| [2] | Hansen C, Johnson CR, Pascucci V, et al.Visualization for data-intensive science//Hey T,Tansley S,Tolle K. The fourth paradigm:data intensive scientific discovery[M].Redmond,WA: Microsoft Research, 2009: 1 |
| [3] | Service RF. Biology's dry future[J]. Science , 2013, 342 (6155) : 186–189 DOI:10.1126/science.342.6155.186 |
| [4] | Committee on a Framework for Development a New Taxonomy of Disease, National Research Council.Toward precision medicine:building a knowledge network for biomedical research and a new taxonomy of disease[M].Washington,DC: National Academies Press, 2011 |
| [5] | Horgan D, Paradiso A, McVie G, et al. Is precision medicine the route to a healthy world?[J]. Lancet , 2015, 386 (9991) : 336–337 DOI:10.1016/S0140-6736(15)61404-0 |
| [6] | Chen R, Snyder M. Promise of personalized omics to precision medicine[J]. Wiley Interdiscip Rev Syst Biol Med , 2013, 5 (1) : 73–82 DOI:10.1002/wsbm.1198 |
| [7] | 俞国培, 包小源, 黄新霆, 等. 医疗健康大数据的种类、性质及有关问题[J]. 医学信息学杂志 , 2014, 35 (6) : 9–12 DOI:10.3969/j.issn.1673-6036.2014.06.002 Yu GP, Bao XY, Huang XT, et al. Medical and health big data:types,characteristics and relevant issues[J]. J Med Intell , 2014, 35 (6) : 9–12 DOI:10.3969/j.issn.1673-6036.2014.06.002 |
| [8] | Sejdić E. Medicine:adapt current tools for handling big data[J]. Nature , 2014, 507 (7492) : 306 DOI:10.1038/507306a |
| [9] | Borangíu T, Purcarea V. The future of healthcare-information based medicine[J]. J Med Life , 2008, 1 (2) : 233–237 |
| [10] | Suhre K, Shin SY, Petersen AK, et al. Human metabolic individuality in biomedical and pharmaceutical research[J]. Nature , 2011, 477 (7362) : 54–60 DOI:10.1038/nature10354 |
| [11] | Aronson SJ, Rehm HL. Building the foundation for genomics in precision medicine[J]. Nature , 2015, 526 (7573) : 336–342 DOI:10.1038/nature15816 |
| [12] | Chen B, Butte AJ. Leveraging big data to transform target selection and drug discovery[J]. Clin Pharmacol Ther , 2016, 99 (3) : 285–297 DOI:10.1002/cpt.318 |
| [13] | Zeng T, Zhang WW, Yu XT, et al. Big-data-based edge biomarkers:study on dynamical drug sensitivity and resistance in individuals[J]. Brief Bioinform , 2015 DOI:10.1093/bib/bbv078 |
| [14] | Milinovich GJ, Magalhaes RJS, Hu WB. Role of big data in the early detection of Ebola and other emerging infectious diseases[J]. Lancet Glob Health , 2015, 3 (1) : e20–21 DOI:10.1016/S2214-109X(14)70356-0 |
| [15] | Kao RR, Haydon DT, Lycett SJ, et al. Supersize me:how whole-genome sequencing and big data are transforming epidemiology[J]. Trends Microbiol , 2014, 22 (5) : 282–291 DOI:10.1016/j.tim.2014.02.011 |
| [16] | Hay SI, George DB, Moyes CL, et al. Big data opportunities for global infectious disease surveillance[J]. PLoS Med , 2013, 10 (4) : e1001413 DOI:10.1371/journal.pmed.1001413 |
| [17] | Meyer AM, Olshan AF, Green L, et al. Big data for population-based cancer research:the integrated cancer information and surveillance system[J]. N C Med J , 2014, 75 (4) : 265–269 |
| [18] | Almalki M, Gray K, Sanchez FM. The use of self-quantification Systems for personal health information:big data management activities and prospects[J]. Health Inf Sci Syst , 2015, 3 (Suppl 1) : S1 DOI:10.1186/2047-2501-3-S1-S1 |
| [19] | Khoury MJ, Ioannidis JPA. Big data meets public health[J]. Science , 2014, 346 (6213) : 1054–1055 DOI:10.1126/science.aaa2709 |
| [20] | Khoury MJ, Gwinn M, Ioannidis JPA. The emergence of translational epidemiology:from scientific discovery to population health impact[J]. Am J Epidemiol , 2010, 172 (5) : 517–524 DOI:10.1093/aje/kwq211 |
| [21] | Khoury MJ, Gwinn M, Yoon PW, et al. The continuum of translation research in genomic medicine:how can we accelerate the appropriate integration of human genome discoveries into health care and disease prevention?[J]. Genet Med , 2007, 9 (10) : 665–674 DOI:10.1097/GIM.0b013e31815699d0 |
| [22] | Nishi A, Milner Jr DA, Giovannucci EL, et al. Integration of molecular pathology,epidemiology and social science for global precision medicine[J]. Expert Rev Mol Diagn , 2016, 16 (1) : 11–23 DOI:10.1586/14737159.2016.1115346 |
| [23] | Khoury MJ, Evans JP. A public health perspective on a national precision medicine cohort:balancing long-term knowledge generation with early health benefit[J]. JAMA , 2015, 313 (21) : 2117–2118 DOI:10.1001/jama.2015.3382 |
| [24] | Weitzel KW, Alexander M, Bernhardt BA, et al. The IGNITE network:a model for genomic medicine implementation and research[J]. BMC Med Genomics , 2016, 9 (1) : 1 DOI:10.1186/s12920-015-0162-5 |
| [25] | Gaziano JM, Concato J, Brophy M, et al. Million veteran program:a mega-biobank to study genetic influences on health and disease[J]. J Clin Epidemiol , 2016, 70 : 214–223 DOI:10.1016/j.jclinepi.2015.09.016 |
| [26] | Banda Y, Kvale MN, Hoffmann TJ, et al. Characterizing race/ethnicity and genetic ancestry for 100000 subjects in the genetic epidemiology research on adult health and aging (GERA) cohort[J]. Genetics , 2015, 200 (4) : 1285–1295 DOI:10.1534/genetics.115.178616 |
| [27] | Collins R. What makes UK Biobank special?[J]. Lancet , 2012, 379 (9822) : 1173–1174 DOI:10.1016/S0140-6736(12)60404-8 |
| [28] | Allen NE, Sudlow C, Peakman T, et al. UK biobank data:come and get it[J]. Sci Transl Med , 2014, 6 (224) : 224ed4 DOI:10.1126/scitranslmed.3008601 |
| [29] | Gulcher JR, Stefánsson K. The Icelandic healthcare database and informed consent[J]. N Engl J Med , 2000, 342 (24) : 1827–1830 DOI:10.1056/NEJM200006153422411 |
| [30] | Spitz MR, Bondy ML. The evolving discipline of molecular epidemiology of cancer[J]. Carcinogenesis , 2010, 31 (1) : 127–134 DOI:10.1093/carcin/bgp246 |
| [31] | Lund E, Dumeaux V. Systems epidemiology in cancer[J]. Cancer Epidemiol Biomarkers Prev , 2008, 17 (11) : 2954–2957 DOI:10.1158/1055-9965.EPI-08-0519 |
| [32] | Hu FB. Metabolic profiling of diabetes:from black-box epidemiology to systems epidemiology[J]. Clin Chem , 2011, 57 (9) : 1224–1226 DOI:10.1373/clinchem.2011.167056 |
| [33] | Savage N. Bioinformatics:big data versus the big C[J]. Nature , 2014, 509 (7502) : S66–67 DOI:10.1038/509S66a |
| [34] | Marx V. Biology:the big challenges of big data[J]. Nature , 2013, 498 (7453) : 255–260 DOI:10.1038/498255a |
| [35] | Costa FF. Big data in biomedicine[J]. Drug Discov Today , 2014, 19 (4) : 433–440 DOI:10.1016/j.drudis.2013.10.012 |
| [36] | 高汉松, 肖凌, 许德玮, 等. 基于云计算的医疗大数据挖掘平台[J]. 医学信息学杂志 , 2013, 34 (5) : 7–12 DOI:10.3969/j.issn.1673-6036.2013.05.002 Gao HS, Xiao L, Xu DW, et al. Medical data mining platform based on cloud computing[J]. J Med Intell , 2013, 34 (5) : 7–12 DOI:10.3969/j.issn.1673-6036.2013.05.002 |
| [37] | 栗美娜. 阿拉巴马大学伯明翰分校临床与转化科学中心及对中国的借鉴[J]. 转化医学杂志 , 2015, 4 (1) : 8–10 DOI:10.3969/j.issn.2095-3097.2015.01.003 Li MN. Experience of University of Alabama Birmingham center for clinical and translational science and reference to China[J]. Transl Med J , 2015, 4 (1) : 8–10 DOI:10.3969/j.issn.2095-3097.2015.01.003 |
| [38] | 孙雨龙. 从Mayo Clinic的转化经验体会转化医学[J]. 医学研究与教育 , 2013, 30 (1) : 5–8 Sun YL. Experience from translational medicine at Mayo Clinic[J]. Med Res Educ , 2013, 30 (1) : 5–8 |
| [39] | 杨汀, 蒋典华. 美国杜克大学的转化医学实践[J]. 转化医学研究:电子版 , 2011, 1 (1) : 7–21 Yang T, Jiang DH. Translational medicine at Duke University[J]. Transl Med J:Electron Ed , 2011, 1 (1) : 7–21 |
| [40] | 周玲君, 高绥之, 贾兆宝, 等. 中美转化医学中心建设现状与展望[J]. 转化医学杂志 , 2014, 3 (5) : 311–314 DOI:10.3969/j.issn.2095-3097.2014.05.014 Zhou LJ, Gao SZ, Jia ZB, et al. The current situation and prospect of China and America centers for translational medicine[J]. Transl Med J , 2014, 3 (5) : 311–314 DOI:10.3969/j.issn.2095-3097.2014.05.014 |
 2016, Vol. 37
2016, Vol. 37