文章信息
- 柯碧霞, 何冬梅, 谭海玲, 曾洪辉, 杨彤, 李柏生, 柯昌文 .
- Ke Bixia, He Dongmei, Tan Hailing, Zeng Honghui, Yang Tong, Li Bosheng, Ke Changwen .
- 基于微球液态阵列分子技术的沙门菌血清分型研究
- Study on Salmonella serotyping by use of Microsphere-based Liquid Array method
- 中华流行病学杂志, 2016, 37(8): 1137-1141
- CHINESE JOURNAL OF EPIDEMIOLOGY, 2016, 37(8): 1137-1141
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.08.017
-
文章历史
收稿日期: 2016-02-24
2. 510440 广州, 广东省生物制品与药物研究所耐药监测室
2. Drug Resistance Monitoring Laboratory, Guangdong Provincial Institute of Biological Products and Material Medicine, Guangzhou 510440, China
沙门菌的血清学分型是了解沙门菌感染和流行的重要分型工具。目前使用的考夫曼-怀特表(Kauffmann-White Scheme)是由WHO标准合作中心和巴斯德研究所沙门菌研究中心修订后的沙门菌血清分型表,其中通过种/亚种、O抗原、1相H抗原和2相H抗原对沙门菌进行血清分型。玻片凝集法是传统的沙门菌血清分型方法,一般需时3 d才能完成O和H抗原的检测。有些沙门菌需反复传代或诱导才能检测到抗原,完成分型需时更长(5~7 d)。而对于粗糙型沙门菌和极少数沙门菌用传统的玻片凝集法不能进行血清分型。应用基于微球的液态阵列分子技术在当天同时完成大量的沙门菌血清分型,且对粗糙型沙门菌和不能分型沙门菌进行血清分型[1-2]。广东省从2010年起在全省30家哨点医院开展食源性疾病监测,至2014年从该监测系统共分离到4 942株沙门菌。为此本研究应用微球液态阵列分子技术对其中腹泻病例分离株进行血清型测定,并与传统血清分型结果比对,分析评价该项技术在广东省公共卫生微生物实验室检测沙门菌血清型的可行性。
材料与方法1. 菌株来源:所选200株菌为2010-2014年广东省食源性疾病监测中从腹泻病例粪便中分离到的89种不同血清型的沙门菌。
2. 仪器和试剂:超微量分光光度计为德国IMPLEN公司产品;LUMINEX200基于微球的液态阵列分子分析仪是美国MERCKMILLIPORE公司产品;PCR仪为美国BIORAD公司产品。xMAP®Salmonella Serotyping Assay Kit(SSA)和基于微球的液态阵列分子分析仪仪器配套校准验证试剂盒是美国Luminex公司产品,HotStarTaqMasterMix是美国Qiagen公司产品,InstaGene matrix为美国BIORAD公司产品,沙门菌全套诊断血清为丹麦SSI公司产品。
3. 试验方法:用InstaGene matrix提取菌株核酸;用超微量分光光度计测定核酸浓度,并稀释至100 ng/μl;用SSA试剂盒中的引物和HotStarTaqMasterMix PCR反应液进行多重PCR扩增;多重PCR产物与用SSA试剂盒中的微球进行杂交和染色;然后在基于微球的液态阵列分子分析仪上进行数据读取;最后用仪器配套的软件进行数据分析。
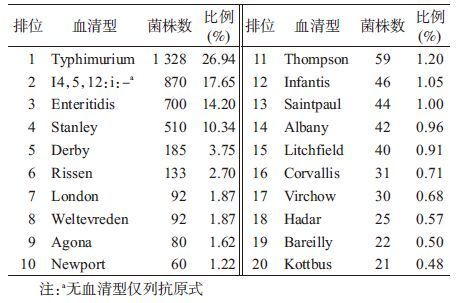
结 果1. 广东省沙门菌病例分离株血清型分布概况:2010-2014年广东省食源性疾病监测中从腹泻病例粪便分离到沙门菌4 942株,除12株不能分型外(采用丹麦SSI沙门菌诊断血清分型),共分到189种血清型,分型率为99.76%。其中99.21%的菌株为常见的A~F群,只有39株(0.79%)为A~F群以外的血清型,分别为O ∶ 13(G)群、O ∶ 6,14(H)群、 O ∶ 16(I)群、O ∶ 18(K)群、O ∶ 28(M)群、 O ∶ 35(O)群、O ∶ 40(R)群、O ∶ 43(U)群、O ∶ 47(X)群。广东省腹泻病例沙门菌感染中最常见的5种血清型分别为Typhimurium(26.94%)、I4,5,12:i:-(17.65%)、Enteritidis(14.20%)、Stanley(10.34%)、Derby(3.75%),前5位血清型占所有菌株的72.88%(表 1)。排在前100位的血清型占所有菌株的98.09%,在2010-2014年有84种血清型只分离到1株菌。SSA试剂盒能同时检测7种O抗原和35种H抗原,显示居前100位的血清型有15种涉及到的O抗原或H抗原不在SSA试剂盒检测范围内,其余85种能用SSA试剂盒分型的血清型数量占前100位血清型菌株数的98%。SSA试剂盒也能检测到100位后的沙门菌完整血清型或血清型部分抗原信息。因此,应用SSA试剂盒可对广东省腹泻病例中96%以上(98%,98.09%)的沙门菌进行分子血清分型。
2. 基于微球液态阵列分子技术对沙门菌分型:
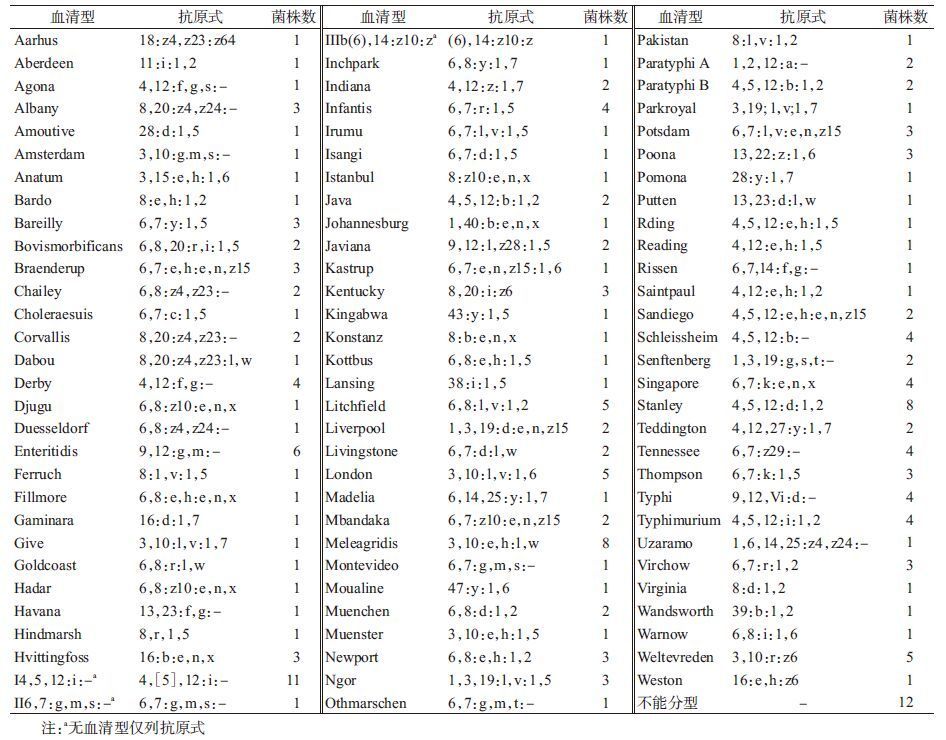
(1)菌株血清型及抗原式:2010-2014年广东省食源性疾病监测中从腹泻病例粪便中分离200株89种血清型沙门菌的抗原式及菌株数见表 2。
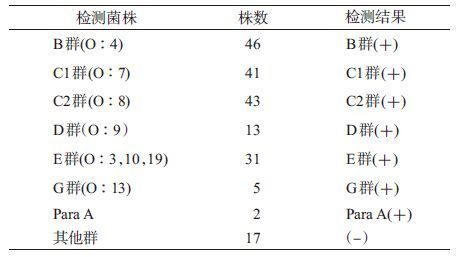
(2)O抗原检测:SSA试剂盒能检测的O群抗原为B、C1、C2、D、E和G群及甲型副伤寒。200株沙门菌中有198株能用传统血清凝集试验确定O抗原,采用SSA试剂盒检测,其中181株菌能检测出O抗原(表 3),与传统血清凝集试验结果的符合率为100%;其余17株,包括F群(O11)、H群(O6,14)、 I群(O16)、K群(O18)、M群(O28)、P群(O38)、Q群(O39)、R群(O40)、U群(O43)和X群(O11),采用SSA试剂盒检测结果均为阴性。
(3)H抗原检测:SSA试剂盒的H抗原目标基因有35个,分别为a、b、c、d、j、eh、i、k、r、z10、z29、z6、y、L-complex、v、z28、EN-complex、x、z15、1-complex、2、5、6、7、z24、G-complex、f、(m/g,m)、(m/m,t)、p、s、t-1、z51、z4-complex。本次所用的沙门菌株除p、(m/m,t)和z51这3个抗原不涉及外,其余的32个抗原均涉及(图 1)。200株菌所包含的H抗原共有537个,有34种不同的H抗原,其中z23和w两个基因不在SSA的检测范围内。98.32%(528/537)的H抗原用SSA检测的结果与传统血清凝集试验的结果相符,其中a、b、c、j、eh、i、k、r、z29、z6、y、z28、EN-complex、x、z15、1-complex、6、7、z24、G-complex、f、(m/g,m)、 s、t-1、z4-complex 25个基因的符合率均为100%,有6个H抗原基因有漏检情况,含z23和w两个基因的菌株(19株)检测结果均为阴性。H抗原基因漏检情况:缺L-complex、 H5、Hv、Hd基因各1株,缺Hz10和H2基因各2株。本次检测菌株有5株菌含有H t抗原,只有1株用SSA能检测到H t抗原。

|
| 图 1 200株沙门菌的34种H抗原数量 |
(4)其他基因检测:SSA试剂盒的其他目标基因(additional targets,AT)包括sdf、fljB和Vi 3个基因。本次采用传统血清凝集试验可明确血清型的188株菌中有134株为双相沙门菌,54株为单相沙门菌,理论上双相沙门菌的fljB基因检测结果应为阳性,而单相沙门菌的fljB基因检测结果应为阴性。134株双相沙门菌中有125株为fljB基因检测结果为阳性,9株为阴性,阴性率为7.35%(9/134);54株单相沙门菌中有50株菌fljB基因检测结果为阴性,4株为阳性,阳性率为7.41%(4/54);fljB基因的符合率为93.09%(175/188)。sdf基因是肠炎沙门菌的特异基因,可将肠炎沙门菌和Gallinarum、Pullorum沙门菌区分开。本次明确血清型为肠炎沙门菌的有6株,sdf基因均检测为阳性,符合率为100%;另有1株血清型不确定沙门菌用SSA试剂盒检测为肠炎沙门菌,sdf基因也检测为阳性。4株伤寒沙门菌,Vi基因检测3株为阳性,1株阴性,符合率为100%。
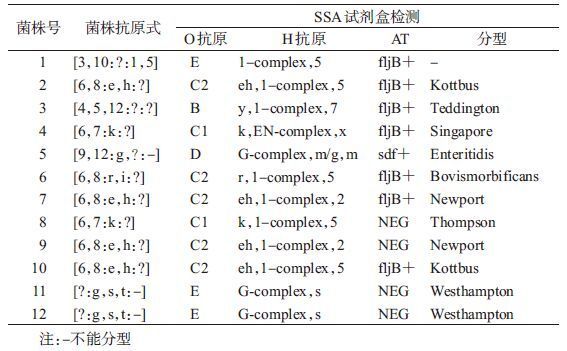
(5)血清学不能分型菌株的检测:本次采用传统血清凝集试验不能分型的菌株有12株。用SSA试剂盒检测,有11株能成功分型(表 4)。其中1号菌株仍不能检出H1相抗原,原因可能是该菌株的H1相抗原不在试剂盒检测范围内。11和12号菌株用血清检测时O抗原与多种血清凝集,但用SSA试剂盒检测很明确O抗原为E群。有9株菌用血清凝集试验不能检测出H2相,2株菌不能检出H1相,用SSA试剂盒检测均可检出相应的H1相和H2相。
2002-2006年美国CDC在该国分离到的人源沙门菌超过161 000株,约有1 000种血清型,其中常见的100种血清型占98%[2]。因此SSA试剂盒是针对美国最常见的100种血清型沙门菌而设计[1-2]。我国广东省2010-2014年分离的4 942株人源沙门菌有189种血清型,前100 种血清型占全部菌株的98.08%;居前100位血清型中有15种涉及到O或H抗原不列在SSA试剂盒检测范围内,SSA试剂盒分型率为98%;SSA试剂盒也能检测到广东省分离菌株的100位后沙门菌完整血清型或血清型的部分抗原信息。因此,应用SSA试剂盒可对广东省96%以上腹泻病例分离的沙门菌进行血清分子分型。
沙门菌的O抗原是由rfb基因编码。SSA试剂盒针对B、C1、C2、D、E、G群和甲型副伤寒O抗原的rfb基因特异片段设计相应的引物和探针,能在同一反应中同时检测这7种O抗原[1]。本次检测有198株沙门菌采用SSA试剂盒检测O抗原,其中181株为阳性,结果与传统血清分型方法完全一致;17株为阴性(因其O抗原不在试剂检测范围内)。故SSA试剂盒O抗原检测的敏感性和特异性均好,且无交叉反应。采用SSA试剂盒检测2株粗糙型沙门菌均可检测到明确的E群O抗原。由于E群与G群均含多种O抗原,SSA试剂盒检测虽能区分菌株是E群或G群,但不能判断其亚群,如E1群还是E4群,G1群还是G2群。因此还需补充做传统血清凝集试验。
沙门菌的H抗原主要由fliC和fljB基因编码,分别编码H第一和第二相抗原。鞭毛蛋白基因fliC和fljB被协调调控,在一个单细胞中只能表达鞭毛的一相抗原[3]。因此,用传统血清玻片凝集方法检测H抗原时需要做位相诱导试验才能检测到沙门菌的两相H抗原。SSA试剂盒的fliC和fljB基因相应引物和探针是针对15种H抗原、 5种复合主要抗原和16种复合次要抗原,能在同一反应中同时检测35种H抗原。本文检测的200株沙门菌中,所有537个H抗原基因的特异性均好,无交叉反应。有19菌株(含H:z23,-w抗原)未检测到完整的两相H抗原,也仅是其H抗原不在试剂盒的检测范围内。有8株(4%)未检测到预期的H抗原基因,主要集中在H:d,-v,-z10,-z,-g,s,t,-1,5,-1,2,L-complex。部分基因没有检出是由于等位基因的多样性。本文中有1株菌未检测到H:1,5,原因可能是其H:1,5基因不在检测范围内。本文菌株中有5株含H t抗原,采用SSA试剂盒只检测到1株,原因可能是其余4株菌的H t抗原均由非H t-1基因编码。此外,未检测到相应H抗原的菌株,应进一步分析其原因。
沙门菌为双相鞭毛抗原细菌,但也有单相菌(如肠炎沙门菌和伤寒沙门菌),为单相血清型。目前已发现有些菌株由典型的双相血清型变种为单相血清型[4-5]。本文中采用SSA试剂盒有4株单相菌被检测为双相菌。可能的解释是当菌株fljB基因缺失,可导致最常见的单相血清型和单相变种,但用传统方法检测可能被鉴定为单相菌。例如,甲型副伤寒沙门菌(抗原式为1,2,12:a:-)含有一个fljBH:1,5等位基因,但是通常不表达[6],但H:1,5等位基因可以检测到。此外,因部分fljB基因缺失成为单相菌的菌株用分子方法来检测也会得到不同的结果,这主要由fljB基因缺失的属性决定。如果fljB基因的某部分与特定的探针同源,且量足够多,引物可与靶标结合扩增,这部分基因可与探针反应,即使该抗原不表达,采用传统方法无法检测到,但分子学方法却可检测到。
综上所述,尽管目前基于微球的液态阵列分子技术对沙门菌血清分型还不能完全替代传统血清学分型方法,但其具有诸多优点,特别是在处理沙门菌暴发事件时,可在数小时内不仅对暴发菌株进行分型,还可排除非暴发菌株,显著减轻实验室工作量。本文显示采用SSA试剂盒检测可解决广东省绝大部分人源沙门菌株的血清分型,且比传统方法更高通量更快速,可应用于食源性疾病的散发和暴发监测。
| [1] | Fitzgerald C, Collins M, van Duyne S, et al. Multiplex,bead-based suspension array for molecular determination of common Salmonella serogroups[J]. J Clin Microbiol , 2007, 45 (10) : 3323–3334 DOI:10.1128/JCM.00025-07 |
| [2] | McQuiston JR, Waters RJ, Dinsmore BA, et al. Molecular determination of H antigens of Salmonella by use of a microsphere-based liquid array[J]. J Clin Microbiol , 2011, 49 (2) : 565–573 DOI:10.1128/JCM.01323-10 |
| [3] | Silverman M, Zieg J, Hilmen M, et al. Phase variation in Salmonella:genetic analysis of a recombinational switch[J]. Proc Natl Acad Sci USA , 1979, 76 (1) : 391–395 DOI:10.1073/pnas.76.1.391 |
| [4] | Garaizar J, Porwollik S, Echeita A, et al. DNA microarray-based typing of an atypicalmonophasic Salmonella enterica serovar[J]. J Clin Microbiol , 2002, 40 (6) : 2074–2078 DOI:10.1128/JCM.40.6.2074-2078.2002 |
| [5] | McQuiston JR, Fields PI, Tauxe RV, et al. Do Salmonella carry spare tyres?[J]. Trends Microbiol , 2008, 16 (4) : 142–148 DOI:10.1016/j.tim.2008.01.009 |
| [6] | McClelland M, Sanderson KE, Clifton SW, et al. Comparison of genome degradation in paratyphi A and typhi, human-restricted serovars of Salmonella entericath atcause typhoid[J]. Nat Genet , 2004, 36 (12) : 1268–1274 DOI:10.1038/ng1470 |
 2016, Vol. 37
2016, Vol. 37