文章信息
- 袁雯雯, 杭栋, 王礼华, 陈苏虹, 丁中兴, 胡志斌, 马红霞 .
- Yuan Wenwen, Hang Dong, Wang Lihua, Chen Suhong, Ding Zhongxing, Hu Zhibin, Ma Hongxia .
- microRNA合成通路基因SNP与头颈鳞癌发病风险的关联研究
- Association between genetic variants in microRNA biosynthesis genes and the risk of head and neck squamous cell carcinoma
- 中华流行病学杂志, 2016, 37(8): 1069-1073
- CHINESE JOURNAL OF EPIDEMIOLOGY, 2016, 37(8): 1069-1073
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.08.003
-
文章历史
收稿日期: 2016-03-23
2. 211166 南京, 江苏省恶性肿瘤生物标志物与防治重点实验室、肿瘤个体化医学协同创新中心
2. Jiangsu Key Laboratory of Cancer Biomarkers, Prevention and Treatment, Collaborative Innovation Center for Cancer Personalized Medicine, Nanjing 211166, China
头颈鳞癌是全球第六大常见恶性肿瘤,约占全部肿瘤的6%,每年新发病例超过60万例,其中2/3发生于发展中国家[1]。中国人群头颈鳞癌发病率约为15.2/10万,由于人口基数大,病例绝对数位居世界前列[2]。虽然吸烟和饮酒被公认为头颈鳞癌的主要危险因素[3],但其中仅有小部分暴露者会发生肿瘤,提示遗传易感性可能在头颈鳞癌的发病过程中起到关键作用[4]。基于候选基因方法和全基因组关联研究(GWAS),研究者鉴定了诸如DNA修复通路基因、凋亡通路基因、4q21、6p21、11q12和12q24等区域的头颈鳞癌易感基因[5-7]。然而,这些区域的遗传变异仅能够解释部分头颈鳞癌遗传易感性,即存在遗传度缺失(heredity missing)现象[8]。进一步探索头颈鳞癌的易感性位点有助于揭示其发病机制,并可用于筛选相关高危人群。研究表明,人类1/3的基因表达受微小RNA(microRNA)调控,microRNA通过其特定序列结合靶microRNA,造成mRNA降解或翻译受阻,从而发挥基因调控作用[9]。除参与正常细胞分化、发育等多种生理过程外,microRNA与多种疾病尤其是肿瘤的发生发展密切相关[10]。microRNA合成通路中的基因参与microRNA的加工、剪切及转运,这些基因的特定单核苷酸多态性(SNP)能影响相关基因的表达和或蛋白质功能,从而导致microRNA合成及调控功能的紊乱。已有研究显示,microRNA合成通路基因的多态性位点与肺癌[11]、食管癌[12]、肝癌[13]等的发病风险密切相关。目前国内外鲜见microRNA合成通路基因多态性与头颈鳞癌的关联研究。因此,本研究开展了576例头颈鳞癌患者和1 552名健康对照的病例对照研究,探讨microRNA合成通路关键基因(DICER1、GEMIN3、PIWIL1)的潜在功能性SNP位点与头颈鳞癌易感性之间的相关性,以期发现头颈鳞癌的遗传标志物,为高危人群的筛检和头颈鳞癌早诊早治提供科学依据。
对象与方法1. 研究对象:病例来自2009年1月至2013年5月在江苏省口腔医院等初次就诊的头颈鳞癌患者,均有病理组织学诊断结果,且排除非头颈原发灶的患者。健康对照选自同一时期参与江苏省非传染性疾病筛查项目的30 000多名社区人群,要求对照无肿瘤病史,按年龄(±5岁)和性别与病例组频数匹配。所有受试者均为无血缘关系的汉族人,由专业的调查人员采集病史,通过标准化的调查问卷收集受试者的社会人口学特征和环境暴露史,包括年龄、性别、吸烟、饮酒等。每名受试者均留取5 ml静脉血用于DNA样本提取和基因分型。本研究通过南京医科大学伦理机构审查委员会批准,受试者在招募前均填写知情同意书。
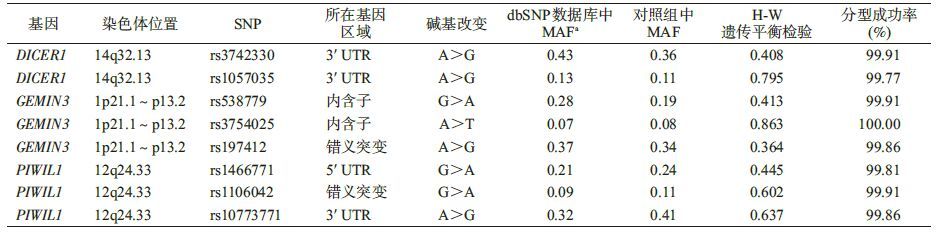
2. SNP筛选与基因分型:目标区域包括基因及其上游10 kb范围,采用生物信息学软件SNPinfo(http://snpinfo.niehs.nih.gov/snpfunc.html)和RegulomeDB数据库(http://regulome.stanford.edu/)筛选具有潜在功能的SNP位点,要求最小等位基因频率(MAF)在中国汉族人群≥5%、r2阈值为0.8以去除高度连锁不平衡位点。最终有8个位点符合筛选条件(表 1)。应用Illumina Infinium BeadChip平台(Illumina Inc)对外周血中提取的DNA样本进行基因分型。所有的SNP基因分型成功率超过99.00%。
3. 统计学方法:采用 χ2检验比较病例组和对照组之间的人口学特征、环境因素以及基因型频率的差异;通过单因素和多因素logistic回归模型评估基因型与肿瘤发生风险之间的关联强度(OR值及其95%CI);拟合优度 χ2比较对照组中观察到的基因型频率与期望频率,从而检验H-W遗传平衡;应用基于 χ2值的Q检验评估亚组间的异质性。统计学分析软件为SAS 9.1,双侧检验P<0.05为差异有统计学意义。
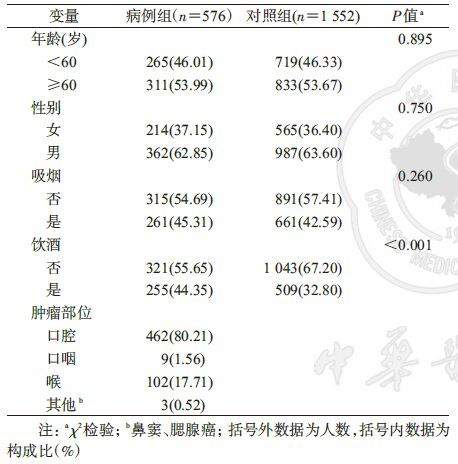
结 果1. 基本情况:在576例头颈鳞癌患者中,462例(80.21%)为口腔癌,9例(1.56%)口咽癌,102例(17.71%)喉癌,3例为鼻窦、腮腺癌(表 2)。病例与对照在吸烟行为方面的差异无统计学意义(P=0.260),但病例组中饮酒者的比例高于对照组,差异有统计学意义(44.35% vs. 32.80%,P<0.001)。
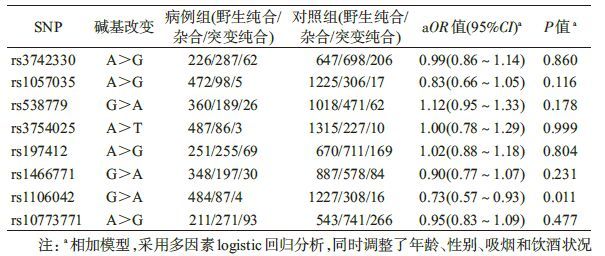
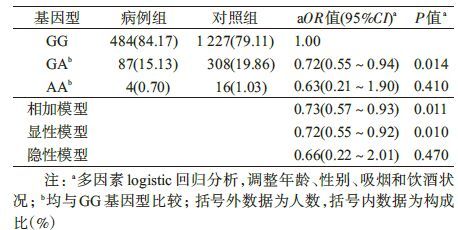
2. SNP位点与头颈鳞癌的关联:所有基因型在对照组中MAF>0.05,且频率分布符合H-W遗传平衡(P>0.05),见表 1。PIWIL1 rs1106042(G>A)的等位基因频率在病例和对照组中差异有统计学意义(P=0.011),见表 3。多因素logistic回归分析显示,rs1106042 A等位基因降低头颈鳞癌的发病风险(相加模型:aOR=0.73,95%CI:0.57~0.93,P=0.011;显性模型:aOR=0.72,95%CI:0.55~0.92,P=0.010),见表 4。经FDR(false discovery rate)校正多重比较,关联结果均无统计学意义。未发现其余7个SNP与头颈鳞癌有统计学关联(表 3)。
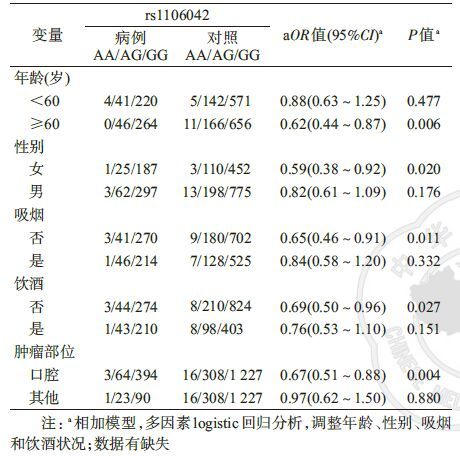
3. 分层分析及交互作用分析:进一步以年龄、性别、吸烟、饮酒及肿瘤部位作为分层因素,基于相加模型分析rs1106042在各亚组与头颈鳞癌的关联(表 5),结果显示,该位点在年龄≥60岁、女性、非吸烟、非饮酒、口腔癌亚组中具有降低肿瘤发病风险的效应(P<0.05)。此外,进一步分析了该位点与吸烟、饮酒等环境因素之间的基因-环境交互作用,以及该位点与其他位点间的位点-位点交互作用,均无统计学意义。
本研究通过病例对照设计探讨了3个microRNA合成通路基因(DICER1、GEMIN3、PIWIL1)上的8个潜在功能性SNP位点与中国人群头颈鳞癌发病风险之间的关联,结果显示,PIWIL1基因rs1106042 G>A可能增加头颈鳞癌尤其是口腔鳞癌的发病风险。
研究表明,microRNA既可作为癌基因参与肿瘤的发生发展,也可作为抑癌基因遏制肿瘤的形成,而microRNA的成熟不良(包括合成过程的中断或者干扰)往往促使正常细胞发生癌变[14]。因此,microRNA合成通路基因的多态性改变可能会阻碍microRNA的成熟,从而间接影响转录或转录后的调控机制,造成个体发病的易感性差异。PIWIL1是RNA诱导基因沉默复合物(RNA-induced silencing complex,RISC)的重要组成部分,microRNA需要与RISC整合形成miRISC后才能负性调控靶基因的表达水平[15]。此外,PIWIL1在干细胞自我更新和配子发育过程中也起到重要作用[16-17]。大量研究证实,PIWIL1在多种恶性肿瘤中呈现异常表达,与肺癌[18]、胃癌[19]、肝癌[20]、结直肠癌[21]等肿瘤的发生发展密切相关,提示PIWIL1可作为一种潜在的新的治疗靶点。已有研究显示,PIWIL1基因的功能性多态位点rs10773771 A>G与HBV阳性肝癌的发病风险降低有关[13]。本研究显示,PIWIL1基因的另一个位点rs1106042 G>A可显著降低头颈鳞癌的发病风险。rs1106042位于PIWIL1基因的外显子区域,该位点G>A的变异可导致错义突变(Arg527Lys) ,可能影响其mRNA前体的剪接方式,产生不同的mRNA剪接异构体,最终影响蛋白的相应功能(http://snpinfo.niehs.nih.gov/snpfunc.htm)。GEMIN3也是RISC组成部分之一,DICER1则主要参与microRNA加工成熟[15]。既往研究显示,GEMIN3 rs197412与结直肠癌发病风险[22]、膀胱癌预后存在关联[23];DICER1 rs3742330与喉癌和结直肠癌的发病风险有关[24-25],rs1057035则影响肝癌和白血病的发病风险[13, 26]。本研究未发现上述DICER1和GEMIN3基因SNP位点与头颈鳞癌发病风险的关联,提示这些位点可能不影响头颈鳞癌易感性。此外,流行病学和分子生物学研究已证明高危型人乳头状瘤病毒(HPV)是头颈鳞癌,尤其是口咽癌的重要危险因素[27]。本研究未检测研究对象的HPV感染情况,因而无法在控制HPV感染的情况下评估PIWIL1 SNP位点的效应。
综上所述,PIWIL1基因rs1106042可能与中国人群头颈鳞癌的发病风险相关。后续有必要采取更大样本的独立研究来验证该遗传标记物在头颈鳞癌高危人群筛选中的应用价值,并确定其具体的生物学功能。
| [1] | Jemal A, Bray F, Center MM, et al. Global cancer statistics[J]. CA Cancer J Clin , 2011, 61 (2) : 69–90 DOI:10.3322/caac.20107 |
| [2] | Franceschi S, Bidoli E, Herrero R, et al. Comparison of cancers of the oral cavity and pharynx worldwide:etiological clues[J]. Oral Oncol , 2000, 36 (1) : 106–115 DOI:10.1016/S1368-8375(99)00070-6 |
| [3] | Rothenberg SM, Ellisen LW. The molecular pathogenesis of head and neck squamous cell carcinoma[J]. J Clin Invest , 2012, 122 (6) : 1951–1957 DOI:10.1172/JCI59889 |
| [4] | Copper MP, Jovanovic A, Nauta JJP, et al. Role of genetic factors in the etiology of squamous cell carcinoma of the head and neck[J]. Arch Otolaryngol Head Neck Surg , 1995, 121 (2) : 157–160 DOI:10.1001/archotol.1995.01890020019005 |
| [5] | Lacko M, Braakhuis BJM, Sturgis EM, et al. Genetic susceptibility to head and neck squamous cell carcinoma[J]. Int J Radiat Oncol Biol Phys , 2014, 89 (1) : 38–48 DOI:10.1016/j.ijrobp.2013.09.034 |
| [6] | Morgan JF. p Value fetishism and use of the Bonferroni adjustment[J]. Evid Based Ment Health , 2007, 10 (2) : 34–35 DOI:10.1136/ebmh.10.2.34 |
| [7] | Lennartsson J, Rönnstrand L. Stem cell factor receptor/c-Kit:from basic science to clinical implications[J]. Physiol Rev , 2012, 92 (4) : 1619–1649 DOI:10.1152/physrev.00046.2011 |
| [8] | Zaitlen N, Kraft P. Heritability in the genome-wide association era[J]. Hum Genet , 2012, 131 (10) : 1655–1664 DOI:10.1007/s00439-012-1199-6 |
| [9] | Bartel DP. MicroRNAs:genomics,biogenesis,mechanism,and function[J]. Cell , 2004, 116 (2) : 281–297 DOI:10.1016/S0092-8674(04)00045-5 |
| [10] | Lu J, Getz G, Miska EA, et al. MicroRNA expression profiles classify human cancers[J]. Nature , 2005, 435 (7043) : 834–838 DOI:10.1038/nature03702 |
| [11] | Pu X, Roth JA, Hildebrandt MAT, et al. MicroRNA-related genetic variants associated with clinical outcomes in early-stage non-small cell lung cancer patients[J]. Cancer Res , 2013, 73 (6) : 1867–1875 DOI:10.1158/0008-5472.CAN-12-0873 |
| [12] | Ye YQ, Wang KK, Gu J, et al. Genetic variations in microRNA-related genes are novel susceptibility loci for esophageal cancer risk[J]. Cancer Prev Res (Phila) , 2008, 1 (6) : 460–469 DOI:10.1158/1940-6207.CAPR-08-0135 |
| [13] | Liu L, An JZ, Liu JB, et al. Potentially functional genetic variants in microRNA processing genes and risk of HBV-related hepatocellular carcinoma[J]. Mol Carcinog , 2013, 52 DOI:10.1002/mc.22062 |
| [14] | Kumar MS, Lu J, Mercer KL, et al. Impaired microRNA processing enhances cellular transformation and tumorigenesis[J]. Nat Genet , 2007, 39 (5) : 673–677 DOI:10.1038/ng2003 |
| [15] | Li CW, Heidt DG, Dalerba P, et al. Identification of pancreatic cancer stem cells[J]. Cancer Res , 2007, 67 (3) : 1030–1037 DOI:10.1158/0008-5472.CAN-06-2030 |
| [16] | Cox DN, Chao AN, Baker J, et al. A novel class of evolutionarily conserved genes defined by PIWI are essential for stem cell self-renewal[J]. Genes Dev , 1998, 12 (23) : 3715–3727 DOI:10.1101/gad.12.23.3715 |
| [17] | Yin H, Lin HF. An epigenetic activation role of PIWI and a PIWI-associated piRNA in Drosophila melanogaster[J]. Nature , 2007, 450 (7167) : 304–308 DOI:10.1038/nature06263 |
| [18] | Navarro A, Tejero R, Viñolas N, et al. The significance of PIWI family expression in human lung embryogenesis and non-small cell lung cancer[J]. Oncotarget , 2015, 6 (31) : 31544–31556 DOI:10.18632/oncotarget.3003 |
| [19] | Liu XY, Sun Y, Guo JP, et al. Expression of HIWI gene in human gastric cancer was associated with proliferation of cancer cells[J]. Int J Cancer , 2006, 118 (8) : 1922–1929 DOI:10.1002/ijc.21575 |
| [20] | Jiang JX, Zhang HJ, Tang QY, et al. Expression of HIWI in human hepatocellular carcinoma[J]. Cell Biochem Biophys , 2011, 61 (1) : 53–58 DOI:10.1007/s12013-011-9160-1 |
| [21] | Raeisossadati R, Abbaszadegan MR, Moghbeli M, et al. Aberrant expression of DPPA2 and HIWI genes in colorectal cancer and their impacts on poor prognosis[J]. Tumour Biol , 2014, 35 (6) : 5299–5305 DOI:10.1007/s13277-014-1690-x |
| [22] | Zhao YF, Du YM, Zhao SN, et al. Single-nucleotide polymorphisms of microRNA processing machinery genes and risk of colorectal cancer[J]. Onco Targets Ther , 2015, 8 : 421–425 DOI:10.2147/OTT.S78647 |
| [23] | Ke HL, Chen M, Ye YQ, et al. Genetic variations in micro RNA biogenesis genes and clinical outcomes in non-muscle-invasive bladder cancer[J]. Carcinogenesis , 2013, 34 (5) : 1006–1011 DOI:10.1093/carcin/bgt006 |
| [24] | Osuch-Wojcikiewicz E, Bruzgielewicz A, Niemczyk K, et al. Association of polymorphic variants of miRNA processing genes with larynx cancer risk in a Polish population[J]. Bio Med Res Int , 2015, 2015 : 298378 DOI:10.1155/2015/298378 |
| [25] | Cho SH, Ko JJ, Kim JO, et al. 3'-UTR polymorphisms in the miRNA machinery genes DROSHA,DICER1,RAN,and XPO5 are associated with colorectal cancer risk in a Korean population[J]. PLoS One , 2015, 10 (7) : e0131125 DOI:10.1371/journal.pone.0131125 |
| [26] | Martin-Guerrero I, Gutierrez-Camino A, Lopez-Lopez E, et al. Genetic variants in miRNA processing genes and pre-miRNAs are associated with the risk of chronic lymphocytic leukemia[J]. PLoS One , 2015, 10 (3) : e0118905 DOI:10.1371/journal.pone.0118905 |
| [27] | zur Hausen H. Papillomaviruses in the causation of human cancers-a brief historical account[J]. Virology , 2009, 384 (2) : 260–265 DOI:10.1016/j.virol.2008.11.046 |
 2016, Vol. 37
2016, Vol. 37