文章信息
- 宋菁, 胡永华.
- Song Jing, Hu Yonghua.
- 精准医疗:分子流行病学的机遇与挑战
- Precision medicine: new opportunities and challenges for molecular epidemiology
- 中华流行病学杂志, 2016, 37(4): 587-592
- Chinese Journal of Epidemiology, 2016, 37(4): 587-592
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.04.030
-
文章历史
- 收稿日期: 2015-11-30
自2003年以来,生物医学界先后完成人类基因组计划(HGP)、人类基因组DNA元件百科全书计划(ENCODE)、基因型-组织表达数据库(GTEx)等一系列具有里程碑意义的工程。在此基础上,2011年美国基因组学与生物医学界智库首次提出“精准医疗(Precision Medicine)”的概念,并规划了 “百万美国人基因组计划(MAGI)”、“2型糖尿病代谢组学项目”、“暴露组学计划”和“电子病历与基因组网络(eMERGE)”等一系列蓝图[1]。2015年伊始,美国宣布将于2016财年拨款2.13亿美元正式启动“精准医疗计划(Precision Medicine Initiative)”。至此,人类初步完成基因组到生物学、生物学到健康、健康到社会的三级跳[2]。作为新型交叉领域,精准医疗的产生和发展离不开生命科学、基础医学、临床医学、流行病学、统计学、社会学和信息学等多学科的共同支撑和促进,而分子流行病学作为流行病学与分子生物学的交叉学科,无疑是促进精准医疗的核心力量。本文将基于精准医疗和分子流行病学各自的特点和研究进展,着重探讨分子流行病学对精准医疗的贡献和意义,并展望精准医疗未来可能的机遇与挑战。
一、精准医疗的兴起1. 精准医疗:是指考虑到个体间基因、环境和生活方式等差异而进行疾病的预防和治疗的新型医疗模式[1]。与过去临床医生根据患者的疾病史、症状体征和实验室检查进行诊疗的传统以症状主导(symptom-oriented)的诊疗方式相比,精准医疗模式根据患者的基因测序及分子标志物信息对患者个体化的诊断、治疗和预防,充分考虑到了个体间的易感性差异和疾病的异质性,其核心是利用系统生物学的有力支持(systems biology-powered)在“个体”水平上对每个患者给予针对性的诊治和预防措施,属于可预测性、可预防性、个性化和参与式的“P4医学”,传统以症状主导的医疗与精准医疗的特点[3, 4]见表 1。
2. 产生与发展背景:回顾精准医疗的历史,其产生发展与基因测序技术、各类组学分析技术以及大数据开发工具的进步息息相关。从1977年Sanger和Coulson[5]发明双脱氧链末端终止法至今,核酸测序技术的发展共经历3代,其测序费用逐年对数递减,而测序速度则呈逐年指数增长趋势,使得快速廉价地获得测序结果成为可能,有利于推动测序技术的临床应用[6, 7, 8, 9]。
除测序技术,基因组学、蛋白组学、代谢组学、表观遗传组学、脂组学、转录组学和宏基因组学等一系列组学分析技术的发展也为精准医疗的产生提供了技术支持,使得疾病的识别和分型更加精准[10, 11, 12, 13, 14, 15, 16, 17]。全基因组关联研究(GWAS)在其蓬勃发展的十年间,已发现200多个疾病或性状关联的共计15 000多个单核苷酸多态性(SNP)位点,发表相关文章共2 000余篇[18, 19]。近年来高通量测序技术使全转录组测序(RNA-seq)、生物大分子质谱分析技术的不断发展,其精确度和灵敏度不断提高,帮助研究人员深入了解复杂的转录和翻译活动,寻找疾病相关或与药物靶点相关的突变分子或结构,并与基因测序信息共同影响癌症等疾病的治疗手段的发展[4]。随着高通量代谢物分析的实现,获得性状或疾病相关的潜在分子图谱成为可能,分子图谱的绘制将大大增加各类疾病分子标志物的诊断灵敏度和特异度[20]。
另外,分子生物学新发现能否顺利转化为精准医疗的临床实践,关键还在于生物大数据工具的开发和利用。目前随着高通量设备的价格持续降低,使用范围越来越广,加上一些公共数据库,如 GenBank、EBI、Uni-Prot等开放,研究人员可通过多种渠道获得丰富的研究数据。然而,数据本身没有任何价值,只有通过大数据挖掘工具对现有的零散数据进行整合、挖掘和分析,才能实现从实验室到临床的转化[21]。目前在生物大数据整合与利用领域已有一些初步成功的案例,如美国临床癌症协会(ASCO)开发的CancerLinQ平台,通过整合和分析癌症患者的基因组信息和电子病历信息,可挖掘出药物副反应、药物敏感性、药物间相互作用等信息,为制定或修订药物指南提供可靠的数据基础[22, 23, 24, 25, 26, 27, 28, 29]。
二、分子流行病学是精准医疗发展的重要方法学1. 分子流行病学:是传统流行病学与分子生物学技术相结合而产生的一个新的流行病学分支,是阐明人群和生物群体中医学相关生物标志的分布及其与疾病/健康的关系和影响因素,并研究防治疾病、促进健康的策略与措施的科学[30]。相对于传统的流行病学主要研究环境、行为等宏观因素的分布规律及其与疾病/健康间的间接关联,分子流行病学以分子生物学技术为工具、以流行病学方法为指导思想,研究的是生物标志的分布规律及其与疾病/健康的直接关系,首要任务为确定和测量从暴露到疾病过程中分子标志物的水平,从微观水平阐明暴露-发病/健康状态连续带的分子机制、将打破传统流行病学的“黑箱”过程、解释疾病的“冰山现象”提供流行病学依据。
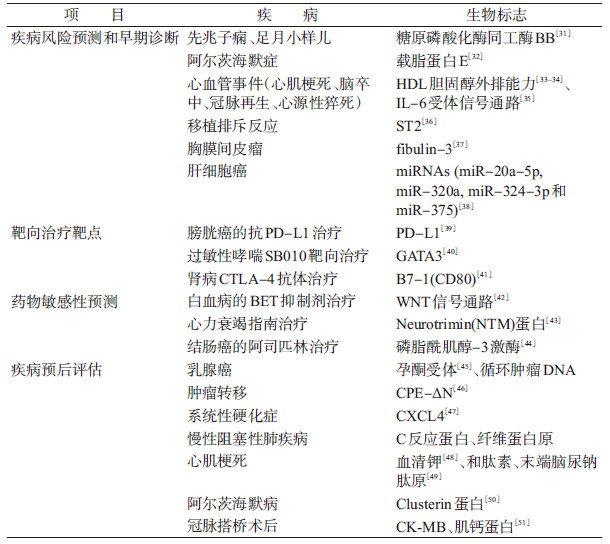
2. 研究进展:随着基因测序技术和组学技术等分子生物学技术的不断提升,分子流行学研究也正在经历飞速发展,研究范围从传染性疾病逐渐转变为以慢性非传染疾病为主的各种疾病和健康状态,研究内容涵盖各类暴露标志、效应标志和易感性标志,用于高危人群筛查、疾病风险预测、疾病早期诊断、药物敏感性检查等(表 2)。
3. 分子流行病学对精准医疗实践的意义:①研究内容:精准医疗的基础是对个体遗传信息及其下游分子标志物的详细解读,而测定人群中疾病/健康相关生物标志的分布及其与疾病/健康状态间的关联,是分子流行病学的重要研究内容。因此在研究内容上分子流行病学和精准医疗一脉相承,二者都需要利用分子生物学技术对生物标志进行测定,并确定与目标疾病或性状相关的分子标志物。分子流行病是通过测定大量个体的分子标志物确定不同人群或亚人群分子标志物的分布规律;而精准医疗则是将上述分布规律应用于个体。因此分子流行病学研究与精准医疗分别属于转化过程的不同阶段,前者为后者提供科学依据,而后者的顺利实施需在前者的基础上进行数据资料的二次分析,并综合考虑利益-风险和成本-效益的平衡,这一过程也是基础研究和流行病学研究转化为临床实践的最大难点[52, 53, 54](图 1)。②研究方法:在采集数据方面,无论分子流行病学研究还是精准医疗的实践都需要利用基因测序技术、各类组学分析技术以及统计学方法采集和处理遗传和生物标志信息。在数据处理方面,精准医疗知识的确立来自对大量基础研究和流行病学研究数据的整合,涉及对现有研究数据进行挖掘和二次分析,所以分子流行病学又为精准医疗提供了数据支持。在研究实施过程中,分子流行病学方法也可以为精准医疗的前期准备提供方法学指导。例如,2015年奥巴马政府的“精准医疗计划”,将建立百万美国人超级队列作为该计划的核心内容,将电子病历系统与DNA、RNA、代谢组和蛋白组等数据连接,用于疾病及用药的监测、评估现有治疗手段在特殊人群的疗效、疾病相关生物标志的识别和疾病风险的评估、预测[55, 56]。③研究意义:分子流行病学作为流行病学的一个分支,其研究目标最终还在于为疾病防治和健康促进策略与措施提供科学依据。而精准医疗作为一门将基础研究转化为临床实践的学科,无疑对于提高诊疗质量、降低成本、提高效益、促进全民健康等方面有里程碑的意义。因此,精准医疗也是实现分子流行病学研究目标的必经之路。
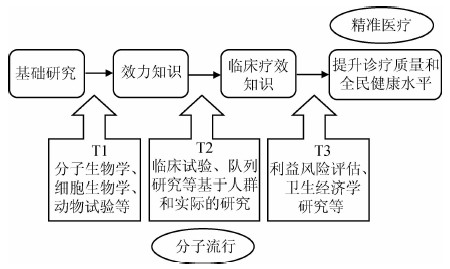
|
| 图 1 “从实验室到临床”转化过程 (根据Dougherty和Conway的3T路线图修改) |
1. 机遇:尽管目前精准医疗刚刚起步,尚未形成独立成熟的学科分支,作为一个极具前瞻性的新领域,现阶段其建设需要通过广泛的基础和流行病学研究为制定诊疗指南提供大量证据,为分子流行病学今后的研究提供了无限机遇[57]。
(1)分子流行病学研究为精准医疗的实现提供人群证据:分子流行病学研究通过对代表性人群中各生物标志物的分布情况进行分析,并根据各生物标记物分布情况将人群划分为多个范围更小的“亚人群”。随着参与疾病分型的遗传信息和生物标志物越来越复杂,“亚人群”不断被缩小,最终获得更加细致的疾病分类,后者是精准医疗实现的基础。以慢性阻塞性肺疾病(COPD)为例,目前已发现至少存在系统性持续炎症反应型、嗜酸细胞和Th2细胞型和细胞间质修复缺陷3个亚型,其中细胞间质修复缺陷亚型的主要由于患者体内缺乏α1AT酶所致,而通过测定患者体内血清α1AT水平、α1AT蛋白质表型和α1AT编码基因型即可对是否属于该亚型进行判断[58]。
(2)分子流行病学研究为精准医疗识别疾病关联基因和位点:精准医疗需要研究者对全基因组内所有可能的致病基因或位点与疾病间的关联进行计算,并阐明其生物学机制。在目前的分子流行病学研究中,我们已经可以通过GWAS等高通量基因检测手段在全基因组范围内同时研究几千个甚至几百万SNP位点,并从中筛选出具有统计学意义(P<10-7)的标签SNP作为疾病或性状相关SNP。在后GWAS时代,我们可以在GWAS研究的基础上,对标签SNP所在的染色体区域进一步精细定位,并利用表达数量性状位点(eQTL)的方法和DNA元件百科全书(ENCODE)等资源实现GWAS特定目标基因和区域的生物学功能研究[59]。
(3)分子流行病学研究为精准医疗建立疾病风险预测模型:精准医疗最终目的不仅在于精确诊断和治疗疾病,更表现在精确预测患者的发病、预后和治疗反应的可能性,相对于传统的临床医学具有更鲜明的公共卫生学意义。在大样本队列研究的基础上,利用分子流行病学方法,如基因评分法等建立包括遗传位点、分子标志物、环境因素和行为因素在内的疾病风险预测模型,并通过统计学方法对模型进行调整实现最优解。相较于传统临床医学中依据医生个人经验和患者疾病史、家族史和症状体征进行粗略的风险评估,分子流行病学方法建立在大量人群多维数据的基础上,疾病风险预测模型的预测能力更强、适用范围更广。
(4)大数据的整合利用:精准医疗的产生与发展离不开医疗大数据资源的挖掘,需要分子流行病学研究者将各种生物大数据、临床大数据和健康大数据资源加以整合并转化为精准医疗的临床实践。这就需要流行病学家积极参与到数据的整理和分析工作中,整合来自不同研究的数据,连接生物大数据与生活方式、环境和临床资料等数据资源。另外,大数据具有容量大、数据类型多、处理速度快和价值高的特点,计算高度复杂性和高度不确定,上述特点决定了大数据的挖掘和分析不同于普通的流行病学研究中的统计学方法,因此需要流行病学和统计学家运用大数据方法对整合后的医疗大数据进行处理,为精准医疗的实践指南提供有力证据[60, 61]。
(5)多学科交叉:精准医疗是一门多学科交叉的领域,需要分子生物学、流行病学、临床医学、信息学、经济学、社会学等多个学科领域的相互交流与协作,要求流行病学研究者同样具备一定的分子生物学、临床医学或信息学知识,在进行分子流行病学研究的时候常常与上述其他领域的专家学者交流合作,共同探讨研究中出现的问题。这有别于过去将临床医学与流行病学甚至公共卫生学分隔开的观念,因此需要包括流行病学家在内的各学科专家学者参与其中,并共同培养出符合精准医疗研究要求的复合型人才。
2. 挑战:任何新生事物总是机遇与挑战并存的,尽管精准医疗带给分子流行病学诸多机遇,我们依然应清醒地意识到,在目前的分子流行病学研究现状下精准医疗的实现仍存在许多问题。
(1)理想的精准医疗的核心是“个体化治疗”,即综合考虑生物标志、基因测序、生理机制、组织病理学、流行病学和药物敏感性等多方面,为每位患者进行“个体”水平的诊疗[58]。而分子流行病学作为一门以人群为基础的学科,其研究结果只能建立在“人群”或“亚人群”的水平上,而非理想中的“个体”水平[54]。
(2)GWAS提供的信息仅能部分预测疾病发生风险,还有一些GWAS尚未能覆盖的区域,如低频或罕见SNP,基因组拷贝数变异和短序列插入和缺失等[59]。有研究表明表观遗传组、转录组和蛋白组学信息以及体力活动、饮食因素的行为因素较基因信息对复杂性疾病发生的影响更大,加上近年来其他软性遗传方式如母婴机制的发现,使得影响人类性状的危险因素更加复杂,无法实现100%预测个体疾病风险[4, 62]。目前常用的方法如基因评分法仅能计算单纯由基因变异所增加的疾病风险,而忽略了基因环境间的交互作用,因此对于复杂性疾病的研究,如糖尿病和心血管疾病,基因评分的方法显得过于片面,其预测准确率也远远达不到精准医疗的要求[63]。
(3)精准医疗对于纳入疾病诊断和靶向治疗的基因标记应具备灵敏度高、特异度高、外显率高、检测具有可操作性以及能够提供普通医学检查无法发现的信息等特征,然而实际研究中几乎不存在这种理想的标志物,尤其是复杂性疾病,通常仅能部分预测疾病发生风险增加或用药的不同反应[62]。因此,如何建立众多微效基因的疾病风险预测模型计算疾病风险也是分子流行病学家目前面临的主要问题之一。
(4)流行病学家如何整合不同来源和平台的研究数据,以及如何将基因测序为代表生物大数据与生活方式、环境和临床等数据资源相互连接,这既需要计算机硬件支持,也需要技术上的支持,还需要相关政策出台促进公共卫生和个体化医疗的专家学者通力合作共享数据。另外,大数据的挖掘和分析不同于普通的流行病学研究中的统计学方法,在利用的过程中可能面临诸如虚假关联、生态学谬论、假阳性信号以及无法解决复杂疾病涉及多个基因通路交互作用等问题,需要开发适用于生物大数据的统计学方法和工具[64]。
综上所述,在精准医疗的大背景下,分子流行病学未来面临的机遇大于挑战,随着分子流行病学技术的发展,上述发展中的问题将会被逐个解决,并且在精准医疗掀起的浪潮下,分子流行病学研究也将迎来新的时代。
利益冲突 无| [1] New NRCU,Disease TO. Toward precision medicine:building a knowledge network for biomedical research and a new taxonomy of disease[M]. Washington,DC:National Academies Press(US),2011. |
| [2] Collins FS,Green ED,Guttmacher AE,et al. A vision for the future of genomics research[J]. Nature,2003,422(6934):835-847. DOI:10.1038/nature01626. |
| [3] Horgan D,Paradiso A,McVie G,et al. Is precision medicine the route to a healthy world?[J]. Lancet,2015,386(9991):336-337. DOI:10.1016/S0140-6736(15)61404-0. |
| [4] Chen R,Snyder M. Promise of personalized omics to precision medicine[J]. Wiley Interdiscip Rev Syst Biol Med,2013,5(1):73-82. DOI:10.1002/wsbm.1198. |
| [5] Sanger F,Coulson AR. A rapid method for determining sequences in DNA by primed synthesis with DNA polymerase[J]. J Mol Biol,1975,94(3):441-448. DOI:10.1016/0022-2836(75)90213-2. |
| [6] Hayden EC. Gene sequencing leaves the laboratory[J]. Nature,2013,494(7437):290-291. DOI:10.1038/494290a. |
| [7] Hayden EC. Technology:The $1,000 genome[J]. Nature,2014,507(7492):294-295. DOI:10.1038/507294a. |
| [8] Sboner A,Mu XJ,Greenbaum D,et al. The real cost of sequencing:higher than you think![J]. Genome Biol,2011,12(8):125. DOI:10.1186/gb-2011-12-8-125. |
| [9] Shendure J,Mitra RD,Varma C,et al. Advanced sequencing technologies:methods and goals[J]. Nat Rev Genet,2004,5(5):335-344. DOI:10.1038/nrg1325. |
| [10] Gu LC,Li C,Aach J,et al. Multiplex single-molecule interaction profiling of DNA-barcoded proteins[J]. Nature,2014,515(7528):554-557. DOI:10.1038/nature13761. |
| [11] Macaulay IC,Haerty W,Kumar P,et al. G&T-seq:parallel sequencing of single-cell genomes and transcriptomes[J]. Nat Methods,2015,12(6):519-522. DOI:10.1038/nmeth.3370. |
| [12] Gika HG,Theodoridis GA,Plumb RS,et al. Current practice of liquid chromatography-mass spectrometry in metabolomics and metabonomics[J]. J Pharm Biomed Anal,2014,87:12-25. DOI:10.1016/j.jpba.2013.06.032. |
| [13] Couldrey C,Cave V. Assessing DNA methylation levels in animals:choosing the right tool for the job[J]. Anim Genet,2014,45 Suppl 1:15-24. DOI:10.1111/age.12186. |
| [14] Ow TJ,Upadhyay K,Belbin TJ,et al. Bioinformatics in otolaryngology research. Part two:other high-throughput platforms in genomics and epigenetics[J]. J Laryngol Otol,2014,128(11):942-947. DOI:10.1017/S0022215114002011. |
| [15] Burgess DJ. Technology:Bead capture for single-cell transcriptomics[J]. Nat Rev Genet,2015,16(4):195. DOI:10. 1038/nrg3919. |
| [16] Liu Y,Xu HM,Chen SC,et al. Genome-wide interaction-based association analysis identified multiple new susceptibility Loci for common diseases[J]. PLoS Genet,2011,7(3):e1001338. DOI:10.1371/journal.pgen.1001338. |
| [17] Qin JJ,Li YR,Cai ZM,et al. A metagenome-wide association study of gut microbiota in type 2 diabetes[J]. Nature,2012,490(7418):55-60. DOI:10.1038/nature11450. |
| [18] Wang LL,Matsushita T,Madireddy L,et al. PINBPA:cytoscape app for network analysis of GWAS data[J]. Bioinformatics,2015,31(2):262-264. DOI:10.1093/bioinformatics/btu644. |
| [19] Klein RJ,Zeiss C,Chew EY,et al. Complement factor H polymorphism in age-related macular degeneration[J]. Science,2005,308(5720):385-389. DOI:10.1126/science.1109557. |
| [20] Suhre K,Shin SY,Petersen AK,et al. Human metabolic individuality in biomedical and pharmaceutical research[J]. Nature,2011,477(7362):54-60. DOI:10.1038/nature10354. |
| [21] Borangíu T,Purcarea V. The future of healthcare-information based medicine[J]. J Med Life,2008,1(2):233-237. |
| [22] Afendi FM,Ono N,Nakamura Y,et al. Data mining methods for omics and knowledge of crude medicinal plants toward big data biology[J]. Comput Struct Biotechnol J,2013,4(5):1-14. DOI:10.5936/csbj.201301010. |
| [23] Cully M. Anticancer drugs:Advancing precision medicine in silico[J]. Nat Rev Drug Discov,2015,14(5):311. DOI:10.1038/nrd4619. |
| [24] Şardaş S,Endrenyi L,Gürsoy UK,et al. A call for pharmacogenovigilance and rapid falsification in the age of big data:why not first road test your biomarker?[J]. OMICS,2014,18(11):663-665. DOI:10.1089/omi.2014.0132. |
| [25] Mcintyre RS,Cha DS,Jerrell JM,et al. Advancing biomarker research:utilizing‘Big Data’approaches for the characterization and prevention of bipolar disorder[J]. Bipolar Disord,2014,16(5):531-547. DOI:10.1111/bdi.12162. |
| [26] Shaikh AR,Butte AJ,Schully SD,et al. Collaborative biomedicine in the age of big data:the case of cancer[J]. J Med Internet Res,2014,16(4):e101. DOI:10.2196/jmir.2496. |
| [27] Costa FF. Big data in biomedicine[J]. Drug Discov Today,2014,19(4):433-440. DOI:10.1016/j.drudis.2013.10.012. |
| [28] Hay SI,George DB,Moyes CL,et al. Big data opportunities for global infectious disease surveillance[J]. PLoS Med,2013,10(4):e1001413. DOI:10.1371/journal.pmed.1001413. |
| [29] Savage N. Bioinformatics:Big data versus the big C[J]. Nature,2014,509(7502):S66-67. DOI:10.1038/509S66a. |
| [30] 詹思延,叶冬青,谭红专. 流行病学[M]. 7版. 北京:人民卫生出版社,2012:292. Zhan SY,Ye DQ,Tan HZ. Epidemiology[M]. 7ed. Beijing:People's Medical Publishing House,2012:292. |
| [31] McCarthy F,Doyle A,Khashan A,et al. Assessment of glycogen phosphorylase isoenzyme BB as a biomarker for pre-eclampsia and small for gestational age[J]. Lancet,2015,385 Suppl 1:S67. DOI:10.1016/S0140-6736(15)60382-8. |
| [32] Jansen WJ,Ossenkoppele R,Knol DL,et al. Prevalence of cerebral amyloid pathology in persons without dementia:a meta-analysis[J]. JAMA,2015,313(19):1924-1938. DOI:10.1001/jama.2015.4668. |
| [33] Rohatgi A,Khera A,Berry JD,et al. HDL cholesterol efflux capacity and incident cardiovascular events[J]. N Engl J Med,2014,371(25):2383-2393. DOI:10.1056/NEJMoa1409065. |
| [34] Voight BF,Peloso GM,Orho-Melander M,et al. Plasma HDL cholesterol and risk of myocardial infarction:a mendelian randomisation study[J]. Lancet,2012,380(9841):572-580. DOI:10.1016/S0140-6736(12)60312-2. |
| [35] Sarwar N,Butterworth AS,Freitag DF,et al. Interleukin-6 receptor pathways in coronary heart disease:a collaborative meta-analysis of 82 studies[J]. Lancet,2012,379(9822):1205-1213. DOI:10.1016/S0140-6736(11)61931-4. |
| [36] Vander Lugt MT,Braun TM,Hanash S,et al. ST2 as a marker for risk of therapy-resistant graft-versus-host disease and death[J]. N Engl J Med,2013,369(6):529-539. DOI:10.1056/NEJMoa1213299. |
| [37] Pass HI,Levin SM,Harbut MR,et al. Fibulin-3 as a blood and effusion biomarker for pleural mesothelioma[J]. N Engl J Med,2012,367(15):1417-1427. DOI:10.1056/NEJMoa1115050. |
| [38] Wen Y,Han J,Chen JG,et al. Plasma miRNAs as early biomarkers for detecting hepatocellular carcinoma[J]. Int J Cancer,2015,137(7):1679-1690. DOI:10.1002/ijc.29544. |
| [39] Powles T,Eder JP,Fine GD,et al. MPDL3280A(anti-PD-L1) treatment leads to clinical activity in metastatic bladder cancer[J]. Nature,2014,515(7528):558-562. DOI:10.1038/nature 13904. |
| [40] Krug N,Hohlfeld JM,Kirsten AM,et al. Allergen-induced asthmatic responses modified by a GATA3-specific DNAzyme[J]. N Engl J Med,2015,372(21):1987-1995. DOI:10.1056/NEJMoa1411776. |
| [41] Yu CC,Fornoni A,Weins A,et al. Abatacept in B7-1-positive proteinuric kidney disease[J]. N Engl J Med,2013,369(25):2416-2423. DOI:10.1056/NEJMoa1304572. |
| [42] Rathert P,Roth M,Neumann T,et al. Transcriptional plasticity promotes primary and acquired resistance to BET inhibition[J]. Nature,2015,525(7570):543-547. DOI:10.1038/nature14898. |
| [43] Cao TH,Quinn PA,Sandhu JK,et al. Identification of novel biomarkers in plasma for prediction of treatment response in patients with heart failure[J]. Lancet,2015,385 Suppl 1:S26. DOI:10.1016/S0140-6736(15)60341-5. |
| [44] Liao X,Lochhead P,Nishihara R,et al. Aspirin use,tumor PIK3CA mutation,and colorectal-cancer survival[J]. N Engl J Med,2012,367(17):1596-1606. DOI:10.1056/NEJMoa1207756. |
| [45] Mohammed H,Russell IA,Stark R,et al. Progesterone receptor modulates ERα action in breast cancer[J]. Nature,2015,523(7560):313-317. DOI:10.1038/nature14583. |
| [46] Mitka M. Researchers discover new biomarker that may improve cancer care strategies[J]. JAMA,2011,305(12):1184-1185. DOI:10.1001/jama.2011.315. |
| [47] van Bon L,Affandi AJ,Broen J,et al. Proteome-wide analysis and CXCL4 as a biomarker in systemic sclerosis[J]. N Engl J Med,2014,370(5):433-443. DOI:10.1056/NEJMoa1114576. |
| [48] Goyal A,Spertus JA,Gosch K,et al. Serum potassium levels and mortality in acute myocardial infarction[J]. JAMA,2012,307(2):157-164. DOI:10.1001/jama.2011.1967. |
| [49] Alehagen U,Dahlstrom U,Rehfeld JF,et al. Association of copeptin and N-terminal proBNP concentrations with risk of cardiovascular death in older patients with symptoms of heart failure[J]. JAMA,2011,305(20):2088-2095. DOI:10.1001/jama.2011.666. |
| [50] Schrijvers EM,Koudstaal PJ,Hofman A,et al. Plasma clusterin and the risk of Alzheimer disease[J]. JAMA,2011,305(13):1322-1326. DOI:10.1001/jama.2011.381. |
| [51] Domanski MJ,Mahaffey K,Hasselblad V,et al. Association of myocardial enzyme elevation and survival following coronary artery bypass graft surgery[J]. JAMA,2011,305(6):585-591. DOI:10.1001/jama.2011.99. |
| [52] Dougherty D,Conway PH. The "3T's" road map to transform US health care:the "how" of high-quality care[J]. JAMA,2008,299(19):2319-2321. DOI:10.1001/jama.299.19.2319. |
| [53] Ioannidis JP,Panagiotou OA. Comparison of effect sizes associated with biomarkers reported in highly cited individual articles and in subsequent meta-analyses[J]. JAMA,2011,305(21):2200-2210. DOI:10.1001/jama.2011.713. |
| [54] Ashley EA. Promise of precision medicine-Reply[J]. JAMA,2015,314(16):1752-1753. DOI:10.1001/jama.2015.11197. |
| [55] Collins FS,Varmus H. A new initiative on precision medicine[J]. N Engl J Med,2015,372(9):793-795. DOI:10.1056/NEJMp1500523. |
| [56] Ashley EA. The precision medicine initiative:a new national effort[J]. JAMA,2015,313(21):2119-2120. DOI:10.1001/jama.2015.3595. |
| [57] Bayer R,Galea S. Public health in the precision-medicine era[J]. N Engl J Med,2015,373(6):499-501. DOI:10.1056/NEJMp 1506241. |
| [58] Woodruff PG,Agusti A,Roche N,et al. Current concepts in targeting chronic obstructive pulmonary disease pharmacotherapy:making progress towards personalised management[J]. Lancet,2015,385(9979):1789-1798. DOI:10.1016/S0140-6736(15)60693-6. |
| [59] 沈洪兵. 后全基因组关联研究时代的基因组流行病学研究策略[J]. 中华预防医学杂志,2013,47(5):389-390. DOI:10.3760/cma.j.issn.0253-9624.2013.05.001. Shen HB. Chin J Prev Med,2013,47(5):389-390. DOI:10.3760/cma.j.issn.0253-9624.2013.05.001. |
| [60] 宁康,陈挺. 生物医学大数据的现状与展望[J]. 科学通报,2015,60(5/6):534-546. DOI:10.1360/N972014-00895. Ning K,Chen T. Big data for biomedical research:Current status and prospective[J]. Chin Sci Bull,2015,60(5/6):534-546. DOI:10.1360/N972014-00895. |
| [61] 王波,吕筠,李立明. 生物医学大数据:现状与展望[J]. 中华流行病学杂志,2014,35(6):617-620. DOI:10.3760/cma.j.issn. 0254-6450.2014.06.001. Wang B,Lyu J,Li LM. Big data in biomedicine:status quo and perspective[J]. Chin J Epidemiol,2014,35(6):617-620. DOI:10.3760/cma.j.issn.0254-6450.2014.06.001. |
| [62] Coote JH,Joyner MJ. Is precision medicine the route to a healthy world?[J]. Lancet,2015,385(9978):1617. DOI:10.1016/S0140-6736(15)60786-3. |
| [63] Joyner MJ,Prendergast FG. Chasing Mendel:five questions for personalized medicine[J]. J Physiol,2014,592(Pt 11):2381-2388. DOI:10.1113/jphysiol.2014.272336. |
| [64] Khoury MJ,Ioannidis JP. Medicine. Big data meets public health[J]. Science,2014,346(6213):1054-1055. DOI:10.1126/science. aaa2709. |
 2016, Vol. 37
2016, Vol. 37