文章信息
- 陈炜, 翁育伟, 何文祥, 朱颖, 张拥军, 黄萌, 谢剑锋, 郑奎城, 严延生.
- Chen Wei, Weng Yuwei, He Wenxiang, Zhu Ying, Zhang Yongjun, Huang Meng, Xie Jianfeng, Zheng Kuicheng, Yan Yansheng.
- 福建省2011-2014年手足口病相关病原柯萨奇病毒A组10型的分子流行病学研究
- Molecular epidemiology of hand-foot-mouth disease associated pathogen Coxsackievirus A10 identified in Fujian province, 2011-2014
- 中华流行病学杂志, 2016, 37(4): 563-567
- Chinese Journal of Epidemiology, 2016, 37(4): 563-567
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.04.025
-
文章历史
- 收稿日期: 2015-10-22
引起手足口病的主要肠道病毒为肠道病毒71型(EV71)及柯萨奇病毒A 组16 型(Cox A16),其他肠道病毒(非EV71及非Cox A16的肠道病毒)占一定的比例[1, 2]。2008年5月手足口病在中国大陆地区大规模流行[3, 4],同年5月2日,手足口病正式被纳入丙类法定传染病进行管理。福建省手足口病疫情监测数据显示,2008-2010年引起手足口病的主要病原体为EV71及Cox A16,但是近几年其他肠道病毒的构成比例明显上升(2011-2014年分别为26.5%、32.2%、65.7%、41.8%)。因此,本研究在对2011-2014年福建省部分手足口病患者感染的其他肠道病毒进行分型鉴定的基础上,对优势血清型之一的柯萨奇病毒A 组10型(Cox A10)开展分子流行病学研究。
材料与方法1. 标本来源:利用福建省手足口病实验室监测系统,收集2011-2014年福建省4个沿海城市(福州、泉州、漳州、宁德)及2个内陆城市(龙岩、南平)其他肠道病毒手足口病标本,共计1 525例其他肠道病毒手足口病病例的标本(其中2011年268例、2012年622例、2013年450例、2014年182例),包括咽拭子和肛拭子等。
2. 病毒培养分离:对上述标本分别用RD细胞和Hep-2细胞(均由中国CDC病毒病预防控制所脊髓灰质炎实验室提供)进行培养分离,收获细胞培养上清。具体操作按照《手足口病实验室手册》[5]标准和步骤进行。
3. 病毒RNA提取:采用德国Qiagen公司的QIAamp viral RNA mini kit提取病毒RNA,按照试剂盒说明书进行操作。从140 μl的细胞培养上清获得50 μl的总RNA溶液。
4. 分型鉴定:上述RNA样本均采用中国CDC病毒病预防控制所脊髓灰质炎实验室推荐的肠道病毒通用引物PE1/PE2进行肠道病毒的分子生物学鉴定[5]。采用Qiagen公司的Onestep RT-PCR试剂盒进行RT-PCR反应,反应条件:50 ℃ 30 min,95 ℃ 15 min; 94 ℃ 30 s,50 ℃ 30 s,72 ℃ 30 s,35个循环;72 ℃ 10 min。取5 μl的PCR产物在1.5%琼脂糖凝胶电泳鉴定。
肠道病毒阳性的RNA样本采用美国Promega公司Access one step RT-PCR试剂盒通过292/222引物进行RT-PCR反应[6],反应条件:45 ℃ 45 min,94 ℃ 2 min;94 ℃ 30 s,45 ℃ 30 s,68 ℃ 50 s,40个循环;68 ℃ 10 min。琼脂糖凝胶电泳阳性的PCR产物进行胶回收并纯化测序,获得的序列通过网站http://www.rivm.nl/mpf/enterovirus/typingtool进行分型鉴定,确定病毒血清型别。
5. VP1区基因序列的获得:对确定为血清型Cox A10的RNA样本采用Qiagen公司的Onestep RT-PCR试剂盒应用A组肠道病毒特异性引物486/488、487/489分段进行完整VP1区基因片段的扩增[7],反应条件:50 ℃ 30 min,95 ℃ 15 min;94 ℃ 30 s,50 ℃ 30 s,72 ℃ 60 s,40个循环;72 ℃ 10 min。琼脂糖凝胶电泳阳性的PCR产物进行胶回收纯化测序。
6. 序列分析:核苷酸和氨基酸序列分析和同源性比较应用DNAStar软件完成,应用Mega 6.0软件的邻接(N-J)法构建系统进化树。
结 果1. 一般情况:共收集1 525例病例的其他肠道病毒标本,对标本分别进行两种细胞(RD/Hep-2细胞)两个代次的细胞培养分离,共获得407例其他肠道病毒病例毒株,其中103例鉴定为Cox A10,构成比(25.3%)仅次于柯萨奇病毒A组6型(Cox A6)(48.9%)。
2. Cox A10流行病学特征:
(1)时间分布:2011-2014年Cox A10病例在其他肠道病毒病例的年度构成比分别为41.6%(32/77)、18.5%(38/205)、28.0%(26/93)和21.9%(7/32),将数据并入疫情监测系统所得到的手足口病实验室检测数据(包括手足口病EV71和Cox A16监测数据)进行综合统计分析,福建省手足口病的病原谱在2008-2010年以EV71和Cox A16为主,EV71所占的比重最高,占到75%以上。从2011年开始发生较大变化,EV71及Cox A16的比重下降,Cox A6、Cox A10等在病原谱的比重增加。2011-2014年,EV71、Cox A16、Cox A6及Cox A10成为手足口病的主要病原。见图 1。
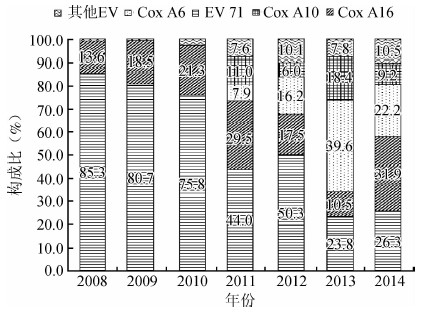
|
| 图 1 2008-2014年福建省手足口病病原谱年度变化情况 |
(2)地区分布:通过对2011-2014年福建省4个沿海城市(福州、泉州、漳州、宁德)、2个内陆城市(龙岩和南平)及全省的手足口病病原谱分析,6个城市至全省的2011-2014年手足口病的病原谱均发生较大变化,EV71及Cox A16构成比降低,Cox A6及Cox A10构成比增加,成为引起手足口病的主要病原。2011-2014年福建全省手足口病病原谱构成中,Cox A10所占的比重为11.0%,见图 2。
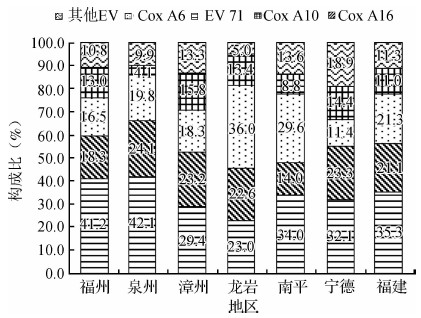
|
| 图 2 2011-2014年福建省及部分城市手足口病病原谱 |
(3)人群分布:103例的Cox A10手足口病病例中,男性病例68例,女性病例35例,男女性别比为1.9:1,与2008-2014年福建省手足口病总体疫情中的男女发病情况(男女性别比约为1.7:1,342 708/ 202 172)相比,差异无统计学意义(χ2=0.43,P=0.51>0.05)。
将103例的Cox A10手足口病病例按照0~、1~、2~、3~、4~、≥5岁6个年龄组进行分析,其病例数年龄分布与总体手足口病年龄分布接近,主要集中在≤4岁的幼儿,占总病例数的97.1%(100/103),与总体手足病病例中≤4岁的患儿所占的比例(94.3%,514 000/544 880)相比,差异无统计学意义( χ2=1.46,P=0.23>0.05)。其中1~岁组所占比例最高,为39.8%(41/103),各个年龄组的例数分布及构成情况见图 3。
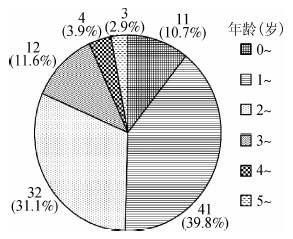
|
| 注:括号外数据为例数,括号内数据为构成比(%) 图 3 Cox A10手足口病病例的年龄组分布 |
(4)病例类型分布:103例Cox A10手足口病病例中,普通病例92例,重症病例11例,无死亡病例。Cox A10感染导致重症和死亡的频率为10.7%(11/103),与手足口病实验室监测系统得到的EV71感染导致重症和死亡的频率(16.3%,1 376/8 449)相比,差异无统计学意义(χ2=2.35,P=0.13>0.05),与Cox A16(3.2%,115/3 630)相比,差异有统计学意义(χ2=15.10,P=0.00<0.05)。
3. VP1区基因序列分析:对103株Cox A10分离株进行完整VP1区片段的扩增测序,获得51株Cox A10(其中2011年11株,2012年21株,2013年13株,2014年6株)的完整VP1区基因序列,VP1区基因序列全长894 bp,与原型株(Cox A10-Kowalik)相比,未发现核苷酸的缺失和插入。
2011-2014年福建省的51株Cox A10分离株的VP1核苷酸序列同源性在93.9%~100.0%之间,氨基酸序列同源性在96.6%~100.0%之间。与原型株(Cox A10-Kowalik)的核苷酸序列同源性在76.0%~77.1%之间,氨基酸序列同源性在91.9%~93.6%之间。与近几年国内山东、云南、安徽、湖南、河北、深圳等地分离株的VP1核苷酸序列同源性在93.8%~99.7%之间,氨基酸序列同源性在96.9%~100.0%之间。与2008-2010年国外法国、德国、西班牙、日本等地方分离株的VP1核苷酸序列同源性在79.9%~95.7%之间,氨基酸序列同源性在92.6%~100.0%之间。
将VP1完整编码区翻译成氨基酸序列进行分析,结果显示,2011-2014年福建省的51株Cox A10分离株氨基酸序列的第14、22、23、141、142、240、283和284位置,是氨基酸变异的频繁区域。
4. 遗传进化分析:将2011-2014年的51株Cox A10福建分离株与从GenBank检索37株Cox A10型病毒代表株构建基于完整VP1区基因以柯萨奇病毒A组12型(Cox A12)原型株为外群的的Cox A10系统进化树(图 4)。进化树分析显示,2011-2014年51株Cox A10福建分离株及37株从GenBank下载的Cox A10型代表株,至少可以划分成7个进化分支(A~G),G分支又分为G1~G4分支。2011-2014年福建省的51株Cox A10分离株分布于G2、G3、G4亚分支,其中仅有1株(2011FJFZ024)分布于G2亚分支,有4株(2011FJLY173、2011FJNP021、2012FJFZ676、2012FJNP339)分布于G3亚分支,其余46株分离株集中于G4分支中。2011-2014年51株Cox A10福建分离株与原型株(Cox A10-Kowalik)及2008-2010年国外地区分离株的遗传距离较远,亲缘关系远,与2010-2014年国内地区(如山东、深圳、河北、云南等省份)分离株的遗传距离近,亲缘关系近。51株Cox A10福建分离株相互之间的遗传距离较近,亲缘关系近。同时,从进化树显示,福建省各个地区的Cox A10分离株没有明显的集中于某个分支或某一簇,而是分散于各个分支中。
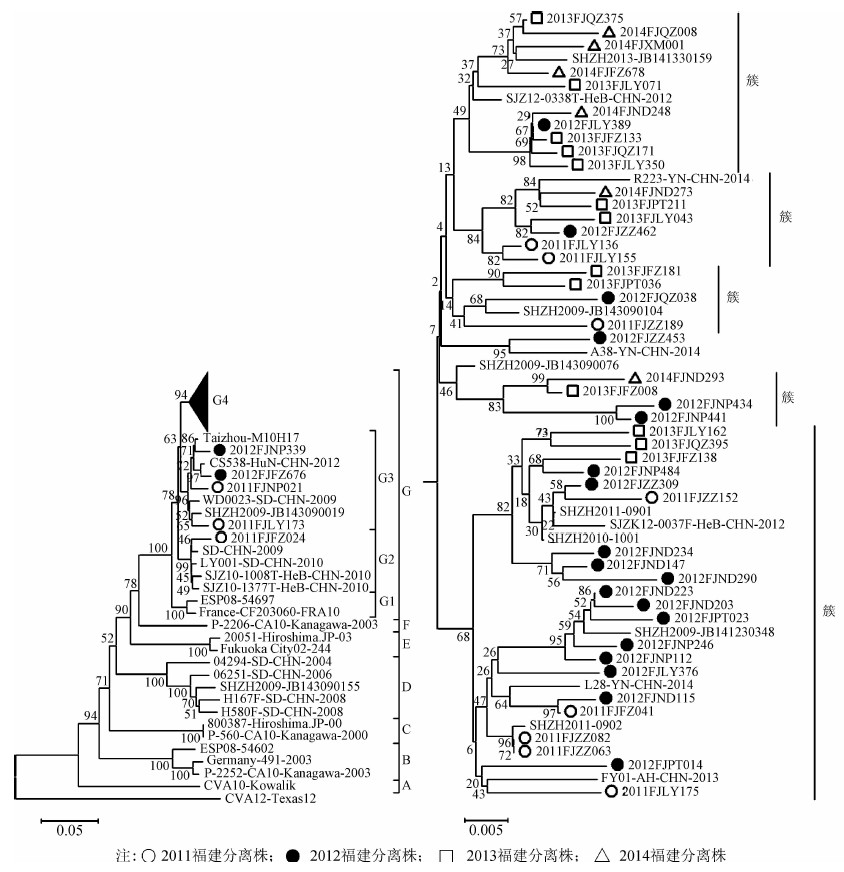
|
| 图 4 福建省Cox A10病毒分离株VP1区基因序列系统进化树 |
本研究对2011-2014年福建省1 525例病例的其他肠道病毒标本进行病毒培养后分型鉴定,共分型鉴定出407例其他肠道病毒病例,其中Cox A10构成比仅次于Cox A6。
本研究对103例Cox A10手足口病病例的时间、空间、人群、病例类型分布等流行病学特征分析。时间分布分析显示,福建省手足口病病原谱在2008-2010年期间变化不明显,以EV71和Cox A16为主,从2011年开始发生较大变化,EV71及Cox A16的比重下降,Cox A6、Cox A10等其他肠道病毒在病原谱的比重增加,与EV71及Cox A16共同成为近些年手足病流行的主要型别。这与2008-2012年国内外有关手足口病病原谱构成变化的报道相似,Cox A6及Cox A10逐渐成为手足口病主要及重要的病原体[8, 9, 10, 11, 12, 13, 14, 15, 16]。地区分布分析显示,2011-2014年福建省各个地区均发生相似的手足口病病原谱转换,主要体现在Cox A6、Cox A10逐渐成为手足口病重要的病原之一。人群分布分析显示,Cox A10在不同性别、不同年龄组的分布特征与手足口病总体疫情相似,没有明显特异性。病例类型分布分析显示,Cox A10与EV71相似,其感染导致重症至死亡手足口病的频率相对较高,其验证及其原因有待进一步的研究。
VP1蛋白不仅是病毒中和决定因子,直接影响到病毒的抗原性,而且VP1基因具有与病毒血清型完全对应的遗传多样性[17]。本研究扩增VP1区基因部分片段以鉴定福建省其他肠道病毒,及扩增完整VP1区基因片段以分析福建省其他肠道病毒的分子流行病特征。通过对2011-2014年的51株Cox A10福建分离株的VP1区基因序列进行分析,51株Cox A10福建分离株与Cox A10原型株(Cox A10-Kowalik)在核苷酸序列及氨基酸序列的差异均较大。从系统进化树分析,2011-2014年51株Cox A10中国福建省分离株与原型株(Cox A10-Kowalik)及2008-2010年国外地区分离株的遗传距离较远,亲缘关系远,与2010-2014年国内地区分离株的遗传距离近,亲缘关系近。可见,2011-2014年Cox A10福建分离株在进化上并不是独立的,而是与国内地区的Cox A10分离株同进化共循环的。2011-2014年福建省的51株Cox A10分离株,分布于G2、G3、G4亚分支,福建省各个地区的Cox A10分离株没有明显地集中于某个分支或某一簇,而是分散于各个分支中,G4亚分支则为福建省Cox A10分离株的主要流行分支。
利益冲突 无| [1] 刘克洲,陈智. 人类病毒性疾病[M]. 2版. 北京:人民卫生出版社,2010:624-628. Liu KZ,Chen Z. Human viral disease[M]. 2nd ed. Beijing:People's Medical Publishing House,2010:624-628. |
| [2] Singh S,Poh CL,Chow VTK. Complete sequence analyses of enterovirus 71 strains from fatal and non-fatal cases of the hand,foot and mouth disease outbreak in Singapore(2000)[J]. Microbiol Immunol,2002,46(11):801-808. DOI:10.1111/j. 1348-0421.2002.tb02767.x. |
| [3] Zhang Y,Zhu Z,Yang WZ,et al. An emerging recombinant human enterovirus 71 responsible for the 2008 outbreak of hand foot and mouth disease in Fuyang city of China[J]. Virol J,2010,7:94. DOI:10.1186/1743-422X-7-94. |
| [4] Yang F,Ren LL,Xiong ZH,et al. Enterovirus 71 outbreak in the People's Republic of China in 2008[J]. J Clin Microbiol,2009,47(7):2351-2352. DOI:10.1128/JCM.00563-09. |
| [5] 国家脊髓灰质炎和国家麻疹实验室. 手足口病实验室手册[R]. 4版. 北京:中国疾病预防控制中心,2010:20-26. National of Polio and National Measles Laboratory. HFMD laboratory manual[R]. 4th ed. Beijing:Chinese Center for Disease Control and Prevention,2010:20-26. |
| [6] Obereste MS,Nix WA,Mahe K,et al. Improved molecular identification of enteroviruses by RT-PCR and amplicon sequencing[J]. J Clin Virol,2003,26(3):375-377. DOI:10. 1016/S1386-6532(03)00004-0. |
| [7] Oberste MS,Maher K,Williams AJ,et al. Species-specific RT-PCR amplification of human enteroviruses:a tool for rapid species identification of uncharacterized enteroviruses[J]. J Gen Virol,2006,87(1):119-128. DOI:10.1099/vir.0.81179-0. |
| [8] Wu Y,Yeo A,Phoon MC,et al. The largest outbreak of hand; foot and mouth disease in Singapore in 2008:the role of enterovirus 71 and coxsackievirus A strains[J]. Int J Infect Dis,2010,14(12):e1076-1081. DOI:10.1016/j.ijid.2010.07.006. |
| [9] Österback R,Vuorinen T,Linna M,et al. Coxsackievirus A6 and hand,foot,and mouth disease,Finland[J]. Emerg Infect Dis,2009,15(9):1485-1488. DOI:10.1128/genomeA.01004-14. |
| [10] Davia JL,Bel PH,Ninet VZ,et al. Onychomadesis outbreak in Valencia,Spain associated with hand,foot,and mouth disease caused by enteroviruses[J]. Pediatr Dermatol,2011,28(1):1-5. DOI:10.1111/j.1525-1470.2010.01161.x. |
| [11] Mirand A,Henquell C,Archimbaud C,et al. Outbreak of hand,foot and mouth disease/herpangina associated with coxsackievirus A6 and A10 infections in 2010,France:a large citywide,prospective observational study[J]. Clin Microbiol Infect,2012,18(5):E110-118. DOI:10.1111/j.1469-0691. 2012.03789.x. |
| [12] Blomqvista S,Klemola P,Kaijalainen S,et al. Co-circulation of coxsackieviruses A6 and A10 in hand,foot and mouth disease outbreak in Finland[J]. J Clin Virol,2010,48(1):49-54. DOI:10. 1016/j.jcv.2010.02.002. |
| [13] Tsuguto F,Setsuko I,Miki E,et al. Hand,foot,and mouth disease caused by coxsackievirus A6,Japan,2011[J]. Emerg Infect Dis,2012,18(2):337-339. DOI:10. 3201/eid1802. 111147. |
| [14] Wei SH,Huang YP,Liu MC,et al. An outbreak of coxsackievirus A6 hand,foot,and mouth disease associated with onychomadesis in Taiwan,2010[J]. BMC Infect Dis,2011,11:346. DOI:10.1186/1471-2334-11-346. |
| [15] Gopalkrishna V,Patil PR,Patil GP,et al. Circulation of multiple enterovirus serotypes causing hand,foot and mouth disease in India[J]. J Med Microbial,2012,61(3):420-425. DOI:10.1099/jmm.0.036400-0. |
| [16] Lu QB,Zhang XA,Wo Y,et al. Circulation of Coxsackievirus A10 and A6 in hand-foot-mouth disease in China,2009-2011[J]. PLoS One,2012,7(12):e52073. DOI:10. 1371/journal.pone.0052073. |
| [17] Obereste MS,Maher K,Kilpatrick DR,et al. Molecular evolution of the human enteroviruses:correlation of serotype with VP1 sequence and application to picornavirus classification[J]. J Virol,1999,73(3):1941-1948. |
 2016, Vol. 37
2016, Vol. 37


