文章信息
- 杜小莉, 李伟, 阚飙, 崔志刚, 崔晶花. 2016.
- Du Xiaoli, Li Wei, Kan Biao, Cui Zhigang, Cui Jinghua. 2016.
- 我国部分地区克罗诺杆菌分离株分种及其方法的比较研究
- Study on identification of Cronobacter spp. species in 11 areas in China
- 中华流行病学杂志, 2016, 37(2): 259-262
- Chinese Journal of Epidemiology, 2016, 37(2): 259-262
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2016.02.022
-
文章历史
- 收稿日期: 2015-07-02
克罗诺杆菌(原阪崎肠杆菌)通过污染婴幼儿配方奶粉或代乳品引起婴幼儿脑膜炎、败血症、坏死性小肠结肠炎等[1, 2, 3]。虽然克罗诺杆菌感染的病例报道并不多,但由于致死率较高,并关系到婴幼儿食品风险问题,近些年受到越来越多关注[4, 5, 6, 7]。目前克罗诺杆菌属[Cronobacter(C.) spp.]可分为7个种,分别为C. sakazakii、C. malonaticus、C. dublinensis、C. turicensis、C. muytjensii、C. universalis和C. condimenti[8, 9],不同“种”之间的生物学特性有较大的区别,流行病学分布情况不同,致病性也可能存在一定差异[10],因此分“种”是克罗诺杆菌研究的基础,对进一步认识了解克罗诺杆菌具有重要意义。国外有报道应用生化反应,或者对16S rRNA、recN、rpoB、 fusA等基因序列分析进行分种研究[11, 12, 13],但对我国分离株采用哪种方法能更快速有效地分种还未见相关报道。为此本研究用不同方法对11个省份分离的105株克罗诺杆菌进行分种,并对各种方法进行比较研究。
材料与方法1. 菌株来源:105株菌主要分离自11个省份(北京、广西、河北、湖北、湖南、辽宁、四川、山东、上海、山西、天津)的食品(67株)、农村饮用水(33株)和健康人肛拭子标本(5株)。其中食品又包括婴幼儿米粉(31株)、婴幼儿配方奶粉(23株)、面条(11株)、饼干(1株)和蔬菜(1株)。标准菌株ATCC51329和ATCC29544由中国检验检疫科学院提供。所有菌株通过目标基因为dnaG的荧光定量PCR鉴定[14]。结果为阳性菌株在TSA琼脂平板上培养,并储存在-80 ℃的冰箱内。
2. 主要试剂和仪器:包括API 20E生化试剂(法国生物梅里埃公司)、基因组DNA提取试剂盒(QIA amp®公司)、Real-Time Premix Ex TaqTM(中国大连TaKaRa公司)、离心机(德国Enppendorf公司)、labcycler standard PCR 仪(德国Senso公司)、 7500Fast荧光定量PCR仪(美国ABI公司)、ABI3730基因序列分析仪(美国ABI公司)。
3. 实验方法:
(1)生化试验:除对所有菌株进行生化反应试验外,还附加了传统的单管生化试验,包括吲哚(Ind)卫矛醇(Dul)、α-甲基-D-吡喃葡萄糖苷(AMG)的产酸,以及丙二酸盐(Malo)合成试验。主要在基础培养基(蛋白胨10 g/L,酵母粉 1 g/L,氯化钠 5 g/L,酚红指示剂 0.018 g/L)上添加5% Dul、AMG。
(2)核酸提取:将培养在TSA琼脂平板上的单克隆菌接种于5 ml的胰蛋白胨培养液中过夜培养。取1 ml培养液8 000 r/min离心5 min后用QIA amp®核酸提取试剂盒提取,方法参照说明书。
(3)16S rRNA和fusA基因测序:扩增16S rRNA基因所使用的引物为616V(5′-AGAGTTT GATYMTGGCTC-3′)和630R(5′-CAKAAAGGA GGTGATC-3′),PCR反应条件:95 ℃ 2 min;94 ℃ 15 s,52 ℃ 30 s,72 ℃ 90 s,30 个循环;94 ℃ 15 s,52 ℃ 30 s,72 ℃ 129 s,15个循环;72 ℃ 7 min。根据Joseph等[9]的研究结果,fusA基因可用于克罗诺杆菌分种研究,所使用的扩增引物为fusA-F(5′-GAAACCGTATGGCGTCAG-3′)和fusA-R(5′-AGAACCGAAGTGCAGACG-3′),PCR反应条件:96 ℃ 1 min;96 ℃ 1 min,58 ℃ 1 min,72 ℃ 2 min,30个循环;72 ℃ 5 min。PCR反应中的聚合酶为TaKaRa公司生产的PreMix。PCR产物纯化后,用BigDye试剂配制测序反应体系,在ABI 3730XL测序仪上进行测序。16S rRNA基因使用的测序引物为16F(5′-AGAGTTTGATYMTGGCTC- 3′),16WR1(5′-CAKAAAGGAGGTGATCC-3′)。fusA基因测序引物为seqfusA-F(5′-GCTGGATGC GGTAATTGA-3′),seqfusA-R(5′-CCCATACCAG CGATGATG-3′)。
4. 数据分析:序列聚类分析采用极大似然比算法(maximum-likelihood algorithm),并利用Mega 5.0软件实现。统计评估时,引导值(bootstrap)使用迭代次数为1 000。参考菌株序列下载于NCBI网站。
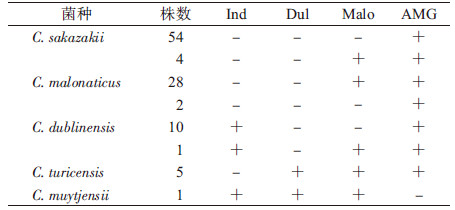
结 果1. 生化分种:用API 20E生化试剂条鉴定所有菌株,结果只能到克罗诺杆菌“属”水平,且鉴定率为51.1%~99.9%。根据Iversen等[8]2007年对各个种生化特点的分析,增加了Ind、Dul、AMG产酸,以及Malo合成试验,结果105株菌4个生化结果呈现6种情况(图 1)。
|
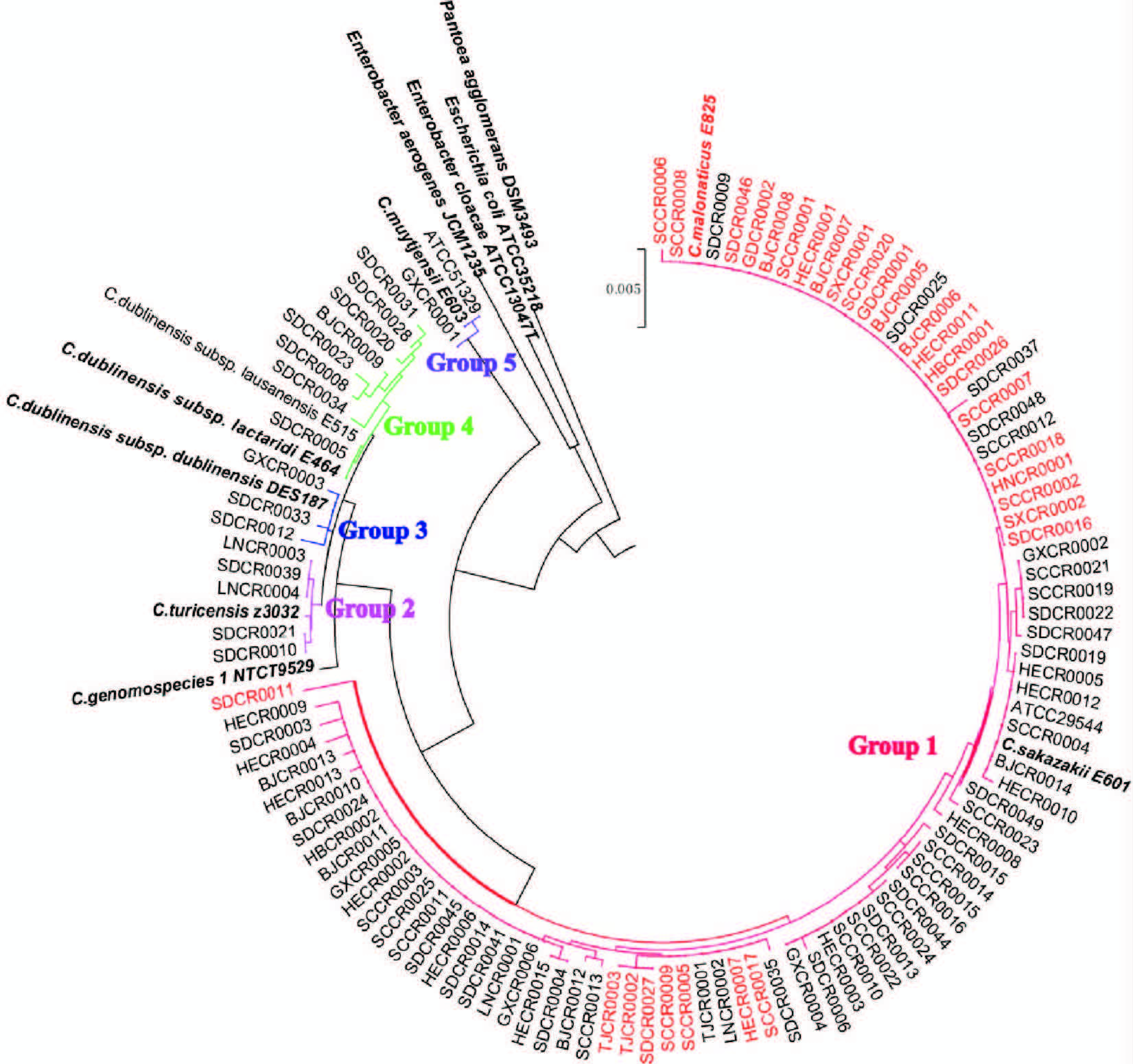
| 图 1 105株克罗诺杆菌16S rRNA基因序列聚类分析的分种结果 注:Group1是C. sakazakii(黑色)和C. malonaticus(红色)的集合;Group2为C. turicensis;Group3和Group4的菌株均属于C. dublinensis; Group5为C. muytjensii |
2. 16S rRNA分种: 将16S rRNA的测序结果拼接,再加上NCBI公布的各种参考菌株序列,通过Mega 5.0软件进行极大似然比算法聚类,可将105株菌分为5组(图 1),Group1是C. sakazakii和C. malonaticus两个种的集合,红色标识是通过生化反应鉴定为C. malonaticus的菌株,黑色标识的是通过生化反应鉴定为C. sakazakii的菌株,聚类结果显示无法将两个种分开。Group2中的4株菌与C. turicensis参考菌株聚在一起,Group3和Group4中的菌株均属于C. dublinensis,Group5仅有1株菌与C. muytjensii聚类在一起。所有菌株的相似度>98%。
3. 基因fusA分种:将基因fusA的测序结果拼接整理后,与数据库(http://www.pubmlst/cronobacter)中的序列比对,获得等位基因编码。本研究中有6株菌的fusA基因序列为新序列,获得的新编码为63~68。序列通过Mega 5.0软件进行极大似然比算法,结果显示所有菌株可分为5个组(图 2)。再根据Joseph等[13]的结论将105株菌分种为C. sakazakii(58株),C. malonaticus(30株),C. dublinensis(11株),C. turicensis(5株),C. muytjensii(1株)(图 1和表 1)。

|
| 图 2 105株克罗诺杆菌fusA基因序列聚类分析的分种结果 |
克罗诺杆菌通过生化反应分为16种生物型,由于随着菌株数目的增加,生物型别种类不断增加且复杂化,因此Iversen等[8, 15]通过分子生物学的方法对菌株进行重新分类,将原来的“种”升为“属”的水平,并对“属”内的菌株分种。目前商业化的生化检测试剂或生化仪器只能将克罗诺杆菌鉴定到“属”的水平,因此本研究首先考虑是否可以通过增加生化项目的方法对克罗诺杆菌属进行进一步分种。根据Iversen等[15]2007年的研究结果,研究中增加了Ind、Dul、AMG、Malo生化反应试验。虽然大致可将菌株分开,但由于C. turicensis和C. genomospecies 1(C. universalis)结果一致,无法将该两个“种”分开,故又对菌株进行16S rRNA测序分析,但亦无法将C. sakazakii和C. malonaticus 两个“种”有效分开。因此将两方法结合才能对克罗诺杆菌进行分种。
有报道认为可通过个别基因,例如recN、 rpoB、fusA等进行序列分析将菌株分种。虽然近些年recN和ropB两种基因较多用于细菌种属鉴定,但由于Joseph等[13]是基于7个等位基因的多位点序列分型(MLST)分析基础上建立的fusA基因分种方法,所以后者较前两种基因更具有科学性。本研究对所有菌株fusA基因序列聚类分析后的分种结果为58株C. sakazakii,30株C. malonaticus,11株C. dublinensis,5株C. turicensis,1株C. muytjensii。这与通过生化和16S rRNA序列分析两种方法结合的分种结果稍有不同,主要是在C. sakazakii和C. malonaticus两个种上有差异。Malo阳性菌株属于C. malonaticus,阴性菌株属于C. sakazakii,而fusA基因序列聚类分种将2株Malo阳性菌株划分到C. sakazakii,2株Malo阴性菌株划分到C. malonaticus中。针对该矛盾结果,2013年Joseph等[16]又比较了生化反应分种方法和序列聚类分析方法,发现生化反应方法存在一定缺陷,不适用于克罗诺杆菌分种。由于Iversen等在对克罗诺杆菌属进行分种时,也主要运用了f-AFLP、16S rRNA测序、DNA杂交等分子生物学方法,并由于生化反应具有不稳定性的特点,因此在本研究中最终以基因fusA序列聚类分析结果作为分种的依据。
综上所述,本研究通过生化反应、16S rRNA和fusA序列分析对克罗诺杆菌属进行分种方法的比较,表明目前采用fusA序列分析进行分种相对简捷有效,即如果获得的fusA等位基因编码是已知的,那么可以直接判断菌株“种”的类别。但该方法仍缺乏直观性,需将在序列分析的基础上研究其他方法,以达到快速检测目的,便于推广应用。
利益冲突 无| [1] Arseni A, Malamou-Ladas E, Koutsia C, et al.Outbreak of colonization of neonates with Enterobacter sakazakii[J].J Hosp Infect, 1987, 9(2):143-150.DOI:10.1016/0195-6701(87)90052-1. |
| [2] Biering G, Karlsson S, Clark NC, et al.Three cases of neonatal meningitis caused by Enterobacter sakazakii in powdered milk[J].J Clin Microbiol, 1989, 27(9):2054-2056. |
| [3] Gallagher PG, Ball WS.Cerebral infarctions due to CNS infection with Enterobacter sakazakii[J].Pediatr Radiol, 1991, 21(2):135-136.DOI:10.1007/BF02015629. |
| [4] Caubilla-Barron J, Hurrell E, Townsend S, et al.Genotypic and phenotypic analysis of Enterobacter sakazakii strains from an outbreak resulting in fatalities in a neonatal intensive care unit in France[J].J Clin Microbiol, 2007, 45(12):3979-3985.DOI:10.1128/JCM.01075-07. |
| [5] Lai KK.Enterobacter sakazakii infections among neonates, infants, children, and adults.Case reports and a review of the literature[J].Medicine, 2001, 80(2):113-122. |
| [6] Gosney MA, Martin MV, Wright AE, et al.Enterobacter sakazakii in the mouths of stroke patients and its association with aspiration pneumonia[J].Eur J Intern Med, 2006, 17(3):185-188.DOI:10.1016/j.ejim.2005.11.010. |
| [7] See KC, Than HA, Tang T.Enterobacter sakazakii bacteraemia with multiple splenic abscesses in a 75-year-old woman:a case report[J].Age Ageing, 2007, 36(5):595-596.DOI:10.1093/ageing/afm092. |
| [8] Iversen C, Mullane N, McCardell B, et al.Cronobacter gen.nov., a new genus to accommodate the biogroups of Enterobacter sakazakii, and proposal of Cronobacter sakazakii gen.nov., comb.nov., Cronobacter malonaticus sp.nov., Cronobacter turicensis sp.nov., Cronobacter muytjensii sp.nov., Cronobacter dublinensis sp.nov., Cronobacter genomospecies 1, and of three subspecies, Cronobacter dublinensis subsp.dublinensis subsp.nov., Cronobacter dublinensis subsp.lausannensis subsp.nov.and Cronobacter dublinensis subsp.lactaridi subsp.nov[J].Int J Syst Evol Microbiol, 2008, 58(6):1442-1447.DOI:10.1099/ijs.0.65577-0. |
| [9] Joseph S, Cetinkaya E, Drahovska H, et al.Cronobacter condimenti sp.nov., isolated from spiced meat, and Cronobacter universalis sp.nov., a species designation for Cronobacter spp.genomospecies 1, recovered from a leg infection, water and food ingredients[J].Int J Syst Evol Microbiol, 2012, 62(6):1277-1283.DOI:10.1099/ijs.0.032292-0. |
| [10] Grim CJ, Kothary MH, Gopinath G, et al.Identification and characterization of Cronobacter iron acquisition systems[J].Appl Environ Microbiol, 2012, 78(17):6035-6050.DOI:10.1128/AEM.01457-12. |
| [11] Kuhnert P, Korczak BM, Stephan R, et al.Phylogeny and prediction of genetic similarity of Cronobacter and related taxa by multilocus sequence analysis(MLSA)[J].Int J Food Micriobiol, 2009, 136(2):152-158.DOI:10.1016/j.ijfoodmicro.2009.02.022. |
| [12] Stoop B, Lehner A, Iversen C, et al.Development and evaluation of rpoB based PCR systems to differentiate the six proposed species within the genus Cronobacter[J].Int J Food Micriobiol, 2009, 136(2):165-168.DOI:10.1016/j.ijfoodmicro.2009.04.023. |
| [13] Joseph S, Sonbol H, Hariri S, et al.Diversity of the Cronobacter genus as revealed by multilocus sequence typing[J].J Clin Microbiol, 2012, 50(9):3031-3039.DOI:10.1128/JCM.00905-12. |
| [14] Seo KH, Brackett RE.Rapid, specific detection of Enterobacter sakazakii in infant formula using a real-time PCR assay[J].J Food Prot, 2005, 68(1):59-63. |
| [15] Iversen C, Lehner A, Mullane N, et al.The taxonomy of Enterobacter sakazakii:proposal of a new genus Cronobacter gen.nov.and descriptions of Cronobacter sakazakii comb.nov.Cronobacter sakazakii subsp.sakazakii, comb.nov., Cronobacter sakazakii subsp.malonaticus subsp.nov., Cronobacter turicensis sp.nov., Cronobacter muytjensii sp.nov., Cronobacter dublinensis sp.nov.and Cronobacter genomospecies 1[J].BMC Evol Biol, 2007, 7:64.DOI:10.1186/1471-2148-7-64. |
| [16] Joseph S, Hariri S, Forsythe SJ.Lack of continuity between Cronobacter biotypes and species as determined using multilocus sequence typing[J].Mol Cell Probes, 2013, 27(3-4):137-139.DOI:10.1016/j.mcp.2013.02.002. |
 2016, Vol. 37
2016, Vol. 37