文章信息
- 朱猛, 程阳, 戴俊程, 谢兰, 靳光付, 马红霞, 胡志斌, 师咏勇, 林东昕, 沈洪兵.
- Zhu Meng, Cheng Yang, Dai Juncheng, Xie Lan, Jin Guangfu, Ma Hongxia, Hu Zhibin, Shi Yongyong, Lin Dongxin, Shen Hongbing.
- 基于全基因组关联研究的中国人群肺癌风险预测模型
- Genome-wide association study based risk prediction model in predicting lung cancer risk in Chinese
- 中华流行病学杂志, 2015, 36(10): 1047-1052
- Chinese Journal of Epidemiology, 2015, 36(10): 1047-1052
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2015.10.002
-
文章历史
- 投稿日期: 2015-06-15
2. 清华大学医学院医学系统生物学研究中心;
3. 上海交通大学上海市精神卫生中心重点实验室;
4. 中国医学科学院北京协和医院肿瘤医院分子肿瘤学国家重点实验室
2 Medical Systems Biology Research Center, Tsinghua University School of Medicine;
3 Bio-X Institutes, Key Laboratory for the Genetics of Developmental and Neuropsychiatric Disorders, Ministry of Education, Shanghai Jiao Tong University;
4 State Key Laboratory of Molecular Oncology, Cancer Institute and Hospital, Chinese Academy of Medical Sciences, Peking Union Medical College
2012年中国肺癌新发病例数约为65.3万,标化发病率为36.1/10万,居我国男性恶性肿瘤发病率的首位、女性恶性肿瘤发病率的第二位;由肺癌导致的死亡病例数约为59.7万,占所有恶性肿瘤相关死亡的27.1%,在男性和女性中均居于首位[1]。
依据个人基因信息为癌症和其他疾病制定个体化医疗方案,从而采取个体化的预防策略,是在预防医学领域推动“精准医学”的重要发展方向。自2008年以来,世界范围内开展了多项肺癌相关的全基因组关联研究(GWAS),鉴别出20多个区域的40多个位点与肺癌易感性相关[2, 3, 4, 5, 6, 7, 8, 9, 10, 11, 12, 13]。利用GWAS提示的肺癌易感位点信息可能改善基于传统风险因素预测肺癌发病风险的能力。为了验证该假设,本研究基于中国汉族人群GWAS数据库,使用来源于南京与上海汉族人群的样本作为训练集,北京与武汉汉族人群的样本作为测试集,评价遗传信息对肺癌风险预测模型的改善能力,并构建中国汉族人群的肺癌风险预测模型,为中国汉族人群肺癌个体发病预测提供有效的评估工具。 对象与方法
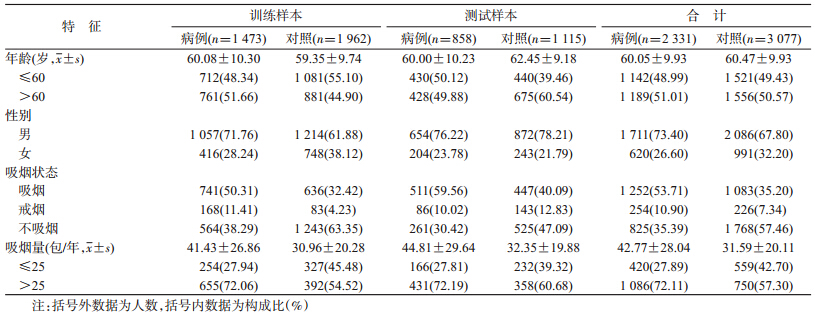
1. 研究对象:基于前期中国汉族人群GWAS数据库[13],选择2 331名肺癌病例和3 077名健康对照,其中1 473名病例与1 962名对照来源于南京和上海,858名病例与1 115名对照来源于北京和武汉。本研究使用南京与上海作为训练集,用于构建肺癌风险预测模型;使用北京与武汉作为测试集,用于验证肺癌风险预测模型的预测效能。
2. GWAS数据集准备:所有样本均采用美国Affymetrix公司的Affymetrix Genome-Wide Human SNP Array 6.0芯片进行基因分型。分型数据进行严格的质量控制以剔除不合格位点和样本。为了增加数据集覆盖度,对GWAS数据集进行基因型填补(imputation)。首先采用SHAPEIT2软件构建单倍型[14],然后采用IMPUTE2软件填补未分型位点的基因型[15],填补以千人基因组计划发布的单倍型数据为参照[16]。
3. 遗传位点选择:系统收集整理GWAS Catalog收录的所有P<5×10-8的肺癌易感位点[17],剔除在中国人群中最小等位基因频率(MAF)<0.05的位点,对于存在连锁不平衡的位点(r2>0.5),保留P值较小的位点。对于满足上述条件的位点,在训练集样本中采用逐步回归(后退法)筛选,至最终模型中所有位点均有统计学意义(P<0.05)。
4. 统计学建模:采用多元logistic回归构建肺癌风险预测模型,根据logistic回归公式,推导出个体发病概率公式:
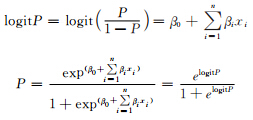
建模流程见图 1。首先在训练集中基于吸烟量构建肺癌传统因素风险预测模型(吸烟模型);然后基于遗传位点使用加权法(wGRS)估算个体遗传得分,构建肺癌遗传风险预测模型(遗传效应模型);最后同时结合吸烟和遗传位点信息构建多因素肺癌风险预测模型(联合模型)。加权法是指在对危险因素合并时,考虑因素本身的单独效应,以logistic回归计算得到的变量OR值进行加权得到每个变量的遗传得分,将得分相加后纳入模型,具体公式:
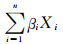
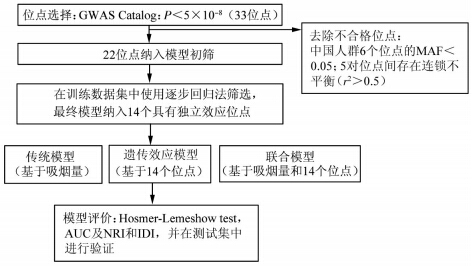 |
| 图 1 肺癌发病风险预测模型建立流程 |
根据受试者工作特征(ROC)曲线下面积(AUC)、净分类指数(NRI)和整体鉴别指数(IDI)评 价上述3种模型的预测效能,并进一步在测试集中进行验证。所有分析利用R3.1.3软件完成。 结果
1. 样本基线特征:研究样本的基线特征见表 1。在训练样本与测试样本中,吸烟状态与吸烟量差异均有统计学意义(P<0.001)。
2. 单核苷酸多态性(SNP)位点选择:截至2015年4月20日,GWAS Catalog共收录21项肺癌全基因组易感性关联研究,共包含41个独立SNP位点,其中33位点P<5×10-8,在中国人群中有6个位点MAF<0.05,5对位点间存在高度连锁不平衡(r2>0.5),最终有22个肺癌相关位点进入模型初筛。在训练样本中,经逐步回归分析后,最终14个位点进入模型(表 2)。在模型纳入的14个位点中,除了rs938682(15q25.1,CHRNA3-CHRNA5)发现于欧美人群之外,其余13个位点均来源于亚洲人群。
3. 不同模型预测肺癌发生风险:吸烟模型显示,在训练集中,与不吸烟者相比,轻度吸烟者患肺癌风险增加1.21倍(调整OR=2.21,95%CI:1.76~2.78),重度吸烟者患肺癌的风险增加3.76倍(调整OR=4.76,95%CI:3.88~5.84)。遗传效应模型显示,根据14个位点计算个体的遗传得分,并根据对照的得分将样本分为4组,随着得分增加,个体患肺癌风险显著增加(P=2.04×10-23)。联合模型显示,根据对照危险得分将样本分为4组,组间风险增加趋势变得更加明显(P=1.13×10-54),得分最高组患肺癌的风险是得分最低组的5.73倍(95%CI:4.52~7.26)。这一变化趋势在测试集中得到了验证(表 3)。
4. 模型预测能力与效度检验:联合模型、吸烟模型和遗传效应模型AUC分别为0.69、0.65和0.60,见表 4。与吸烟模型和遗传效应模型相比,联合模型的预测效能较高(P<0.001),见图 2。Hosmer-Lemeshow检验表明3种模型的拟合度均较好(P>0.05)。这种趋势在测试集中得到了验证(图 2),吸烟模型和遗传效应模型的AUC均为0.61(95%CI:0.58~0.63),联合模型的AUC增加为0.65(95%CI:0.62~0.67),预测效能提高(P<0.001)。但是基于训练样本建立的联合模型和吸烟模型,在测试样本中拟合度较差(P<0.05)。进一步对年龄、性别进行分层分析,发现不同层间该趋势保持一致(图 3)。在中国大部分女性为不吸烟者的基本国情下,吸烟模型预测效能差,AUC为0.54(95%CI:0.53~0.56),而联合模型的预测效能为0.64(95%CI:0.61~0.67)。
 |
| 图 2 基于GWAS结果构建的中国人群肺癌风险预测模型预测能力 |
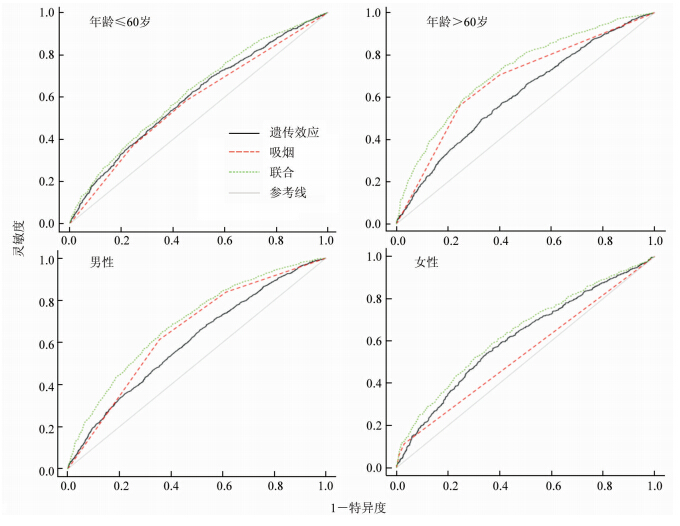 |
| 图 3 中国人群肺癌风险预测模型分层分析的ROC曲线 |
5. 阳性界值与重分类:根据联合模型,使用Youden法在训练样本中确定的最佳阳性界值为0.4,此时预测的灵敏度和特异度分别为64.70%和64.93%。以0.4为阳性界值,进一步评价吸烟模型和联合模型对病例和对照重分类的影响(表 5),结果显示,在训练集中联合模型NRI增加4.57%(95%CI:2.23%~6.91%),IDI增加3.11%(95%CI:2.52%~3.69%),差异均有统计学意义(P<0.001)。在测试样本中,NRI增加2.77%,但差异无统计学意义(P=0.069),而IDI增加了3.16%,差异有统计学意义(P<0.001)。
肺癌发病率和死亡率均位于我国恶性肿瘤的首位,且起病隐匿、恶性程度高、病程进展快、易复发和转移、预后较差。因此,通过对肺癌高危人群进行筛查,达到早诊早治是降低肺癌发病率和死亡率的理想手段。近年来,研究者们通过GWAS这一最新的研究手段,发现了一系列与肺癌易感性密切相关的遗传位点。本研究主要探讨了如何有效的利用这些研究成果,改善肺癌传统的风险预测模型,进一步鉴别肺癌高危人群,以减少疾病筛查的经济和健康负担。
目前,国内外已发表多种肺癌发病风险评估模型,如Bach模型、LLP 模型和Etzel模型等[19, 20, 21]。纳入的危险因素分别包括吸烟、戒烟、石棉接触史、痰细胞学检查及呼吸性疾病史等。Bach等[19]通过使用年龄、吸烟(吸烟量、吸烟年数和戒烟年数)和石棉暴露等危险因素在18 172名吸烟或戒烟人群中构建肺癌风险预测模型,AUC可达到0.7~0.9,然而该模型缺乏外部数据的验证,且目前石棉已经淘汰出日常生活,该模型已经失去其原有的使用价值。而LLP和Etzel模型[20, 21]主要使用年龄、吸烟、肿瘤家族史和职业暴露等因素,AUC仅在0.55~0.70间。 Hoggart等[22]使用初始吸烟年龄、吸烟量和10个职业环境暴露因素,根据初始吸烟年龄分层构建风险预测模型,预测 效能较好,AUC为0.84(0.81~0.88),该研究尝试在模型构建时引入GWAS发现的2个遗传位点(5p15、15q25),然而仅有小部分样本拥有分型信息,引入遗传信息并没有显著改善模型的预测效能。对于上述模型,多只适用于吸烟或戒烟者,并不适用于不吸烟者,且上述模型主要基于欧美人群构建,能否直接应用于中国汉族人群,尚有待进一步验证。
传统模型主要基于流行病调查数据(如年龄、吸烟状态和职业暴露等),在信息收集的过程中难免存在信息偏倚,与此相比,基于遗传易感位点的遗传得分可以稳定存在,检测方法稳定且可重复,是用于风险预测的可靠资源。本研究基于前期中国汉族人群GWAS数据库,以南方样本作为训练集用于构建肺癌风险预测模型、北方样本作为测试集验证肺癌风险预测模型的预测效能,系统评价了遗传信息对于传统肺癌风险预测模型的改善能力。经过系统筛选,最终选择14个既往GWAS报道的SNPs用于构建遗传得分,联合模型在训练集和测试集中均可以显著改善单纯基于吸烟信息的模型。在分层分析中,不同组间这种趋势均得以验证,提示模型稳定可靠。
| [1] Bray F,Ren JS,Masuyer E,et al. Global estimates of cancer prevalence for 27 sites in the adult population in 2008[J]. Int J Cancer,2013,132(5):1133-1145. |
| [2] Dong J,Hu ZB,Wu C,et al. Association analyses identify multiple new lung cancer susceptibility loci and their interactions with smoking in the Chinese population[J]. Nat Genet,2012,44(8): 895-899. |
| [3] Lan Q,Hsiung CA,Matsuo K,et al. Genome-wide association analysis identifies new lung cancer susceptibility loci in never-smoking women in Asia[J]. Nat Genet,2012,44(12): 1330-1335. |
| [4] Dong J,Jin GF,Wu C,et al. Genome-wide association study identifies a novel susceptibility locus at 12q23.1 for lung squamous cell carcinoma in han chinese[J]. PLoS Genet,2013,9 (1):e1003190. |
| [5] Hu ZB,Xia YK,Guo XJ,et al. A genome-wide association study in Chinese men identifies three risk loci for non-obstructive azoospermia[J]. Nat Genet,2012,44(2):183-186. |
| [6] Broderick P,Wang YF,Vijayakrishnan J,et al. Deciphering the impact of common genetic variation on lung cancer risk:a genome-wide association study[J]. Cancer Res,2009,69(16): 6633-6641. |
| [7] Shiraishi K,Kunitoh H,Daigo Y,et al. A genome-wide association study identifies two new susceptibility loci for lung adenocarcinoma in the Japanese population[J]. Nat Genet,2012, 44(8):900-903. |
| [8] Yoon KA,Park JH,Han J,et al. A genome-wide association study reveals susceptibility variants for non-small cell lung cancer in the Korean population[J]. Hum Mol Genet,2010,19(24): 4948-4954. |
| [9] Miki D,Kubo M,Takahashi A,et al. Variation in TP63 is associated with lung adenocarcinoma susceptibility in Japanese and Korean populations[J]. Nat Genet,2010,42(10):893-896. |
| [10] Wang YP,Lu Y,Zhang Y,et al. The draft genome of the grass carp (Ctenopharyngodon idellus) provides insights into its evolution and vegetarian adaptation[J]. Nat Genet,2015,47(6): 625-631. |
| [11] Landi MT,Chatterjee N,Yu K,et al. A genome-wide association study of lung cancer identifies a region of chromosome 5p15 associated with risk for adenocarcinoma[J]. Am J Hum Genet, 2009,85(5):679-691. |
| [12] Deng QF,Guo H,Dai JC,et al. Imputation-based association analyses identify new lung cancer susceptibility variants in CDK6 and SH3RF1 and their interactions with smoking in Chinese populations[J]. Carcinogenesis, 2013, 34 (9) : 2010-2016. |
| [13] Hu ZB,Wu C,Shi YY,et al. A genome-wide association study identifies two new lung cancer susceptibility loci at 13q12.12 and 22q12.2 in Han Chinese[J]. Nat Genet,2011,43(8):792-796. |
| [14] Delaneau O,Marchini J. Integrating sequence and array data to create an improved 1000 Genomes Project haplotype reference panel[J]. Nat Commun,2014,5:3934. |
| [15] Howie BN,Donnelly P,Marchini J. A flexible and accurate genotype imputation method for the next generation of genome-wide association studies[J]. PLoS Genet,2009,5(6):e1000529. |
| [16] 1 000 Genomes Project Consortium,Abecasis GR,Auton A,et al. An integrated map of genetic variation from 1 092 human genomes[J]. Nature,2012,491(7422):56-65. |
| [17] Welter D,MacArthur J,Morales J,et al. The NHGRI GWAS Catalog,a curated resource of SNP-trait associations[J]. Nucleic Acids Res,2014,42(Database issue):D1001-1006. |
| [18] Dai J,Hu Z,Jiang Y,et al. Breast cancer risk assessment with five independent genetic variants and two risk factors in Chinese women[J]. Breast Cancer Res,2012,14(1):R17. |
| [19] Bach PB,Kattan MW,Thornquist MD,et al. Variations in lung cancer risk among smokers[J]. J Natl Cancer Inst,2003,95(6): 470-478. |
| [20] Cassidy A,Myles JP,van Tongeren M,et al. The LLP risk model: an individual risk prediction model for lung cancer[J]. Br J Cancer,2008,98(2):270-276. |
| [21] Spitz MR,Hong WK,Amos CI,et al. A risk model for prediction of lung cancer[J]. J Natl Cancer Inst,2007,99(9):715-726. |
| [22] Hoggart C,Brennan P,Tjonneland A,et al. A risk model for lung cancer incidence[J]. Cancer Prev Res (Phila),2012,5(6): 834-846. |
 2015, Vol. 36
2015, Vol. 36