文章信息
- 薛泽润, 王颖芳, 段广才, 杨海燕, 郗园林, 王鹏飞, 王琳琳, 郭向娇. 2015.
- Xue Zerun, Wang Yingfang, Duan Guangcai, Yang Haiyan, Xi Yuanlin, Wang Pengfei, Wang Linlin, Guo Xiangjiao. 2015.
- 志贺菌中成簇规律间隔短回文重复序列的分子分布特征
- Molecular characteristics of Clustered Regularly Interspaced Short Palindromic Repeat in Shigella
- 中华流行病学杂志, 2015, 36(8): 875-878
- Chinese Journal of Epidemiology, 2015, 36(8): 875-878
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2015.08.023
-
文章历史
- 投稿日期:2015-01-04
2. 河南科技大学;
3. 郑州大学公共卫生学院;
4. 新乡医学院分子诊断与医学检验技术河南省协同创新中心
2. Henan University of Science and Technology;
3. School of Public Health, Zhengzhou Universtiy;
4. Henan Innovation Center of Molecular Diagnosis and Laboratory Medicine, Xinxiang Medical University
近年来在细菌中发现的成簇规律间隔短回文重复序列(CRISPR)能抵御外源遗传物质入侵[1]。CRISPR系统包括重复序列、间隔序列和前导序列,间隔序列是核心,呈现多态性,能反映曾经入侵细菌的外源遗传物质,在不同菌株间分布不同[2]。CRISPR有免疫记忆和防御功能,当外源基因第一次入侵细菌时,CRISPR识别和整合前间隔序列(protospacer),形成新的间隔序列,当外源基因再次侵袭时,CRISPR识别外源基因、转录间隔序列和部分重复序列,crRNA与trans-activating crRNA(tracrRNA)、Cas 蛋白结合形成复合体,破坏入侵的外源基因[3],细菌CRISPR可识别与间隔序列相同的外源遗传物质,并启动免疫防御。志贺菌能导致细菌性痢疾(菌痢)[4]。菌痢是全球都存在的公共卫生问题,尤其对发展中国家造成重大危害,在我国属于乙类传染病,发病率居我国甲乙类传染病第3位,是传染病控制的重点[5]。按血清分型志贺菌分为4群:福氏、宋内、痢疾和鲍氏。发达国家流行的主要是宋内志贺菌,而我国以福氏志贺菌为主[6]。我国CRISPR相关研究较少[7],探索志贺菌中CRISPR的分子流行病学特征具有重要意义。 材料与方法
1. 菌株:52株志贺菌均为河南省分子医学重点实验室收集保存,1996年江西分离株6株,均为福氏志贺菌。河南分离株41株(福氏志贺菌28株、宋内志贺菌11株、痢疾志贺菌2株),其中1998年2株,1999年1株,2000年6株,2001年7株,2003年2株,2004年3株,2005年3株,2006年5株,2007年2株,2008年10株。2013年北京分离株5株,均为福氏志贺菌。
2. 试剂:诊断血清购自卫生部兰州生物制品研究所。胰蛋白胨、酵母浸粉(英国Oxoid公司)。细菌基因组DNA提取试剂盒、PCR相关试剂和100 bp DNA Ladder购于上海莱枫生物科技有限公司。
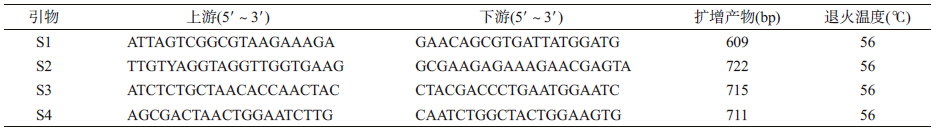
3. 引物:从GenBank和CRISPR Database数据库中查询志贺菌CRISPR基因相关序列,S1、S2、S3、S4为CRISPR Database数据库中已经发现的志贺菌CRISPR位点,选择保守序列,利用Primer 5.0设计引物,引物合成由生工生物工程(上海)股份有限公司完成(表 1)。
4. PCR反应:以细菌基因组DNA为模板进行PCR扩增,扩增体系:三蒸水19 μl,2×Taq PCR Master Mix 25 μl,上下游引物各1 μl,模板DNA 4 μl,共50 μl。反应条件为94 ℃ 5 min;94 ℃ 60 s,56 ℃ 45 s,72 ℃ 60 s,32 个循环;72 ℃ 10 min。取5 μl PCR 产物进行2%琼脂糖凝胶电泳,用凝胶成像仪观察结果并照相。
5. 核酸序列分析:PCR产物由生工生物工程(上海)股份有限公司进行测序,用CRISPR Finder对志贺菌的CRISPR进行比对。
6. 统计学分析:用SPSS 17.0软件进行统计学检验,P<0.05为差异有统计学意义。 结 果
1. 志贺菌CRISPR位点分布:志贺菌4个CRISPR位点的阳性率分别为33. 69%(S1)、50.00%(S2)、82.69%(S3)和73.08%(S4);每个CRISPR位点有1~3个间隔序列,S1和S2仅在个别菌株有重复序列退化,在2株志贺菌发现了新的S3重复序列,S4的间隔序列呈现多态性,有4种不同模式(表 2)。
2. 志贺菌中CRISPR的重复序列和间隔序列:志贺菌CRISPR按照重复序列和间隔序列分成11组,其中S1包括2种亚型,S2包括3种亚型,S3包括2种亚型,S4包括4种亚型(表 3)。
3. 志贺菌CRISPR与菌株分离时间的关系:河南分离株以分离时间M(2004年)作为界点分组,2004年及以前分离株21株,为A组;2004年以后分离株20株,为B组。A组包括14种CRISPR亚型,共获得95个间隔序列;B组包括7种CRISPR亚型,共获得68个间隔序列。6株江西分离株的CRISPR亚型:0110型2株,0111型1株,0011型1株,1111型1株,0002型1株。5株北京分离株的CRISPR亚型:0110型2株,0010型1株,0112型1株,0114型1株。 2004年前后分离的河南志贺菌S1分布差异有统计学意义(P<0.05),2004年后分离的菌株中均未检出该位点;S2、S3 和S4在两组中的分布差异无统计学意义(表 4)。
CRISPR偕同Cas蛋白发挥免疫抑制作用,已有研究显示,志贺菌中广泛存在Cas1和Cas2蛋白[8],提示志贺菌中可能存在CRISPR系统。间隔序列与噬菌体、质粒等外源基因具有同源性[1],记录了外源基因入侵细菌的过程,能反映细菌的进化史。本研究在志贺菌中发现了不同的CRISPR亚型,提示曾入侵的外源基因不同,发现志贺菌CRISPR各位点的间隔序列较少,以1~2个为主。相对于大肠埃希菌,志贺菌含有的间隔序列较少,可能与志贺菌的生存环境有关,志贺菌多寄生于人的结肠和直肠黏膜,环境中噬菌体较少[9]。4个CRISPR位点在志贺菌中均被检出,但是不同位点的CRISPR阳性率不同,提示志贺菌不同的CRISPR位点其在获取间隔序列的能力上有差异,其在免疫防御的作用和地位也不同,深入研究间隔序列有助于更深刻地理解细菌的进化机制。
河南分离的福氏志贺菌S1在时间分布上有差异,2004年以后分离的菌株中均未检出该位点,可能是与S1的间隔序列同源的外源基因对志贺菌的危险降低,细菌通过删除一些相对古老和无用的间隔序列,保证细菌基因组长度的稳定;此外,志贺菌多寄生于人的结肠和直肠黏膜,其生存环境中噬菌体较少,而抗生素对其的威胁更大,相关研究表明细菌的耐药程度和CRISPR位点呈负相关,同时CRISPR能限制耐药基因在细菌间的传播[10]。随着噬菌体的危害降低和抗生素威胁的增加,志贺菌通过丢失CRISPR位点以降低其对外源基因的抑制,从而有利于耐药基因通过水平转移在细菌间扩散。而S2、S3和S4没有差异,说明志贺菌中CRISPR各位点的稳定程度不同,有些CRISPR位点的间隔序列相对稳定,而有些进化较快。相对于分离时间较早的志贺菌,分离时间较晚菌株含有的间隔序列更少,提示志贺菌随着时间的推移会丢失一些间隔序列,与其生活的环境密切相关。由于本研究的菌株较少,有一定的局限性。
| [1] Grissa I, Vergnaud G, Pourcel C. The CRISPRdb database and tools to display CRISPRs and to generate dictionaries of spacers and repeats[J]. BMC Bioinform, 2007, 8(1):172. |
| [2] Kunin V, Sorek R, Hugenholtz P. Evolutionary conservation of sequence and secondary structures in CRISPR repeats[J]. Genome Biol, 2007, 8(4):R61. |
| [3] Brouns SJJ, Jore MM, Lundgren M, et al. Small CRISPR RNAs guide antiviral defense in prokaryotes[J]. Science, 2008, 321(5891):960-964. |
| [4] Pupo GM, Lan R, Reeves PR. Multiple independent origins of Shigella clones of Escherichia coli and convergent evolution of many of their characteristics[J]. Proc Natl Acad Sci USA, 2000, 97(19):10567-10572. |
| [5] Zhong HJ, Chang ZR, Zhang J. Analysis on bacillary dysentery surveillance data collected from the National Surveillance System in 2007[J]. Chin J Epidemiol, 2010, 31(3):304-307. (in Chinese) 钟豪杰, 常昭瑞, 张静. 中国2007年细菌性痢疾监测分析[J]. 中华流行病学杂志, 2010, 31(3):304-307. |
| [6] Mu YJ, Zhang BF, Zhao JY, et al. Analysis of distribution and drug resistance of Shigella in Henan province during 2010-2013[J]. Chin J Health Lab Tec, 2014, 24(24):3616-3618. (in Chinese) 穆玉姣, 张白帆, 赵嘉咏, 等. 河南省2010-2013年志贺菌群分布及其耐药分析[J]. 中国卫生检验杂志, 2014, 24(24):3616-3618. |
| [7] Bian FN, Li WG, Li XP, et al. Application of GoPubMed in bibliometric analysis of literature on CRISPR[J]. Chin J Epidemiol, 2014, 35(12):1400-1403. (in Chinese) 边富宁, 李文革, 李先平, 等. 利用GoPubMed对CRISPR相关文献的计量学分析[J]. 中华流行病学杂志, 2014, 35(12):1400-1403. |
| [8] Xue ZR, Wang YF, Duan GC, et al. Clustered regularly interspaced short palindromic repeat associated protein genes cas1 and cas2 in Shigella[J]. Chin J Epidemiol, 2014, 35(5):581-584. (in Chinese) 薛泽润, 王颖芳, 段广才, 等. 志贺菌成簇规律间隔短回文重复序列相关蛋白基因cas1和cas2研究[J]. 中华流行病学杂志, 2014, 35(5):581-584. |
| [9] Díez-Villaseñor C, Almendros C, García-Martínez J, et al. Diversity of CRISPR loci in Escherichia coli[J]. Microbiology, 2010, 156(Pt 5):1351-1361. |
| [10] Xue ZR, Wang YF, Duan GC. Progress on the studies of association between clustered regularly interspaced short palindromic repeat and antibiotic resistance[J]. Chin J Epidemiol, 2015, 36(3):293-296. (in Chinese) 薛泽润, 王颖芳, 段广才. 成簇规律间隔短回文重复序列及其与细菌耐药的研究进展[J]. 中华流行病学杂志, 2015, 36(3):293-296. |
 2015, Vol. 36
2015, Vol. 36