文章信息
- 宋伟, 朱贝贝, 田瑶, 钟荣, 田晶, 缪小平, 王丽. 2014.
- Song Wei, Zhu Beibei, Tian Yao, Zhong Rong, Tian Jing, Miao Xiaoping, Wang Li. 2014.
- U2依赖型mRNA剪接体与肝癌的相关性研究
- Research on the association between U2-dependent spliceosome gene and hepatocellular cancer
- 中华流行病学杂志,2015, 36(6): 634-638
- Chinese Journal of Epidemiology,2015, 36(6): 634-638
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2015.06.021
-
文章历史
- 投稿日期:2014-12-15
2. 华中科技大学同济医学院公共卫生学院流行病和卫生统计学系
2. Department of Epidemiology and Biostatistics, School of Public Health, Tongji Medical College, Huazhong University of Science and Technology
原发性肝癌病死率高,目前居于全球癌症死因第三位[1],其发生是遗传和环境因素共同作用的结果[2]。 mRNA选择性剪接影响着人类90%以上的基因表达,一旦发生异常,则可能通过影响包括细胞增殖、发育与分化等在内的多个基因的正常剪接过程,在肿瘤发生中扮演着重要角色[3]。2011年以来,Nature、Cell等杂志先后报道剪接体基因,尤其是U2核糖核蛋白体依赖型剪接体相关基因(U2AF35、U2AF65、SF1、SRSF1、SF3A1、SF3B1和PRPF40B)的突变与血液肿瘤如慢性髓细胞性白血病以及实体瘤如胰腺癌、乳腺癌以及黑色素瘤等发生有关[4]。本课题组前期研究也发现U2AF65基因多态性与胰腺癌发生有关[5]。本研究采用两阶段病例对照研究方法,探讨U2核糖核蛋白体依赖型剪接体相关基因(U2AF1、U2AF2、SRSF1、SRSF2、SF3B1、SF3A1、SF1和PRPF40B)与肝癌易感性的关系。
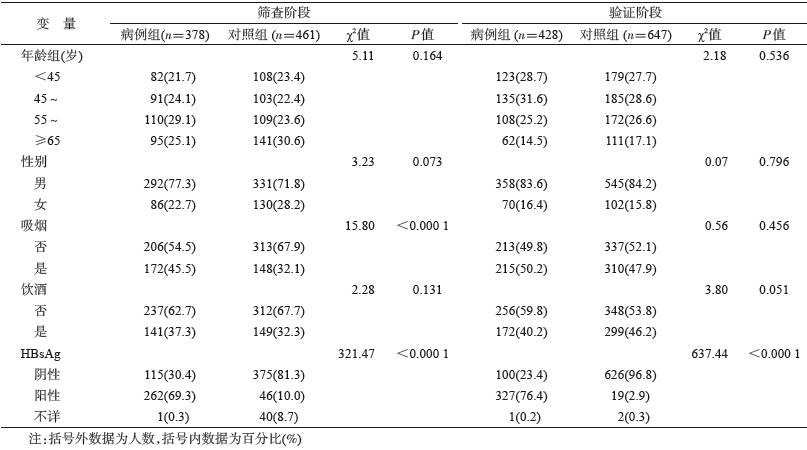
对象与方法1.研究对象:采用两阶段病例对照研究,筛查阶段共纳入378例肝癌患者和461例对照,病例为2009年5月至2012年6月湖北省武汉市同济医院经病理组织确诊的新发肝癌患者。对照来自同期武汉市同济医院参加体检的非肿瘤个体。验证阶段共纳入468例肝癌患者和707例对照,病例为2007年9月至2009年9月北京市佑安医院经病理组织确诊的新发肝癌患者。对照来源于同期在北京市佑安医院参加体检的非肿瘤个体。上述两阶段研究人群病例和对照均按照年龄(±5岁)和性别进行匹配。问卷收集性别、年龄、吸烟和饮酒状况等资料。将每天至少吸烟1支,连续或累计吸烟≥6个月定义为吸烟;将每天至少饮酒1次,连续饮酒≥3个月定义为饮酒。HBsAg携带状况从医院电子病历系统中获得。所有病例和对照均为汉族且无亲缘关系,在资料收集和血样采集前获得所有研究对象签署的知情同意书。
2.候选SNP挑选:在HapMap中下载中国北京汉族人群(Han Chinese in Beijing,CHB)关于剪接体关键基因U2AF1、U2AF2、SF1、SRSF1、SRSF2、SF3A1、SF3B1和PRPF40B的数据(HapMap Data Release 27 phase Ⅰ+Ⅱ+Ⅲ,http://www.Hapmap.org)后采用HaploView 4.2 软件,以参数R2>0.8、MAF>0.05的标准在整个基因上(包括基因上下游各2 kb)筛选能代表这个基因的标签SNP(tagSNPs)82个,然后采用功能预测软件SNP info挑选具有潜在功能的21个SNP。
3.基因分型:筛查阶段采用TaqMan Openarray 基因分型系统(Applied Biosystem,Foster City,CA,USA)对上述SNP进行基因分型;对阳性位点在验证阶段人群中使用美国Applied Biosystems公司的TaqMan RT-PCR进行分型验证。抽取5%样本验证分型的准确性,结果显示重复样本分型一致率为100%。
4.统计学分析:采用χ2检验比较病例组和对照组间性别、年龄分组、吸烟、饮酒和HBsAg携带状况的差异,对两阶段人群对照组中各位点基因型频率分布进行拟合优度χ2检验判断分型结果是否符合Hardy-Weinberg遗传平衡。按照显性模型并采用非条件logistic回归模型校正吸烟、饮酒和HBsAg因素后分析SNP与肝癌发生的关联。采用错误发现率(false discovery rate,FDR)对所有研究结果进行校正。利用SAS 9.3软件完成各种统计学分析,所有检验均为双侧,以P<0.05为检验水准;运用R语言V3.1.2评价加法交互作用,具体指标包括交互作用的超额危险度(RERI)、归因交互百分比(AP)和协同指数(SI)[6]。
结果1. 一般人口学特征:两阶段共纳入肝癌患者806例,对照1 108例(表 1)。筛查阶段共纳入378例肝癌患者和461例对照,病例组平均年龄(55.7±12.5)岁,男女性别比为3.4 ∶ 1;对照组平均年龄(55.7±14.0)岁,男女性别比为2.5 ∶ 1。验证阶段共纳入428例肝癌患者和647例对照,病例组平均年龄(51.7±12.0)岁,男女性别比为5.1 ∶ 1;对照组平均年龄(51.3±14.6)岁,男女性别比为5.3 ∶ 1。两阶段人群中病例组和对照组年龄、性别分布及饮酒史差异均无统计学意义(P>0.05);筛查阶段病例组吸烟者所占比例(45.5%)高于对照组(32.1%),差异有统计学意义(P<0.000 1),而验证阶段两者差异无统计学意义。筛查、验证两阶段均观察到病例组HBsAg阳性者所占比例(分别为69.3%和76.4%)高于对照组(分别为10.0%和2.9%),差异有统计学意义(P<0.000 1)。
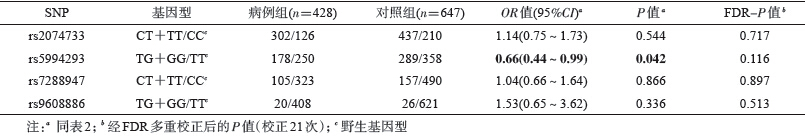
2.筛查阶段SNP位点与肝癌发病风险的关联分析:对照组21候选个位点中17个位点(表 2)符合Hardy-Weinberg遗传平衡,纳入下一步分析。仅观察到SF3A1的4个位点(rs2074733、rs5994293、rs7288947和rs9608886)的基因型分布在病例组和对照组中差异有统计学意义。校正性别、年龄、吸烟、饮酒和HBsAg因素后,携带SF3A1 rs2074733 T等位基因的个体较携带野生纯合型CC基因型的个体发生肝癌风险降低64%(OR=0.36,95%CI:0.25~0.53,FDR-P<0.000 1)。携带SF3A1 rs5994293 G等位基因者较携带野生纯合型TT基因型者发生肝癌风险降低58% (OR=0.42,95%CI:0.29~0.61,FDR-P<0.000 1)。携带SF3A1 rs7288947 T等位基因者较CC基因型者发生肝癌风险降低46%(OR=0.54,95%CI:0.41~0.72,FDR-P=0.001)。携带SF3A1 rs9608886 G等位基因者较TT基因型者发病风险降低83%(OR=0.17,95%CI:0.10~0.31,FDR-P<0.000 1)。
3.验证阶段单位点与肝癌发病风险的关联分析:4个位点rs2074733、rs5994293、rs7288947和rs9608886在验证阶段人群中进行验证。其在对照组中的基因型频率分布均满足Hardy-Weinberg遗传平衡。校正性别、年龄、吸烟、饮酒和HBsAg因素后,携带rs5994293 G等位基因者较TT基因型者发生肝癌风险降低34%(OR=0.66,95%CI:0.44~0.99,P=0.042),与筛查阶段的结果一致。但经FDR校正后差异无统计学意义(FDR-P=0.116)。其余3个位点均未发现与肝癌发病风险存在关联(表 3)。
4.合并两阶段人群SF3A1 rs5994293与肝癌发病风险的关联分析:将两阶段人群合并分析,校正性别、年龄、吸烟、饮酒和HBsAg因素后,携带rs5994293 G等位基因者较TT基因型者发生肝癌风险降低30%(OR=0.70,95%CI:0.58~0.84,FDR-P=0.000 5)。
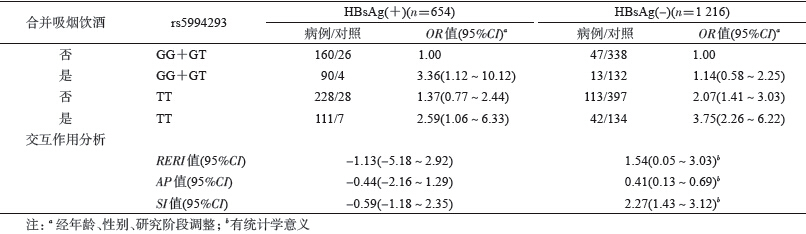
5.合并阶段按照HBsAg因素分层后SF3A1 rs5994293与合并吸烟饮酒的交互作用:合并阶段按照HBsAg因素分层后研究结果显示,HBsAg阴性者中合并吸烟饮酒与SF3A1 rs5994293 TT基因型之间存在协同效应,加法交互协同指数SI为2.27(95%CI:1.43~3.12)。但未观察到HBsAg阳性者中合并吸烟饮酒与上述基因型之间存在加法交互作用(表 4)。
本研究发现U2依赖型mRNA剪接复合体相关基因中SF3A1 rs5994293可能与肝癌发病相关,携带G等位基因的个体较携带TT基因型者发病风险降低30%,且SF3A1的基因多态性及其与吸烟的协同作用可能与肝癌发生风险相关。
SF3A1是剪接体催化核心U2-snRNP的重要组分,后者能识别3′端剪接位上游的分支位点,定位mRNA上被剪切除去的内含子,从而决定内含子的切除位置正确与否[7]。SF3A1主要功能是与U2- snRNP结合,共同参与3′剪接位点的识别,其发生突变将会影响U2-snRNP与分支点结合,从而最终抑制剪接过程[7]。目前慢性淋巴细胞性白血病和骨髓增生异常综合征以及实体瘤包括肺癌、乳腺癌以及胰腺癌患者中均发现SF3A1基因突变[8, 9]。SF3A1的表达研究显示人类非小细胞、小细胞肺癌细胞中SF3A1蛋白表达量较人正常气管上皮细胞高4.7~11.0倍[10],Chin等[11]在头颈部癌以及Rimkus等[12]在晚期直肠癌中也发现类似现象。功能分析显示,rs5994293为转录因子结合位点,其基因多态可能会影响到SF3A1的表达,而SF3A1的异常表达可能会改变剪切位点的选择[13],从而最终影响原癌基因和抑癌基因的正常剪接,最终直接或间接地导致肿瘤发生。有意思的是,SF3A1基因位于染色体22q.12.2上,Hu等[14]对中国汉族GWAS研究中发现22q.12.2这一区段2个SNP位点rs17728461与rs36600与肺癌发生密切相关。结合本研究结果,提示22q.12.2这一区段可能与肿瘤发生密切相关。
本研究在合并阶段HBsAg阴性者中观察到合并吸烟饮酒与SF3A1 rs5994293有协同作用。目前对于交互作用的解释尚不清楚。大量流行病学研究表明,嗜酒是肝癌发生的危险因素,酒精在人体内一系列代谢产物能与肝脏DNA形成赘合物从而引起细胞DNA的损伤,如P53基因损伤;而现烟草中含有多种致癌化学物质,与肺癌、口腔癌、胰腺癌、食管癌、胃癌和咽喉癌等13种癌症发生有关。吸烟产生的多环芳烃能诱导某些原癌基因如MDM2的剪接异构体产生,而MDM2是调节P53通路的重要基因,且已证实P53基因突变是肝癌发生的原因之一[15],提示吸烟、饮酒可能会导致肝癌相关的原癌基因或抑癌基因的选择性剪切,从而增加携带SF3A1 rs5994293 TT基因型者发生肝癌的风险。HBsAg阳性人群中未观察到合并吸烟饮酒与SNP的交互作用,其原因可能与HBV感染自身与肝癌发生效应太强(OR=7.59,95%CI:6.13~9.40)或样本量不够的缘故。有待加大样本量,在多个人群中对该基因与肝癌的发生风险进行深入地研究。
| [1] Jemal A,Bray F,Center MM,et al. Global cancer statistics[J]. CA Cancer J Clin,2011,61(2):69-90. |
| [2] Yu MC,Yuan JM. Environmental factors and risk for hepatocellular carcinoma[J]. Gastroenterology,2004,127(5 Suppl 1):S72-78. |
| [3] Wang ET,Sandberg R,Luo SJ,et al. Alternative isoform regulation in human tissue transcriptomes[J]. Nature,2008,456(7221):470-476. |
| [4] Padgett RA. New connections between splicing and human disease[J]. Trends Genet,2012,28(4):147-154. |
| [5] Tian J,Zhu BB, Tian Y, et al. Association between pancreatic cancer risk and the interaction of U2AF65 gene polymorphisms and smoking[J]. Chin J Epidemiol,2014,35(6):710-713. (in Chinese)田晶,朱贝贝,田瑶,等. 剪接蛋白U2AF65基因多态性和吸烟交互作用与胰腺癌风险的关联[J]. 中华流行病学杂志,2014,35(6):710-713. |
| [6] Källberg H, Ahlbom A, Alfredsson L. Calculating measures of biological interaction using R[J]. Eur J Epidemiol,2006,21(8):571-573. |
| [7] Tanackovic G,Krämer A. Human splicing factor SF3a, but not SF1,is essential for pre-mRNA splicing in vivo[J]. Mol Biol Cell,2005,16(3):1366-1377. |
| [8] Alexandrov LB, Nik-Zainal S, Wedge DC,et al. Signatures of mutational processes in human cancer[J]. Nature,2013,500 (7463):415-421. |
| [9] Ellis MJ,Ding L,Shen D,et al. Whole-genome analysis informs breast cancer response to aromatase inhibition[J]. Nature,2012,486(7403):353-360. |
| [10] Difilippantonio S, Chen Y, Pietas A, et al. Gene expression profiles in human non-small and small-cell lung cancers[J]. Eur J Cancer,2003,39(13):1936-1947. |
| [11] Chin D,Boyle GM,Williams RM,et al. Novel markers for poor prognosis in head and neck cancer[J]. Int J Cancer,2005,113(5):789-797. |
| [12] Rimkus C,Friederichs J,Boulesteix AL,et al. Microarray-based prediction of tumor response to neoadjuvant radiochemotherapy of patients with locally advanced rectal cancer[J]. Clin Gastroenterol Hepatol,2008,6(1):53-61. |
| [13] Matlin AJ,Clark F,Smith CW. Understanding alternative splicing:towards a cellular code[J]. Nat Rev Mol Cell Biol,2005,6(5):386-398. |
| [14] Hu ZB,Wu C,Shi YY,et al. A genome-wide association study identifies two new lung cancer susceptibility loci at 13q12.12 and 22q12.2 in Han Chinese[J]. Nat Genet,2011,43(8):792- 796. |
| [15] Bressac B,Kew M,Wands J,et al. Selective G to T mutations of p53 gene in hepatocellular carcinoma from southern Africa[J]. Nature,1991,350(6317):429-431. |
 2015, Vol. 36
2015, Vol. 36