文章信息
- 潘玥, 邵聪文, 王晓辉, 朱艳菊, 陈俊英, 陈伟, 马绍辉, 刘建生. 2014.
- Pan Yue, Shao Congwen, Wang Xiaohui, Zhu Yanju, Chen Junying, Chen Wei, Ma Shaohui, Liu Jiansheng. 2014.
- 人埃可病毒20型KM/EV20/2010分离株的全基因组序列分析
- Genomic characteristics of an echovirus 20 strain (KM/EV20/2010) isolated in Kunming, Yunnan, China
- 中华流行病学杂志, 2015, 36(5): 501-505
- Chinese Journal of Epidemiology, 2015, 36(5): 501-505
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2015.05.019
-
文章历史
- 投稿日期:2014-12-17
1. 毒株来源:KM/EV20/2010分离自与HAV混存的甲型肝炎患儿粪便标本,其分离鉴定见文献[12]。毒株保存于中国医学科学院北京协和医学院医学生物学研究所分子流行病室。
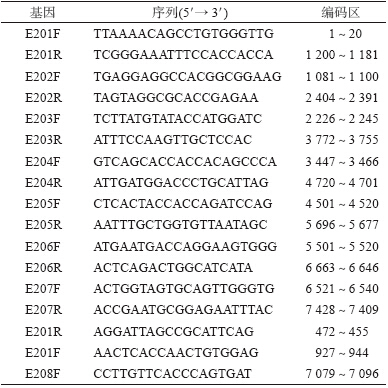
2. 病毒RNA提取、引物设计和RT-PCR:按照Axygen Body Fluid Viral DNA/RNA Miniprep Kit说明书从产生CPE的细胞培养上清中提取病毒RNA,并保存于-70 ℃。 根据ECHO20 原型株JV-1(基因登录号:AY302546),采用Primier 5.0软件设计覆盖其全长基因组的引物对(表 1)。RT-PCR采用TaKaRa公司(中国)生产的One-step RNA PCR Kit(AMV)试剂盒。分7段扩增:2×reaction buffer 25 μl,RT-PCR MIX 1 μl,引物F 10 pmol/L,引物R 10 pmol/L,病毒RNA 5 μl,用无RNA酶超纯水补足至50 μl,混匀、瞬时离心后50 ℃反转录30 min;94 ℃ 3 min,94 ℃ 30 s,50 ℃ 30 s,72 ℃ 1.5 min,30个循环扩增;72 ℃延伸10 min。然后取扩增产物3 μl,1.0%琼脂糖电泳,根据DL2000 Marker确认扩增片段。
3. 序列测定和数据分析:由华大基因科技股份有限公司(北京)对扩增产物进行纯化和测序(3次重复/样品)。从GenBank下载其他ECHO20及肠道病毒的全基因序列和VP1基因序列,利用Mega 5.1、BLAST和Geneious Basic 5.6.5软件分别进行系统进化分析(Neighbor-Joining方法)和核苷酸(氨基酸)同源性比对以及应用RDP3和SimPlot 3.5.1软件对ECHO20分离株KM/EV20/2010序列进行重组分析。 结 果
1. RT-PCR及测序:采用特异性引物进行一步法RT-PCR,取3 μl PCR产物经1%琼脂糖凝胶电泳后,将符合预期大小的目的片段扩增产物送检测序,然后将各片段拼接成一个完整序列,通过核苷酸的同源性分析发现该病毒分离株与ECHO20原型株JV-1的同源性为80.0%。
2. KM/EV20/2010全基因组序列测定:KM/EV20/2010的全基因组核苷酸序列长度为7 395 bp(基因登录号:JQ801739),其中5′端非编码区(5′UTR)长744 bp,病毒基因组编码区全长6 549个核苷酸,编码一含2 183个氨基酸残基的多聚蛋白;3′端非编码区末端长96 bp。KM/EV20/2010基因组的组成和结构符合肠道病毒特征。KM/EV20/2010整个基因组的核苷酸和氨基酸与JV-1同源性最高,分别为80.1%和96.7%。
3. KM/EV20/2010各区段核苷酸序列BLAST比对:将KM/EV20/2010全基因组各区段通过PUBMED BLAST与GenBank中的序列比对(表 2),发现其结构蛋白VP4、VP2、VP3和VP1与ECHO20血清型病毒株的相应基因区段同源性最高,其中VP4与ECHO20株EPSE20同源性最高,VP1与104-YN-2005AFP ECHO20株同源性最高;2A、2C、3A、3′UTR、5′UTR则分别与EV86、ECHO7、EV88、EV98、EV74株同源性最高;而2B、3B、3C则与ECHO30株同源性最高。另外,KM/EV20/2010分离株各基因区段与ECHO20原型株JV-1、ECHO30原型株Bastianni核苷酸和氨基酸进行比对(表 2),结果在结构蛋白编码区745~3 291,KM/EV20/2010分离株结构蛋白VP4、VP2、VP3和VP1各基因区段的核苷酸及氨基酸与ECHO20原型株JV-1同源性均在80%以上;而在非结构蛋白编码区3 292~7 293,KM/EV20/2010分离株非结构蛋白2B、2C、3A、3B、3C和3D基因区段的核苷酸及氨基酸与ECHO30原型株Bastianni的同源性均高于JV-1。
4. ECHO20全基因组种系进化分析:利用Mega 4.1软件对KM/EV20/2010分离株和GenBank中的ECHO20原型株JV-1以及其他ECHO病毒型别代表株,选用Neighbor-Joining方法构建基于全基因组序列的进化树(图 1)。结果表明,KM/EV20/2010分离株与ECHO20原型株JV-1遗传距离很远,而与ECHO30株遗传距离较近。迄今为止,GenBank中仅有1株ECHO20全长基因病毒株,并且未与KM/EV20/2010聚簇分布。
 |
| 图 1 ECHO20全基因核苷酸序列的种系进化分析 |
5. ECHO20基因分型:基于ECHO20全长VP1进化树图显示(图 2),JV-1原型株(1956年)构成基因型Ⅰ;N-948印度株(2011年)形成基因型Ⅱ;99357和99358 中国山东分离株(1999年)形成基因型Ⅲ;KM/EV20/2010则与01352中国山东分离株(2001年)、94CF1666法国分离株(1998年)聚簇为基因型Ⅳ;基因型Ⅴ和Ⅵ主要由2010年和2011年印度分离株聚簇形成。对各基因型进行相互之间核苷酸和氨基酸同源性比对分析(表 3),发现这6个基因型相互之间核苷酸的差异在9.4%~21.7%,氨基酸差异为0%~5.3%。
 |
| 图 2 ECHO20全基因核苷酸序列的种系进化分析 |
6. KM/EV20/2010重组事件分析:利用RDP3重组软件进行KM/EV20/2010和JV-1原型株及GenBank中同源性最高的肠道病毒代表株基于病毒基因组全长序列的SimPlot相似性分析,发现RDP、MaxChi、Chimaera和SiScan 3Seq分别检测出KM/EV20/2010重组事件(RDP法,P=1.82×10-13),亲本序列为ECHO88/BAN00-10354、JV-1、ECHO33/Toluca和ECHO30/GX10/05。为进一步证实重组,利用SimPlot 3.5.1软件进行相似性分析(图 3),结果显示,KM/EV20/2010在其基因组区段821~3 321与JV-1相似度在70%以上;在其基因组区段3 381~7 341则与ECHO30、ECHO86 相似度高于80%。
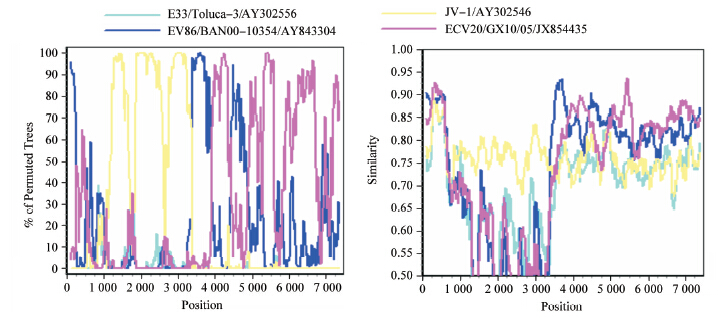 |
| 图 3 KM/EV20/2010与JV-1原型株及GenBank中同源性最高的肠道病毒代表株 基于病毒基因组全长序列的SimPlot相似性分析 |
ECHO20的原型株JV-1于1956年分离自一名临床无症状儿童的粪便标本。一般认为ECHO20在儿童中仅引起轻度发热、出疹性疾病[13, 14]。另有报道ECHO20感染新生儿引致肝衰竭而死亡[15]。国内外均在NPEV致AFP病例及环境污水中监测到ECHO20株,这些病毒可能在环境中以静默态循环,在人群中未显现相关疾病暴发或流行。
肠道病毒结构蛋白VP1基因与人类肠道病毒血清型具有良好的遗传关联度,是肠道病毒基因分型理想的靶标,常用于血清型别的分子鉴定[16]。本研究中KM/EV20/2010株的VP1序列与2005年分离自云南地区104-YN-2005AFP ECHO20株核苷酸同源性最高为97%,与JV-1原型株核苷酸、氨基酸同源性分别为80.1%和96.7%,从分子水平可鉴定为ECHO20基因型。 GenBank中与ECHO20 相关的序列绝大多数为结构蛋白VP1非完整序列,本研究选取库中完整的结构蛋白VP1基因序列,构建了基于全长VP1基因序列的进化树图,并进行基因分型初步分析。结果显示,ECHO20 形成6个基因型,JV-1原型株(1956年)构成基因型Ⅰ;N-948印度株(2011年)形成基因型Ⅱ;99357和99358 中国山东分离株(1999年)形成基因型Ⅲ;KM/EV20/2010则与01352中国山东分离株(2001年)、94CF1666法国分离株(1998年)聚簇为基因型Ⅳ;基因型Ⅴ和Ⅵ主要由2010年和2011年印度分离株聚簇形成。
鉴于GenBank中仅有ECHO20 JV-1原型株的全长基因组序列,选取库中ECHO病毒代表株的全长基因组序列,构建了基于ECHO病毒全长基因序列的进化树图。结果表明,KM/EV20/2010分离株与ECHO20原型株JV-1遗传距离很远,未与原型株JV-1聚簇分布,而与ECHO30病毒株遗传距离较近。该特征可能的原因:首先,全基因组序列比对显示,KM/EV20/2010基因组核苷酸序列与JV-1、ECHO30的同源性分别为80.1%、77.0%,仅相差3.1%;其次,KM/EV20/2010分离株结构蛋白与JV-1原型株的同源性高于ECHO30原型株Bastianni,而在非结构蛋白编码区,KM/EV20/2010分离株与ECHO30原型株Bastianni同源性高于JV-1,但是由于结构蛋白编码基因只占整个基因组的35%,而非结构蛋白基因则占比超过50%,从而使得KM/EV20/2010分离株在病毒全长基因序列的进化树图上与ECHO30病毒株遗传距离更近。
对KM/EV20/2010病毒分离株进行基因重组分析,结果显示KM/EV20/2010在其结构蛋白编码区(745~3 291)与ECHO20原型株相似性最高,而在非结构蛋白编码区(3 292~7 293)则与ECHO30、EV86相似度高于80%。可能的重组事件主要聚集发生在其基因组非结构蛋白编码区,这与各基因区段核苷酸同源性比对结果相一致。
从时间、空间进化分布而言,由于缺少中间过渡株,EV20病毒分子变异和进化特征信息很单薄,但是KM/EV20/2010病毒株全长基因序列的测定,无疑可丰富ECHO20病毒基因组信息的多态性,为进一步研究其遗传特性以及监测提供基础数据。
| [1] Pallansch MA, Roos RP. Enteroviruses:polioviruses, coxsackieviruses, echoviruses, and newer enteroviruses[M]//Knipe DM, Howley PM, Griffin DE, et al. Fields Virology. 5th ed. Philadelphia, PA:Lippincott, Williams &Wilkins, 2007:839-893. |
| [2] Tao Z, Wang H, Liu Y, et al. Non-polio enteroviruses from acute flaccid Paralysis surveillance in Shandong province, China, 1988-2013[J]. Sci Rep, 2014, 4:6167. doi:10.1038/srep06167. |
| [3] Wang HY, Liu K, Tao ZX, et al. Genotype distribution of enterovirus group B isolated in Shandong province, China[J]. Chin J Virol, 2010, 26(5):357-362. (in Chinese) 王海岩, 刘昆, 陶泽新, 等. 肠道病毒B 组山东地方株的基因型分布[J]. 病毒学报, 2010, 26(5):357-362. |
| [4] Chen HL, Liu Q, Wang DL, et al. Surveillance and analysis of enteroviruses in water environments in Shenzhen from 2010 to 2011[J]. Arch Virol, 2013, 158(6):1343-1347. |
| [5] Dhole TN, Ayyagari A, Chowdhary R, et al. Non-polio enteroviruses in acute flaccid paralysis children of India:vital assessment before polio eradication[J]. J Paediatr Chld H, 2009, 45(7/8):409-413. |
| [6] Adeniji JA, Faleye TOC. Isolation and identification of enteroviruses from sewage and sewage-contaminated water in lagos, Nigeria[J]. Food Environ Virol, 2014, 6(2):75-86. |
| [7] Laxmivandana R, Yergolkar P, Gopalkrishna V, et al. Characterization of the non polio enterovirus infections associated with acute flaccid paralysis in South-Western India[J]. PLoS One, 2013, 8(4):e61650. doi:10.1371/journal.pone.0061650. |
| [8] Apostol LN, Suzuki A, Bautista A, et al. Detection of non-polio enteroviruses from 17 years of virological surveillance of acute flaccid paralysis in the Philippines[J]. J Med Virol, 2012, 84(4):624-631. |
| [9] Khetsuriani N, Kutateladze T, Zangaladze E, et al. High degree of genetic diversity of non-polio enteroviruses identified in Georgia by environmental and clinical surveillance, 2002-2005[J]. J Med Microbiol, 2010, 59(Pt 11):1340-1347. |
| [10] Rao CD, Yergolkar P, Shankarappa KS. Antigenic diversity of enteroviruses associated with nonpolio acute flaccid paralysis, India, 2007-2009[J]. Emerg Infect Dis, 2012, 18(11):1833-1840. |
| [11] Oyero OG, Adu FD, Ayukekbong JA. Molecular characterization of diverse species enterovirus-B types from children with acute flaccid paralysis and asymptomatic children in Nigeria[J]. Virus Res, 2014, 189:189-193. |
| [12] Pan Y, Shao CW, Ji M, et al. Analysis of complete VP1 nucleotide sequence of the echovirus 20 KM/EV20/2010 strain isolated from Kunming in 2010[J]. J Patho Biol, 2014, 9(2):97-100, 134. (in Chinese) 潘玥, 邵聪文, 吉玛, 等. ECHO20 病毒株KM/EV20/2010的分离鉴定及其VP1基因序列特征分析[J]. 中国病原生物学杂志, 2014, 9(2):97-100, 134. |
| [13] Abbasian F, Saberbaghi T, Moosapour A. Role of non- polioviruses in acute flaccid paralysis (AFP)[J]. J Gastroenterol Hepatol Res, 2012, 1(4):44-48. |
| [14] Verboon-Maciolek MA, Krediet TG, van Loon AM, et al. Epidemiological survey of neonatal non-polio enterovirus infection in the Netherlands[J]. J Med Virol, 2002, 66(2):241-245. |
| [15] Chambon M, Delage C, Bailly JL, et al. Fatal hepatic necrosis in a neonate with echovirus 20 infection:use of the polymerase chain reaction to detect enterovirus in liver tissue[J]. Clin Infect Dis, 1997, 24(3):523-524. |
| [16] Oberste MS, Maher K, Kilpatrick DR, et al. Molecular evolution of the human enteroviruses:correlation of serotype with VP1 sequence and application to picornavirus classification[J]. J Virol, 1999, 73(3):1941-1948. |
 2015, Vol. 36
2015, Vol. 36