文章信息
- 谈潘莉, 汪浙炯, 孙爱华, 严杰, 赵金方. 2014.
- Tan Panli, Wang Zhejiong, Sun Aihua, Yan Jie, Zhao Jinfang. 2014.
- 大肠埃希菌临床菌株优势β-内酰胺酶基因型及其诱导表达与抑制的研究
- Predominant β-lactamase genotypes of Escherichia coli isolates and induction and inhibition mechanisms of β-lactamase gene expression
- 中华流行病学杂志, 2015, 36(5): 484-490
- Chinese Journal of Epidemiology, 2015, 36(5): 484-490
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2015.05.016
-
文章历史
- 投稿日期:2014-11-29
2. 浙江医学高等专科学校;
3. 浙江大学医学院病原生物学系
2. Zhejiang Medical College;
3. Department of Medical Microbiology and Parasitology, Medical School of Zhejiang University
大肠埃希菌(E. coli)常作为条件致病菌引起尿道炎、败血症、肺炎和新生儿脑膜炎等疾病[1]。临床上通常采用β-内酰胺类头孢菌素治疗E. coli感染性疾病,但该菌对β-内酰胺类抗生素有较高耐药率,其机制主要是产生β-内酰胺酶[2, 3, 4]。β-内酰胺酶有多种类型,其中以KPC、TEM、CTX-M、SHV和OXA较为常见,但不同地区E. coli的优势β-内酰胺酶基因型有一定差异[5, 6, 7, 8]。近年发现,通常由组氨酸激酶(histidine kinase,HK)和应答调节蛋白(response regulatory protein,RRP)组成的细菌二元信号系统(two-component signaling system,TCSS)与细菌耐药性密切相关[9, 10]。为此本研究对分离自浙江省部分地区的E. coli临床菌株检测β-内酰胺类抗生素耐药性及其β-内酰胺酶基因型,探讨β-内酰胺类抗生素诱导细菌β-内酰胺酶基因表达以及HK抑制剂氯氰碘柳胺(closantel,CLO)抑制细菌耐药性的作用,以了解当地优势β-内酰胺酶基因型及其携带模式,也可为抗细菌耐药新药研发提供依据。 材料与方法
1. 菌株来源及鉴定:462株E. coli临床分离菌株来自浙江省杭州、宁波和金华地区8家医院的下呼吸道感染病例痰液、感染性胸膜炎病例胸水、新生儿脑膜炎脑脊液、菌血症或败血症病例外周血、泌尿道重度感染病例尿液。上述菌株均经VITEK Compact 2型全自动细菌检测分析系统及其配套的细菌鉴定卡GNI鉴定。质控菌株为E. coli ATCC25922株由浙江大学医学院病原生物学系严杰教授惠赠。
2. 药物敏感试验:采用美国国家临床实验室标准委员会(NCCLS)推荐的微量稀释法检测上述临床菌株对青霉素(CPN)、氨苄青霉素(AMP)、头孢西丁(CFX)、头孢噻肟(CTX)、头孢他啶(CAZ)的最低抑菌浓度(MIC)[11]。
3. β-内酰胺酶及其基因检测:采用NCCLS 推荐的纸片法β-内酰胺酶确证试验[11]。将E. coli耐药菌株涂布于M-H琼脂平板,贴上CAZ、CAZ/克拉维酸(CA)或CTX、CTX/CA纸片(bioMerieux),35 ℃培养24 h后观察结果,若复合药物纸片抑菌环直径较相应单一药物纸片抑菌环直径≥5 mm为β-内酰胺酶确证试验阳性。
采用细菌基因组DNA提取试剂盒(Axygen)提取E. coli耐药菌株基因组DNA,紫外分光光度法测定其浓度和纯度[12]。根据文献报道的KPC、TEM、CTX-M、SHV和OXA基因通用引物序列[13, 14, 15, 16, 17],委托上海Invitrogen公司合成各引物(表 1)。采用高保真PCR试剂盒(TaKaRa)检测各菌株β-内酰胺酶基因。PCR反应总体积为100 μl,内含2.5 mol/L dNTP、200 nmol/L各引物、20 mol/L MgCl2、2.5 U EX-Taq DNA聚合酶、100 ng DNA模板和1×PCR缓冲液(pH 8.3)。PCR参数:94 ℃ 5 min;94 ℃ 30 s、52 ℃或55 ℃ 30 s、72 ℃ 120 s,30个循环;72 ℃ 10 min。采用溴乙锭预染色的1.5%琼脂糖凝胶电泳检测PCR结果,KPC、TEM、CTX-M、SHV和OXA基因扩增片段分别为1 050、972、882、1 017和441 bp。采用PCR产物纯化试剂盒(TaKaRa)回收各靶基因扩增片段,采用T-A克隆试剂盒(TaKaRa)将其克隆至pMD-19T质粒中。重组pMD-19T电转化入E. coli DH5α(Invitrogen),然后在含AMP(Sigma)LB培养液(bioMerieux)中扩增。采用质粒提取试剂盒(Axygen)提取E. coli DH5α中扩增的重组pMD-19T,委托上海Invitrogen公司测序,采用BLAST软件将测序结果与GenBank中相应序列进行比较,确定其β-内酰胺酶基因型[12]。
4. E-test:采用法国bioMerieux公司CTX和CPN纸片及其实验流程,以获得携带β-内酰胺酶单基因耐药菌株更精确的MIC。
5. 抗生素作用前后β-内酰胺酶基因mRNA检测:用1/4 MIC头孢噻肟或青霉素(Sigma)分别作用于89株携带β-内酰胺酶单基因耐药菌株0.5、1、2和4 h,8 000 r/min离心10 min(4 ℃),细菌沉淀用PBS洗涤、离心3次。取细菌沉淀用TRIzol试剂(Sigma)提取总RNA,分光光度法测定其浓度和纯度[12]。采用反转录试剂盒(TaKaRa)将等量各细菌总RNA反转录后获得cDNA。KPC、TEM、CTX-M、SHV、OXA基因以及作为内参照的16S rRNA基因实时荧光定量RT-PCR(qRT-PCR)引物见表 1,引物由上海Invitrogen公司合成。采用SYBR Premix Ex-TaqTM荧光定量PCR试剂盒(TaKaRa)及ABI-7500型实时荧光定量PCR仪,对各反转录产物进行扩增。反应体系20 μl,引物浓度为0.2 μmol/L,2 μl反转录产物为模板。反应参数:95 ℃ 30 s;95 ℃ 5 s、60 ℃ 20 s,40个循环。获得的qRT-PCR数据用REST2005软件及ΔΔCT相对定量法进行定量,并与抗生素作用前β-内酰胺酶基因mRNA水平进行比较[18]。采用E. coli 16S RNA基因作为内参照(表 1)[17]。
6. CLO毒性试验:100株耐药菌株中加入25、50、100、250或500 μg/ml的CLO(Sigma)后37 ℃作用0.5 h[19],然后分别采用微量稀释法及NCCLS介绍的最低杀菌浓度(MBC)检测方法分别测定CLO对E. coli的MIC和MBC[11]。以CLO未作用的菌株作为对照。
7. CLO作用前后抗生素诱导β-内酰胺酶基因mRNA水平检测:89株携带β-内酰胺酶单基因的耐药菌株用25、50、100、250或500 μg/ml的CLO(Sigma)37 ℃作用0.5 h。上述抗生素作用前后β-内酰胺酶基因mRNA检测显示,除KPC基因外,上述1/4 MIC头孢噻肟或青霉素作用1、2或4 h时TEM、CTX-M、SHV和OXA基因mRNA升高水平相似,故各菌株CLO预处理后加入1/4 MIC的CTX或CPN后37 ℃作用1 h,然后采用上述qRT-PCR检测细菌KPC、TEM、CTX-M、SHV和OXA基因mRNA水平变化。同样以CLO未作用菌株作为对照。
8. CLO抑制β-内酰胺酶表达及协同抑菌作用的检测:根据上述CLO毒性试验及CLO相关qRT-PCR检测结果,先用100 μg/ml的CLO(Sigma)37 ℃作用耐药菌株0.5 h,然后用上述微量稀释法和β-内酰胺酶确证试验分别检测5种β-内酰胺类抗生素对细菌MIC及β-内酰胺酶活性变化,检测结果与CLO未作用菌株进行比较。以E. coli ATCC25922为质控菌株。
9. 统计学分析:采用SPSS 13.0软件分别对细菌耐药率、β-内酰胺酶及其基因检出率、β-内酰胺酶mRNA水平进行χ2和t检验,P<0.01为差异有统计学意义。 结 果
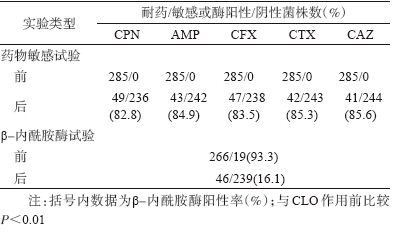
1. 耐药菌株β-内酰胺酶检测:采用微量稀释法测定,462株E. coli中有61.7%(285/462)的菌株对CPN、AMP、CFX、CTX和CAZ耐药。285株β-内酰胺类抗生素耐药菌株中有93.3%(266/285)的菌株β-内酰胺酶确证试验为阳性,采用PCR,285株β-内酰胺类抗生素耐药菌株可检出KPC、TEM、SHV、CTX-M、 SHV和/或OXA基因(图 1),与GenBanK中公布的相应β-内酰胺酶基因序列比较,其序列相似性高达99.6%~100%。
 |
| 注:M:DNA Marker(TaKaRa); 1:空白对照; 2~6:分别为KPC(1 050 bp)、TEM(972 bp)、CTX-M(882 bp)、SHV(1 017 bp)和OXA(441 bp)基因扩增条带图 1 E. coli临床分离株的β-内酰胺酶基因扩增条带 |
2. CLO作用后细菌耐药性和β-内酰胺酶阳性率变化:CLO对E. coli的MIC和MBC均>500 μg/ml。与100 μg/ml CLO作用前比较,285株耐药菌株经CLO作用后有82.8%(236株)、84.9%(242株)、83.5%(238株)、85.3%(243株)和85.6%(244株)菌株转为对CPN、AMP、CFX、CTX和CAZ敏感(P<0.01);β-内酰胺酶确证试验阳性率也从CLO作用前的95.1%(271/285)降低至作用后的16.1%(46/285)(P<0.01)。见表 2。
3. 优势β-内酰胺酶基因型及其携带模式:285株β-内酰胺类抗生素耐药菌株中1.8%(5/285)的菌株检出KPC基因、83.2%(237/285)检出TEM基因、75.1%(214/285)检出CTX-M 基因、10.2%(29/285)检出SHV基因、1.4%(4/285)检出OXA基因,TEM和CTX-M基因检出率显著高于KPC、SHV和OXA基因(P<0.01)。31.2%(89/285)菌株携带KPC、TEM、CTX-M、SHV或OXA单基因,68.8%(196/285)菌株携带两种以上β-内酰胺酶基因,其检出率显著高于携带β-内酰胺酶单基因菌株(P<0.01)。携带两种以上β-内酰胺酶基因菌株中,89.3%(175/196)菌株同时携带TEM和CTX-M基因(TEM+CTX-M),明显高于TEM+SHV(5.6%,11/196)、CTX-M+OXA(0.5%,1/196)、SHV+OXA(0.5%,1/196)和TEM+CTX-M+SHV(4.1%,8/196)基因携带模式(P<0.01),见表 3。
4. 低浓度抗生素对β-内酰胺酶mRNA水平的影响:与抗生素作用前比较,89株携带TEM、CTX-M、SHV或OXA单基因的耐药菌株经1/4 MIC 的CTX或CPN作用0.5 h后,TEM-mRNA、CTX-M-mRNA、SHV-mRNA和OXA-mRNA水平均显著提高(P<0.01),并在作用1或2 h时达到峰值,但KPC-mRNA无明显变化(图 2)。
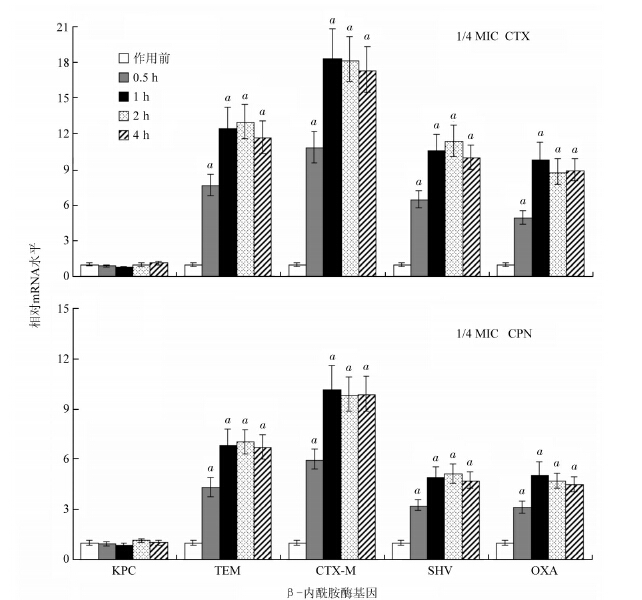 |
| 注:携带KPC、TEM、CTX-M、SHV和OXA β-内酰胺酶单基因的E. coli分别为5、43、30、9和2株; a 与1/4 MIC的CTX或CPN未作用菌株靶基因mRNA水平比较P<0.01图 2 1/4 MIC 的CTX和CPN诱导β-内酰胺酶基因转录效果 |
5. CLO抑制抗生素诱导β-内酰胺酶mRNA升高的作用:89株携带TEM、CTX-M、SHV或OXA单基因耐药菌株经采用浓度为50~500 μg/ml CLO作用后,明显抑制了上述1/4 MIC 的CTX或CPN可诱导细菌TEM-mRNA、CTX-M-mRNA、SHV-mRNA和OXA-mRNA水平升高现象(P<0.01),但对KPC-mRNA无明显影响(图 3)。
 |
| 注:携带TEM、CTX-M、SHV和OXAβ-内酰胺酶单基因的E. coli分别为43、30、9和2株;a与1/4MIC的CTX或CPN未作用菌株靶基因 mRNA水平比较P<0.01;b与CLO未预处理、1/4MIC的CTX或CPN作用菌株靶基因mRNA水平比较P<0.01图 3 CLO抑制CTX和CPN诱导β-内酰胺酶mRNA升高的作用 |
β-内酰胺酶根据作用机制不同可分为丝氨酸β-内酰胺酶和金属β-内酰胺酶[6]。而根据水解抗生素的不同,β-内酰胺酶又可分为广谱(broad-spectrum)和超广谱(extended-spectrum)两类,TEM-1、TEM-2和SHV-1属前者,可使细菌对青霉素类和一代头孢菌素头孢唑林(cefazolin)耐药,其余TEM和SHV以及KPC、CTX-M、OXA为后者,可使细菌耐药谱扩展至二代及以上头孢菌素和氨曲南(aztreonam)[5, 13]。
E. coli可产生多种广谱和超广谱β-内酰胺酶,且不同地区菌株优势β-内酰胺酶基因型有一定差异,如TEM和SHV型流行于北美和西欧、CTX-M型多见于东欧和亚洲及南美[2, 5, 7, 20]。我国临床分离菌株β-内酰胺基因型,广东地区以CTX-M型为主,山西地区常见TEM型[3, 4, 21]。本研究462株临床菌株中,61.7%对多种青霉素类和头孢菌素类抗生素耐药,耐药率与国内其他地区相似[3, 4];285株耐药菌株中,分别有83.2%和75.1%的菌株检出TEM和CTX-M基因,远高于SHV(10.2%)、KPC(1.8%)和OXA(1.4%)基因(P<0.01),表明TEM和CTX-M基因均为浙江地区E. coli的优势β-内酰胺酶基因型。此外,285株耐药菌株中,68.8%的菌株携带两种以上β-内酰胺酶基因,其中64.2%菌株同时携带TEM和CTX-M基因(61.4%为TEM+CTX-M,2.8%为TEM+CTX-M+SHV),表明TEM+CTX-M是浙江地区E. coli耐药菌株β-内酰胺酶基因主要携带模式。
临床上常将β-内酰胺酶抑制剂与头孢菌素联合应用,治疗β-内酰胺类抗生素耐药菌株感染性疾病,但一些抑制剂可与β-内酰胺酶结合使之失活,不能抑制其表达[22, 23]。业已证明,细菌HK胞外区与环境信号分子结合后发生变构,从而将信号传入胞内并使胞内区激酶催化位点中组氨酸被ATP磷酸化激活,RRP接受HK传递的磷酰基团后也随之活化,具备了与靶基因启动子结合并调控其表达的能力,若HK被抑制或失活,细菌无法对信号分子作出反应[9]。CLO可特异与细菌HK胞内酶催化区结合,引起HK分子变构、磷酸化障碍而失活,故CLO成为目前实验中常用的细菌HK特异性抑制剂[24]。β-内酰胺类抗生素主要有头孢菌素和青霉素两大类,本研究选择CTX和CPN。为防止不同β-内酰胺酶基因之间可能的干扰,试验中选择携带TEM、CTX-M、SHV或OXA单基因的耐药菌株。与真核细胞不同,原核细胞仅能在转录水平调控基因表达,故mRNA水平可直接反映基因的表达水平[10]。为了避免较高浓度抗生素时因细菌生长抑制而影响靶基因表达,还采用1/4 MIC的CTX或CPN以探讨其诱导β-内酰胺酶基因mRNA水平升高以及CLO抑制该mRNA升高的作用。qRT-PCR结果显示,在1/4 MIC的CTX或CPN作用下,TEM、CTX-M、SHV和OXA基因mRNA水平迅速显著升高,但可被50~500 μg/ml 的CLO抑制,提示低浓度CTX或CPN可能作为外源性信号分子经HK相关TCSS诱导E. coli β-内酰胺酶基因表达。然而,该剂量的CTX或CPN均未显示出可诱导KPC基因mRNA升高的作用,提示这些抗生素可能不是诱导KPC基因表达的外源性信号分子。
为了排除CLO对E. coli毒性而导致β-内酰胺酶基因mRNA水平降低,本研究还检测了CLO对E. coli的MIC和MBC,结果显示25~500 μg/ml的 CLO均无抑菌或杀菌作用。此外,药敏试验和β-内酰胺酶确证试验表明,经100 μg/ml CLO作用后82.8%~85.6%的耐药菌株转为对5种β-内酰胺类抗生素敏感,β-内酰胺酶检出率也从95.1%降至16.1%,不仅提示CLO具有协同β-内酰胺类抗生素抑菌的作用,同时也从蛋白水平上证实CLO可抑制E. coli 的β-内酰胺酶基因表达。
| [1] Da Silva GJ, Mendonça N. Association between antimicrobial resistance and virulence in Escherichia coli[J]. Virulence, 2012, 3(1):18-28. |
| [2] Rodríguez-Baño J, Navarro MD, Romero L, et al. Epidemiology and clinical features of infections caused by extended-spectrum β-lactamase-producing Escherichia coli in non-hospitalized patients[J]. J Clin Microbiol, 2004, 42(3):1089-1094. |
| [3] Hu FP, Zhu DM, Wang F, et al. 2011 CHINNET surveillance of bacterial resistance in China[J]. Chin J Infect Chemother, 2012, 12(5):321-329. (in Chinese) 胡付品, 朱德妹, 汪复, 等. 2011年中国CHINNET细菌耐药性监测 [J]. 中国感染与化疗杂志, 2012, 12(5):321-329. |
| [4] Wang F, Zhu DM, Hu FP, et al. 2012 CHINNET surveillance of bacterial resistance in China [J]. Chin J Infect Chemother, 2013, 13(5):321-330. (in Chinese) 汪复, 朱德妹, 胡付品, 等. 2012年中国CHINNET 细菌耐药性监测 [J]. 中国感染与化疗杂志, 2013, 13(5):321-330. |
| [5] Shah AA, Hasan F, Ahmed S, et al. Characteristics, epidemiology and clinical importance of emerging strains of Gram-negative bacilli producing extended-spectrum β-lactamases[J]. Res Microbiol, 2004, 155(6):409-421. |
| [6] Perez F, Endimiani A, Hujer KM, et al. The continuing challenge of ESBLs[J]. Curr Opin Pharmacol, 2007, 7(5):459-469. |
| [7] Chong Y, Ito Y, Kamimura T. Genetic evolution and clinical impact in extended-spectrum β-lactamase-producing Escherichia coli and Klebsiella pneumoniae[J]. Infect Genet Evol, 2011, 11(7):1499-1504. |
| [8] Sheng WH, Badal RE, Hsueh PR. Distribution of extended- spectrum β-lactamases, AmpC β-lactamases, and carbapenemases among Enterobacteriaceae isolates causing intra-abdominal infections in the Asia-Pacific region:results of the study for Monitoring Antimicrobial Resistance Trends (SMART)[J]. Antimicrob Agents Chemother, 2013, 57(7):2981-2988. |
| [9] Gotoh Y, Eguchi Y, Watanabe T, et al. Two-component signal transduction as potential drug targets in pathogenic bacteria[J]. Curr Opin Microbiol, 2010, 13(2):232-239. |
| [10] Sun AH, Fang JQ, Yan J. Advance and development in research of bacterial drug-resistance signaling mechanism and multiple antigenic peptide-based vaccines[J]. J Zhejiang Univ:Med Sci, 2013, 42(2):125-130. (in Chinese) 孙爱华, 方佳琪, 严杰. 病原菌耐药信号传导机制及多抗原肽疫苗研究进展与发展趋势[J]. 浙江大学学报:医学版, 2013, 42(2):125-130. |
| [11] National Committee for Clinical Laboratory Standards(NCCLS). Performance standards for antimicrobial susceptibility testing[M]. 20th ed. 21th Informational Supplement(M100-S21), NCCLS, Wayne:NCCLS, 2011. |
| [12] Sambrook J, Fritsch EF, Maniatis T. Molecular Cloning:A Laboratory Manual[M]. 2nd ed. New York:Cold Spring Harbor Laboratory Press, 1989:1.21-1.52, 2.60-2.80, 7.30-7.35, 9.14-9.22. |
| [13] Bradford PA. Extended-spectrum β-lactamases in the 21st century:characterization, epidemiology, and detection of this important resistance threat[J]. Clin Microbiol Rev, 2001, 14(4):933-951. |
| [14] Nuesch-Inderinen MT, Hachler H, Kayser FH. Detection of genes coding for extended-spectrum SHV beta-lactamases in clinical isolates by a molecular genetic method, and comparison with E test[J]. Eur J Clin Microbiol Infect Dis, 1996, 15(5):398-402. |
| [15] Holstein A, Grillon A, Yzon L, et al. Prevalence of extended- spectrum β-lactamases of the CTX-M type producing Escherichia coli and Klebsiella pneumoniae in Bretonneau hospitals (CHRU Tours)[J]. Pathol Biol (Paris), 2010, 58(1):67-69. |
| [16] Yu YS, Ji SJ, Chen YG, et al. Resistance of strains producing extended-spectrum β-lactamases and genotype distribution in China[J]. J Infect, 2007, 54(1):53-57. |
| [17] Wu YF, Sun AH, Zhao JF, et al. Distribution of drug inactive enzyme genes in bacterial isolates and mechanism of its induction and inhibition [J]. J Zhejiang Univ:Med Sci, 2013, 42(2):131-140. (in Chinese) 吴亦斐, 孙爱华, 赵金方, 等. 细菌药物钝化酶基因分布及其表达诱导与抑制机制的研究[J]. 浙江大学学报:医学版, 2013, 42(2):131-140. |
| [18] Pfaffl MW, Horgan, GW, Dempfle L. Relative expression software tool(REST) for group-wise comparison and statistical analysis of relative expression results in real-time PCR[J]. Nucleic Acids Res, 2002, 30(9):e36-45. |
| [19] Kumagai YM, Cheng ZH, Lin MQ, et al. Biochemical activities of three pairs of Ehrlichia chaffeensis two-component regulatory system proteins involved in inhibition of lysosomal fusion[J]. Infect Immun, 2006, 74(9):5014-5022. |
| [20] Falagas ME, Karageorgopoulos DE. Extended-spectrum β-lactamase- producing organisms[J]. J Hosp Infect, 2009, 73(4):345-354. |
| [21] Zhuo C, Su DH, Li HY, et al. Study on CTX-M type ESBLs- producing Escherichia coli and Klebsiella pneumoniae in Guangzhou[J]. Chin J Lab Med, 2009, 32(10):1114-1119. (in Chinese) 卓超, 苏丹虹, 李红玉, 等. 广州地区产CTX-M型超广谱β内酰胺酶大肠埃希菌和肺炎克雷伯菌的研究[J]. 中华检验医学杂志, 2009, 32(10):1114-1119. |
| [22] Thomson JM, Distler AM, Bonomo RA. Overcoming resistance to β-lactamase inhibitors:comparing sulbactam to novel inhibitors against clavulanate resistant SHV enzymes with substitutions at Ambler position 244[J]. Biochemistry, 2007, 46(40):11361-11368. |
| [23] Sood S. Comparative evaluation of the in-vitro activity of six β-lactam/β-lactamase inhibitor combinations against Gram negative bacilli[J]. J Clin Diagn Res, 2013, 7(2):224-228. |
| [24] Stephenson K, Yamaguchi Y, Hoch JA. The mechanism of action of inhibitors of bacterial two-component signal transduction systems[J]. J Biol Chem, 2000, 275(49):38900-38904. |
 2015, Vol. 36
2015, Vol. 36