文章信息
- 王碧琦, 周斌, 高文静, 李立明. 2014.
- Wang Biqi, Zhou Bin, Gao Wenjing, Li Liming. 2014.
- 双生子在表观遗传学研究中的价值
- The value of twin study in epigenetic research
- 中华流行病学杂志, 2015, 36(4): 402-404
- Chinese Journal of Epidemiology, 2015, 36(4): 402-404
- http://dx.doi.org/10.3760/cma.j.issn.0254-6450.2015.04.023
-
文章历史
- 投稿日期:2014-10-10
表观遗传现象(epigenetics)一般定义为DNA序列未发 生变化但基因功能发生了可遗传改变的现象,包括DNA甲 基化(DNA methylation) 及染色质重塑(Chromatin remodeling)等[1, 2]。表观遗传流行病学是研究表观遗传现象 在人群中的分布及其与人类健康关系的学科[3],运用双生子 人群可区分遗传变异贡献大小,在控制基因结构变异和大部 分环境因素的条件下,分析背后表观遗传学变化机制[4]。本 文综述双生子人群在表观遗传学研究中的优势,特别是不一 致同卵双生子(discordant monozygotic twins,DMZ)研究在 表观遗传流行病学中的价值与进展,以期为后续相关研究提 供思路。
1. 双生子人群在表观遗传学研究中的优势:
(1)可以证实表观遗传现象既与环境因素相关又与遗传 因素相关。遗传结构高度一致的同卵双生子(monozygotic twins,MZ)对内比较时,调控实验检测误差和随机误差后表 观遗传现象存在差异[5, 6],差异可能与环境暴露(特别是生 命早期暴露,例如母体孕期和胎儿生命早期的环境因素)相 关[7, 8]。MZ 表观遗传现象的相关性明显高于异卵双生子 (dizygotic twins,DZ)的水平,说明遗传因素对表观遗传现象 也有影响[6, 9]。
(2)可通过分析比较MZ对内表观遗传现象一致程度和 DZ对内一致程度,估计表观遗传现象变异中遗传或环境变 异的贡献大小。例如,将DNA甲基化水平作为结局表型,利 用双生子模型计算甲基化水平的遗传度。值得注意的是, DNA甲基化水平的遗传度可能是随时间变化的动态变量, 可结合纵向数据进行分析[10]。有研究显示,DRD4 基因DNA 甲基化水平的遗传度在5 岁双生子人群是0.36,而在同一人 群10 岁时为0.06[11]。家系研究同样可以计算表观遗传现象 的家族聚集性和遗传度,但其主要目的是验证表观遗传现象 可在亲代与子代间遗传,而双生子研究的主要目的是估计遗 传和环境变异对表观遗传现象变异的影响。
(3)双生子研究可以发现累积环境因素暴露可能与表观 遗传现象随时间的变化相关。Fraga 等[12]对40 对MZ全基因 组DNA甲基化进行测定,发现不同年龄段甲基化水平存在 差异,且随着年龄增加,环境因素愈加复杂,MZ对内DNA甲 基化差异水平越高。双生子研究特别是MZ研究,在不同年 龄段均有一个高度匹配的个体与其同胞对进行比较,双生子 表观遗传现象对内差异可能是匹配去除年龄、基因结构等影 响后对内其他环境因素差异造成的,如果双生子表观遗传现 象的对内差异在不同年龄段之间有所不同,在控制其他环境 因素对内差异后,可表现为年龄(或时间)对表观遗传现象的 影响。而在一般人群不同年龄段的横断面研究中,需要控制 基因结构及其他环境因素等多种混杂因素才可了解年龄对 表观遗传现象的影响。队列研究可直接发现表观遗传现象 随时间的变化,但研究时间较长,花费人力物力较多。
(4)疾病不一致双生子研究(disease discordant twins)在 确定效应值强度和显著性水平条件下,用较少的样本量即可 发现与疾病相关的表观遗传现象变化。疾病不一致的双生 子研究可作为1 ∶ 1 匹配的病例对照研究与无匹配的病例对 照研究进行比较,假设单个DNA甲基化水平均值在对照组 为13%,疾病组为14%,标准差均为4%,显著性水平取0.05, 统计学效力为80%的情况下[13, 14],用SAS 9.2 计算样本量的 结果是无匹配病例对照研究需要样本量为病例组和对照组 均为506 人,而1 ∶ 1 匹配病例对照研究需要样本量在病例组 和对照组均为153 人,假设病例和对照对内相关性越高,需 要的样本量越少,当对内相关性达到0.9 时,病例和对照均为 28 人即可满足足够样本量。MZ对内相关性较高,因而需要 样本量较少[15]。
(5)疾病DMZ研究由于基因、生活环境较为一致,可控 制更多未知混杂因素,简化分析过程,更易于解释。假设表 型(P)=基因(G)+环境(E)+基因-环境交互作用(GEI),基 因-环境交互作用为表观遗传现象与其他GEI 的作用,那么 该公式可修订为P=G+E+epigenetics+其他GEI,疾病不 一致双生子可确定等式左边表型P 存在差异,等式右边基因 结构G相同,那么表型的变化很可能是环境、表观遗传现象 和/或其他GEI 作用变异造成的[4]。实验研究因随机分组可 控制潜在混杂因素在实验组和对照组的分布,但目前实验研 究较少,样本量、观察时间受限,部分研究并不是随机对照试 验而具有一定局限性[16],而MZ研究可能是相对实验研究较 为经济、省时,可控制较多混杂因素的研究设计。
上述前三项双生子研究优势主要通过双生子模型拟合 估计表观遗传现象遗传度的方法实现,而后两项双生子研究 优势主要通过比较疾病或暴露表型DMZ的研究方法实现。 从疾病病因机制学角度的价值来看,表观遗传现象的遗传度 估计可能为寻找病因提供一定线索,而DMZ研究对寻找到 疾病相关的表观遗传现象及其与遗传、环境因素的关系应 用价值更大,其应用价值在于尽管基因结构很难改变,但可 以通过改变基因表达为疾病预防和控制提供思路。因此, 本文后半部分将对目前DMZ表观遗传学研究进展展开进一 步分析。
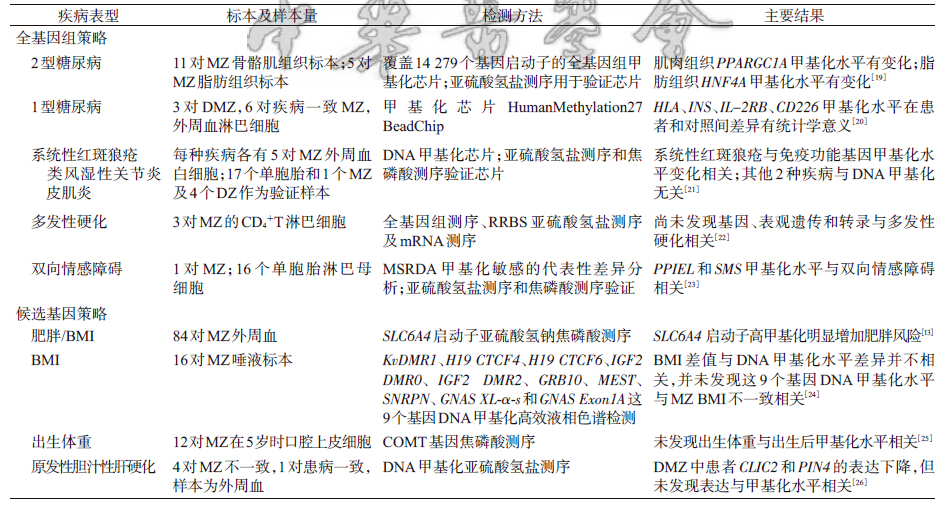
2. DMZ表观遗传学研究进展:DMZ研究设计应用在多 种疾病表型和表观遗传现象关系的研究中(表 1)。进行表观 遗传学研究的疾病主要为免疫系统疾病、心理疾病及代谢性 疾病,研究策略分为全基因组和候选基因策略,所用MZ样 本量均不到100 对。Bell和Spector[15]关于疾病不一致双生子 的综述认为当用某种芯片测定DNA甲基化水平时,若需要 发现不一致双生子之间甲基化水平之比至少为1.2 的效应强 度,且要求统计效力达到80%以上,所需要疾病DMZ大约 15~25 对。但这种估计仅适用于芯片研究,并不适合亚硫酸 氢盐测序等方法。检测方法主要为表观遗传学相关芯片或 测序。从研究结果来看,同一疾病在不同样本组织中得到的 结果可能不同。基因结构在不同组织的细胞中基本相同,但 DNA甲基化却是组织特异性的[17]。因此在利用便于获得的 标本(例如外周血标本)检测替代目标疾病相关组织DNA甲 基化检测时需要慎重。即使在使用外周血标本用于DNA甲 基化检测时,外周血中不同细胞种类(例如淋巴细胞、嗜碱性 粒细胞等)DNA甲基化情况也可能不同,DNA甲基化的改变 很可能是由于血液中细胞构成的变化(例如病毒感染状况下 淋巴细胞会增多)而并非细胞本身基因表达发生变化[18]。
除了需要注意不同组织会影响研究结果之外,在开展 DMZ表观遗传学研究时还需要注意以下问题:①人群代表 性,研究发现的表观遗传现象差异位点有可能是双生子人群 特殊表达的位点,因此需要验证样本进行验证,最近Lancet 发表的一篇全基因组DNA 甲基化水平与BMI 的关联研究 就是用1 个双生子队列(MuTHER 队列)及3 个一般人群队 列(Cardiogenics、MARTHA及KORA队列)进行相互验证得 到的结果[14]。②样本量和统计效力考虑,在涉及全基因组 表观遗传现象的研究中需要注意不同表观遗传相关位点之 间多重比较的问题,避免假阳性出现。而候选基因策略下 的表观遗传研究设计需要在假定效应强度下对样本量统计 效力进行估计,观察获得的样本量是否能达到可接受的统 计效力[19]。③横断面研究的局限,某些暴露或结局表型可能 是暂时的(例如肥胖表型可能是暂时的),而表观遗传现象 也可能是暂时的,长期肥胖相关DNA甲基化水平可能与短 期肥胖不同[27]。横断面研究结果需要在纵向研究中得到验 证。④病因推断考虑,在分析表观遗传现象与表型之间因 果关系时,需考虑效应修饰、未调控的混杂以及反向因果关 系等问题[28]。
3. 总结与展望:双生子研究在表观遗传学中的应用有助 于认识表观遗传现象中遗传和环境所起到的作用,MZ基因 结构及部分环境因素高度匹配,利于寻找与疾病相关的表观 遗传现象[29]。表观遗传现象的数据结合基因、转录、蛋白及 代谢相关数据,系统、全面分析某种疾病的发病机制是今后 发展的方向[30, 31]。已有研究在双生子人群中分析基因结构 与表达之间的关系,使双生子研究成为识别和理解复杂疾病 表型背后分子机制的一个有力途径[32, 33]。
| [1] Ledford H. Language:disputed definitions[J]. Nature, 2008, 455(7216):1023-1028. |
| [2] Miller G. Epigenetics. The seductive allure of behavioral epigenetics[J]. Science, 2010, 329(5987):24-27. |
| [3] Liang MB, Hu YH, Wu YQ. Emergence of a new branch on epidemiology:epigenetic epidemiology[J]. Chin J Epidemiol, 2009, 30(10):1078-1080. (in Chinese)梁明斌, 胡永华, 武轶群. 表观遗传流行病学——新兴流行病学分支学科[J]. 中华流行病学杂志, 2009, 30(10):1078-1080. |
| [4] Petronis A. Epigenetics and twins:three variations on the theme[J]. Trends Genet, 2006, 22(7):347-350. |
| [5] Zhao SM, Zhang SH, Chen JZ, et al. Differences of DNA methylation profiles in monozygotic twins' blood samples[J]. J Forensic Med, 2011, 27(4):260-264. (in Chinese)赵书民, 张素华, 陈金中, 等. 同卵双生子外周血DNA甲基化谱的差异[J]. 法医学杂志, 2011, 27(4):260-264. |
| [6] Kaminsky ZA, Tang T, Wang SC, et al. DNA methylation profiles in monozygotic and dizygotic twins[J]. Nat Genet, 2009, 41(2):240-245. |
| [7] Mill J, Heijmans BT. From promises to practical strategies in epigenetic epidemiology[J]. Nat Rev Genet, 2013, 14(8):585-594. |
| [8] Gordon L, Joo JHE, Andronikos R, et al. Expression discordance of monozygotic twins at birth:effect of intrauterine environment and a possible mechanism for fetal programming[J]. Epigenetics, 2011, 6(5):579-592. |
| [9] Hösler R, Begun AA, reitag-Wolf S, et al. Genetic control of global gene expression levels in the intestinal mucosa:a human twin study[J]. Physiol Genomics, 2009, 38(1):73-79. |
| [10] Madrigano J, Baccarelli A, Mittleman MA, et al. Aging and epigenetics:longitudinal changes in gene-specific DNA methylation[J]. Epigenetics, 2012, 7(1):63-70. |
| [11] Wong CCY, Caspi A, Williams B, et al. A longitudinal study of epigenetic variation in twins[J]. Epigenetics, 2010, 5(6):516-526. |
| [12] Fraga MF, Ballestar E, Paz MF, et al. Epigenetic differences arise during the lifetime of monozygotic twins[J]. Proc Natl Acad Sci USA, 2005, 102(30):10604-10609. |
| [13] Zhao J, Goldberg J, Vaccarino V. Promoter methylation of serotonin transporter gene is associated with obesity measures:a monozygotic twin study[J]. Int J Obes, 2013, 37(1):140-145. |
| [14] Dick KJ, Nelson CP, Tsaprouni L, et al. DNA methylation and body-mass index:a genome-wide analysis[J]. The Lancet, 2014, 383(9933):1990-1998. |
| [15] Bell JT, Spector TD. A twin approach to unraveling epigenetics[J]. Trends Genet, 2011, 27(3):116-125. |
| [16] Milagro FI, Campión J, Cordero P, et al. A dual epigenomic approach for the search of obesity biomarkers:DNA methylation in relation to diet-induced weight loss[J]. FASEB J, 2011, 25(4):1378-1389. |
| [17] Feinberg AP. Epigenetics at the epicenter of modern medicine[J]. JAMA, 2008, 299(11):1345-1350. |
| [18] Michels KB. Epigenetic epidemiology[M]. Germany:Springer, 2012. |
| [19] Ribel-Madsen R, Fraga MF, Jacobsen S, et al. Genome-wide analysis of DNA methylation differences in muscle and fat from monozygotic twins discordant for type 2 diabetes[J]. PLoS One, 2012, 7(12):e51302. |
| [20] Stefan M, Zhang W, Concepcion E, et al. DNA methylation profiles in type 1 diabetes twins point to strong epigenetic effects on etiology[J]. J Autoimmun, 2014, 50:33-37. |
| [21] Javierre BM, Fernandez AF, Richter J, et al. Changes in the pattern of DNA methylation associate with twin discordance in systemic lupus erythematosus[J]. Genome Res, 2010, 20(2):170-179. |
| [22] Baranzini SE, Mudge J, van Velkinburgh JC, et al. Genome, epigenome and RNA sequences of monozygotic twins discordant for multiple sclerosis[J]. Nature, 2010, 464(7293):1351-1356. |
| [23] McGowan PO, Kato T. Epigenetics in mood disorders[J]. Environ Health Prev Med, 2008, 13(1):16-24. |
| [24] Souren NYP, Tierling S, Fryns JP, et al. DNA methylation variability at growth-related imprints does not contribute to overweight in monozygotic twins discordant for BMI[J]. Obesity(Silver Spring), 2011, 19(7):1519-1522. |
| [25] Mill J, Dempster E, Caspi A, et al. Evidence for monozygotic twin (MZ) discordance in methylation level at two CpG sites in the promoter region of the catechol-O-methyltransferase (COMT) gene[J]. Am J Med Genet B Neuropsychiatr Genet, 2006, 141B(4):421-425. |
| [26] Mitchell MM, Lleo A, Zammataro L, et al. Epigenetic investigation of variably X chromosome inactivated genes in monozygotic female twins discordant for primary biliary cirrhosis[J]. Epigenetics, 2011, 6(1):95-102. |
| [27] Kim M, Long TI, Arakawa K, et al. DNA methylation as a biomarker for cardiovascular disease risk[J]. PLoS One, 2010, 5(3):e9692. |
| [28] Relton CL, Smith GD. Epigenetic epidemiology of common complex disease:prospects for prediction, prevention, and treatment[J]. PLoS Med, 2010, 7(10):e1000356. |
| [29] van Dongen J, Slagboom PE, Draisma HH, et al. The continuing value of twin studies in the omics era[J]. Nat Rev Genet, 2012, 13(9):640-653. |
| [30] Cornelis MC, Hu FB. Systems epidemiology:a new direction in nutrition and metabolic disease research[J]. Cur Nutr Reports, 2013, 2(4):225-235. |
| [31] Hu FB. Metabolic profiling of diabetes:from black-box epidemiology to systems epidemiology[J]. Clin Chemistry, 2011, 57(9):1224-1226. |
| [32] Bell JT, Tsai P, Yang T, et al. Epigenome-wide scans identify differentially methylated regions for age and age-related phenotypes in a healthy ageing population[J]. PLoS Genet, 2012, 8(4):e1002629. |
| [33] Moayyeri A, Hammond CJ, Valdes AM, et al. Cohort profile:twinsUK and healthy ageing twin study[J]. Internat J Epidemiol, 2013, 42(1):76-85. |
 2015, Vol. 36
2015, Vol. 36